摘要
背景
长穿插元素1 (LINE-1或L1)逆转录转座子是构成人类基因组17-20%的可移动元素。L1异常表达与几种人类疾病之间有很强的相关性。这激发了人们对任何给定生物标本中L1拷贝数量的精确量化的兴趣。实现这一目标的一个主要障碍是l1是具有高变异性区域的相对较长的DNA片段,或者主要以截断片段的形式存在于人类基因组中。这些特殊性使得传统的对齐策略(如播种-扩展)效率低下,因为与l1相似的段的数量呈指数级增长。本研究使用模式匹配方法更准确地识别l1。我们通过实验验证了模式匹配L1检测优于其他方法,并讨论了一些潜在的应用。
结果
模式匹配检测全长L1副本精度高,计算时间合理,且无需事先输入信息。它还以相对较高的精度检测到截断和显著改变的L1副本。该方法被有效地用于注释目标基因组中的l1,并计算相对于参考基因组的拷贝数变化。成功实施的关键是从一组在基因组中呈现稳定分布模式的序列中选择了一小组k-mer探针。与播种-扩展方法一样,模式匹配算法播种了这些k-mer探针,但不是在种子周围使用启发式扩展,而是基于基因组内的分布模式进行分析。期望的精度水平可以调整,但会有一定的回忆损失。
结论
模式匹配比种子-扩展方法更有效地检测L1段,其特征依赖于具有低变异性的公共区域的有限序列集。我们提出模式匹配可能有助于建立L1拷贝数与L1动员和进化相关的疾病状态之间的相关性。
背景
转座元件或转座子,如长插入元件I (L1), Alu和SVA元件,是在基因组中从一个位置移动到另一个位置的DNA序列。这些元素是基因组进化、遗传变异和基因组不稳定性的重要贡献者,并与几种疾病有关,包括神经纤维瘤病、脉络膜缺乏症、胆碱酯酶缺乏症、Apert综合征、Dent’s病和Walker-Warburg综合征[1].L1是已知的人类基因组中唯一活跃的自主非ltr转座子。这意味着它有能力将自己或其他非自主转座子复制粘贴到不同的基因组位置[2,3.,从而加剧了其有害影响。目前有超过50万个l1序列,占人类基因组的17%。l1由两个开放阅读帧组成,即所谓的ORF1和ORF2, 5 '和3 '非翻译区域(utr),一个orf间区域和一个聚(a)尾[3.].虽然大多数l1由于重排、点突变和截断而不活跃[2,3.],全长、活性l1可能是致病的,并且最有可能以显著的速率发生逆转录转位,至少有124个l1介导的插入与遗传疾病有关[3.,4].
因此,我们的方法集中于全长L1序列。我们的主要数据来源是L1Base 2数据库[5,6].在基因组参考联盟的人类参考基因组GRCh38中,L1Base 2报告了146个orf完整的l1, 107个ORF2完整的l1(中断的ORF1,但完整的ORF2),以及13418个反转录转位不活跃的全长不完整的l1。
现有的用于移动元素研究的检测工具
我们考虑了一些现有的工具,如VariationHunter [7]、茶[8], RetroSeq [9]及七巧板[10]用于检测l1。但是,这些工具主要用于检测移动元素的插入,而不是删除。这是一个相当大的限制,因为L1副本可能会经历从几个到数百个碱基对的片段丢失,而L1插入或删除对人类基因组的影响尚不清楚。因此,这些工具被认为不适合进行L1的全面检测。
一个众所周知且常用的工具是RepeatMasker。它的描述说,它是一个工具,旨在“筛选DNA序列的穿插重复和低复杂性DNA序列”[11].因此,RepeatMasker更广泛,包括不同类型的反转录转座子、DNA转座子和其他转座子元素。在L1检测中,范围的振幅是以损失精度为代价的。在我们团队执行的一些实验中,报告的LINE-1s的起始位置并不正确,并且在l1内部发现了零星的Alus和哺乳动物范围的穿插重复序列(MIRs),这迫使我们进行后过滤过程以获得真实的l1。此外,LINE-1的最后一个组件是poly(a)尾部,而RepeatMasker通常报告在poly(a)尾部之后的另一个LINE-1段。
播种和扩展的缺点
我们还研究了一些不太专业的工具,比如BLAST [12],这是用于近似基因比对的经典快速工具。一般来说,我们假定基于种子-扩展策略的工具,如BLAST,对于精确的L1检测是不够的。这些工具将短段长度对齐k,被称为k-mers,目标序列中的段。这样的对齐被称为种子,它们被启发式地扩展以找到完全相似的段。在l1的情况下,扩展的启发式产生了大量的假阳性。例如,使用BLAST将L1Base2中的L1与1000基因组计划中的基因组对齐[13],即使e值为\ (10 ^ {-250} \),返回数万个结果。因此,寻找真正的l1需要对结果进行昂贵且耗时的后期处理。这种结果并不令人惊讶,因为种子-扩展策略以计算时间换取精度。
Seed-and-pattern匹配
在本文中,我们介绍了一种模式匹配策略来检测l1,该策略在合理的计算时间和用户提供的信息量最小的情况下达到足够的精度。在这里,模式匹配指的是一种完善的计算技术,其中测试表达式以确定它们是否匹配某个模式的组成部分。与上述研究的种子-扩展策略相比,引入的策略是一种种子-模式匹配策略,它用模式匹配取代了种子-扩展扩展阶段的启发式,以减少输入和后处理负担,同时提高检测的精度。
我们的算法在一个小的固定探针集上播种,并使用关于探针在查询集中位置的信息来决定一组种子是否位于L1中。这避免了扩展的启发式及其局限性,并提供了一种在人类基因组中检测l1的有效方法。
方法与结果
一般来说,要解决的问题可以表述如下:给定一个查询类,由有限的序列集描述,这些序列共享具有局部相似性的列范围,称为查询集,查找基因组中属于该类的所有片段。一般来说,转座子和可移动遗传元素是我们所说的查询集的例子。这些元素中高度保守的部分,如基因,反过来是探针的天然候选。在我们的特定情况下,l1组成我们的查询类。
如上所述,模式匹配策略使用一小组k-mer段或探针。探测是与查询集中的大多数序列具有高度局部相似性的段,因为本节将解释如何为我们的算法生成它们的细节。探针存储在5 '到3 '方向,以及它们从ORF1开始的平均偏移距离。探测和与它们关联的信息只计算一次,然后在每个后续应用程序中重用。为了将目标基因组的一个片段分类为查询类的成员,探针首先使用快速映射算法映射到基因组上。在播种-扩展方法中,我们称种子为目标基因组中与探针近似匹配的片段。作为模式匹配,我们在最小数量的种子中定义一个种子序列米,与查询集中探针的顺序和距离一致。种子之间的距离是用碱基对来测量的,可能会因为插入或节段损失而变化。为了解释这些变化,我们使用了一个输入参数\ (t > 0 \)表示阈值界限。另一个输入参数是种子的最小数量米必须符合模式匹配。与t、参数米解释了目标基因组中部分或全部丢失探针的片段。
因此,在符号意义上,模式与\(m = 3\)如下:Let\ (p_1 \),\ (p_2 \)而且\ (p_3 \)在5 '到3 '方向上进行探测并让\ (d_1 \)是之间的平均距离\ (p_1 \)而且\ (p_2 \)在查询集的序列中。同样,让\ (d_2 \)是之间的平均距离\ (p_2 \)而且\ (p_3 \).假设\ (s_1 \),\ (s_2 \)而且\ (s_3 \)对应的种子是否在目标基因组上被letδ_1 \ \ (\)而且δ_2 \ (\ \)为碱基对之间的距离\ (s_1 \)而且\ (s_2 \),\ (s_2 \)而且\ (s_3 \),分别。然后,\ (s_1 \),\ (s_2 \),\ (s_3 \)模式匹配当且仅当\(|d_1 - \delta _1| \le t\)而且\(|d_2 - \delta _2| \le t\).
为了使用模式匹配将基因组中的片段分类为L1类的成员,我们以L1Base2中146个orf完整的L1作为查询集。探测是通过查询集中序列的多个序列对齐的连续细化提取的,如下所述。由于多序列对齐问题没有客观的数学函数来优化,大多数从业者手动修改计算输出,或者从头进行手动对齐。在我们的例子中,多重对齐是使用bioSyntax手动完成的[14在vim编辑器中高亮显示包。我们发现,虽然utr在一定范围内表现出一些相似之处,但5 ' -UTR片段在前1000个碱基内变化很大。在3 ' -UTR区域观察到类似的行为。然而,ORF1和ORF2区域始终表现出规律的行为。我们决定只从orf中提取探针,并分别处理orf1和orf2。
orf的多重比对显示,至少95%的序列中出现最高核苷酸碱基的列。有几块这样的柱子有50根甚至更多。因此,我们从这些区块中提取了一组50-mer作为探测的预候选。为了获得实际的探针,我们通过一个三步细化过程对该集合进行筛选。
- 1
为了优化覆盖范围,我们保留了50个mer,它们只映射到查询集的每个成员中对应的ORF,这样做是为了避免探针映射到两个ORF。
- 2
从剩下的50个mers中,我们选择了那些在基因组上有最少的地图命中数的人,这样我们就可以降低假阳性的数量。
- 3.
我们从剩下的集合中选择了所有不重叠的50-mer的子集。
在这个过滤过程结束时,我们从orf1中得到了5个50-mers,从orf2中得到了11个。我们用这16个50口径的探针。直观地说,这组探针应该提供足够强的匹配模式来避免扩展,但是这个假设需要实验验证。数字1说明模式匹配策略的概述。
需要注意的是,所使用的值(95%相似度,50列,50毫秒)将根据应用模式匹配策略的序列类别而变化。
LINE-1模式检测算法
LINE-1模式检测(L1PD)算法是一种模式匹配策略的计算机实现,用于检测给定目标基因组中可以分类为l1的字符串片段。
上述16个50英寸探针,以及它们的5 '到3 '方向和平均偏移距离,在L1PD中实现为一个查找表。该算法使用mrFAST将探针映射到目标基因组[15,16],这是一种快速映射算法,强调发现结构变异和分段重复。在播种探针之后,L1PD找到所有的序列米或者更多符合模式匹配的种子,并返回它们在目标基因组中的位置。
L1PD的输入为米,t而且\三角洲(\ \),以及目标基因组。它的输出是分类为L1的所有片段在目标基因组中的位置。
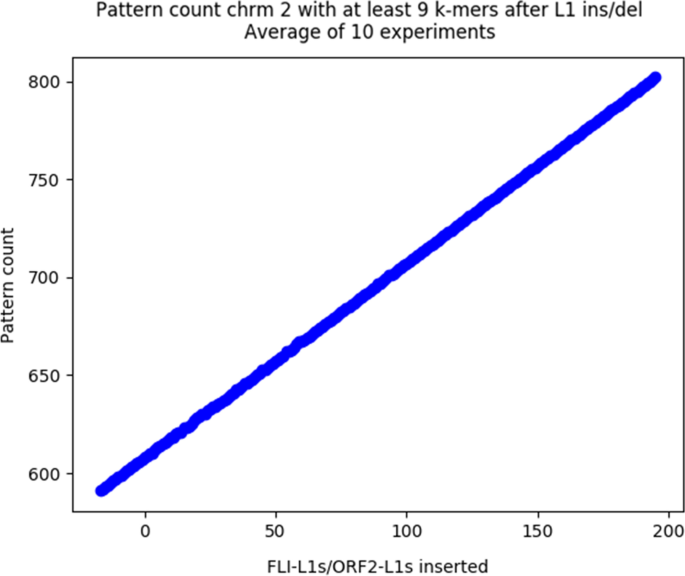
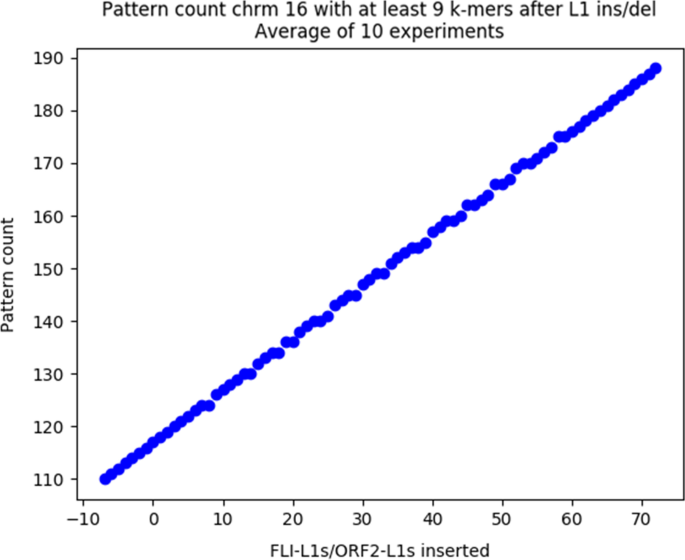
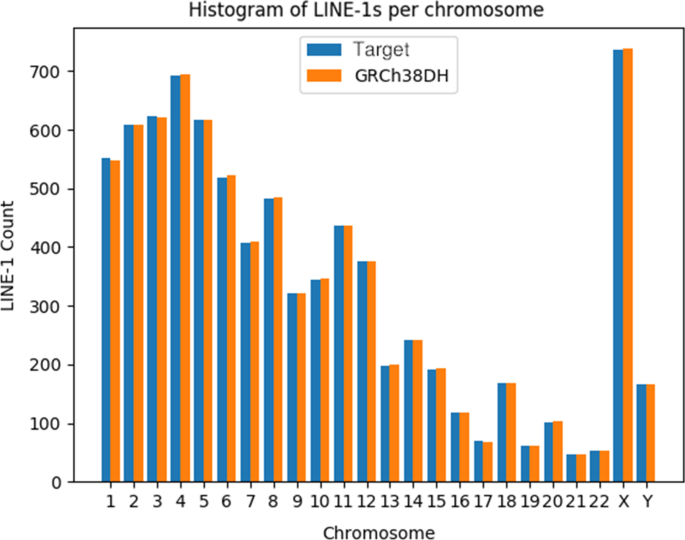
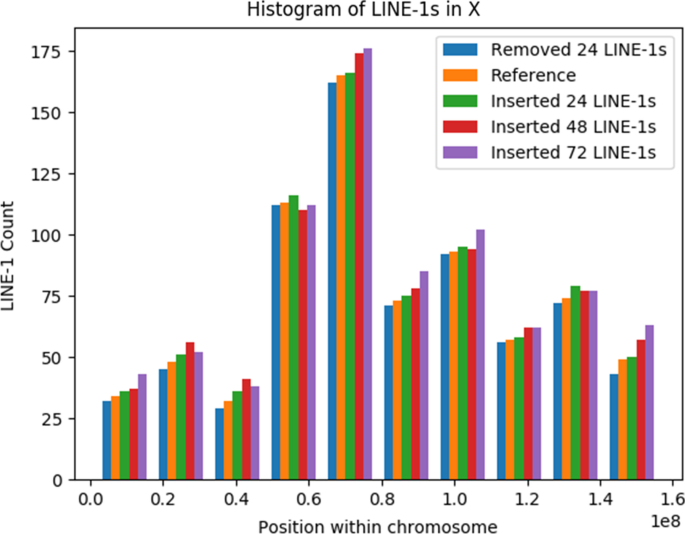
我们进行了第一次测试\(\delta = 5\),\(t = 50\),米范围从2到最大16。该测试旨在评估模式匹配和l1之间的对应关系。为此,我们在GRCh38基因组的不同染色体上随机添加或删除orf完整的l1和orf2完整的l1,从而导致l1改变的基因组。我们对每个l1改变的基因组进行了L1PD,并比较了模式匹配的数量和修饰目标中l1的数量。结果如图所示。2而且3..图表显示了模式匹配和L1计数之间的线性相关,验证了模式匹配和L1的对应关系。
为\ (m = 2 \)来\ (m = 14 \),所有146个orf完整的l1均被准确检测到,但与\ (m = 15 \)而且\ (m = 16 \)其中一个orf完整的l1未被检测到。这个orf完整的L1在其ORF2中删除了大约396个碱基。因此,探针对齐的位置被移动了大于阈值50的量,并且没有获得模式匹配\(m > 14\).
我们进一步研究了L1PD对输入参数的敏感性。我们在该方法检测L1Base2中所有l1的能力上下文中做到了这一点。也就是说,有146个orf完整,107个orf2完整,以及13418个未完整的l1。虽然orf完整l1和orf2完整l1确实在复制粘贴活性中起主要作用,但L1Base2文档中的一个注释指出,非完整l1也包括在内,因为一些“可能保留了表达的能力,尽管频率较低,但可能被逆转录转位活性全长完整l1编码的蛋白质反转录转位”[4].我们根据L1PD输出的精度和召回率评估了这种敏感性。
在信息检索中,精确一词指的是尽量只获得相关的结果(真阳性),尽量减少不相关的结果(假阳性)。另一方面,回忆一词指的是试图获得所有相关的结果(真阳性),而不遗漏任何相关的结果(假阴性)。关系式为:
在这种特殊情况下,精度计算为L1PD检测到的真实l1在L1PD报告的所有l1中所占的比例,使用L1Base 2 [5,6]作为参考,以确定哪些是真正的l1。类似地,召回率计算为L1PD检测到的真实l1在基因组中所有l1中所占的比例。
由于DNA的性质不同,我们的算法需要考虑可能的插入或删除,这意味着不是所有的探针都能被找到。因此,需要在模式中建立最少的探测命中量。正如预期的那样,较高的最小探针数量减少了假阳性的数量,但也减少了真阳性的数量。最终结果是,更高的最小探针数量导致更高的精度,但更低的召回率。F1评分(也称为F评分或F测量),基于Van Rijsbergen的有效性测量[17,是精度和召回率的调和平均值,用于建立平衡,因为它惩罚极值。公式为:
F1分数是记录分类或查询分类性能的标准度量,我们用它来确定L1PD输入的一组默认值。
用数学术语来说,我们的问题是为实值映射找到最大值
在哪里\(2\le m \le 16\),\(25 \le t \le 800\)而且\(5 \le \delta \le 30\).800的界限为t30美元\三角洲(\ \)实验定义。作为地图f取其在有限离散集上的值,通过直接计算可以找到最大值。作为t在25到700的范围内增加,F1分数也是如此(除了F1分数稳定在一个水平)\(t = 675\),和另一个之间的区别\(t = 675\)而且\(t = 700\)可以忽略不计)。一次t超过700,F1分数开始下降(同样,除了前面提到的两个例外)。另一方面,F1分数也随着年龄的增长而增加\三角洲(\ \),最高为\三角洲(\ \)接近读取长度的50%。表中显示了实际结果的部分清单1.
我们的计算表明,对于几乎每一个值\三角洲(\ \)时,F1分数的最大值为\(t = 700\),不管价值米.唯一的例外是\(\delta = 10\), F1的最高分数是在这里取得的\(t = 675\).因此,我们将700设置为默认阈值。我们还发现,对于几乎每一个值\三角洲(\ \)时F1得分最高\(m = 9\).唯一的例外是\(\delta = 5\)F1的最高分数是在\(m = 7\).因此,我们设\ (m = 9 \)为L1PD的默认值。最后,在比较了不同值的F1得分最高后\三角洲(\ \),我们发现F1总分最高的时候\(\delta = 20\).因此,我们将其设置为算法中mrFAST的默认编辑距离。
表格1显示了在搜索最大F1分数中获得的一些值,其中我们只包含了每个编辑距离和阈值组合的最高F1分数。更详细的列表可以在附加文件中找到1,可用于指导希望修改输入参数值以提高精度或查全率的用户。它还确定了这种修改所能达到的限度。
当前版本的L1PD将染色体组装成基因组作为输入。不幸的是,大多数公开可用的基因组数据库将其基因组数据存储在具有基因组短长度成对末端读取的FASTQ文件中。因此,对于这些数据库,需要一个耗时的组装管道,以GRCh38为参考,从FASTQ文件中重建目标基因组。该管道包括现有的工具,如BWA [18]、mrFAST、SAMtools和BCFtools [19],以及我们自己的剧本。我们的脚本是在Python 3中实现的,我们使用了Biopython [20.]模块在研究过程中的几个中间步骤,如生成k-mer探针。
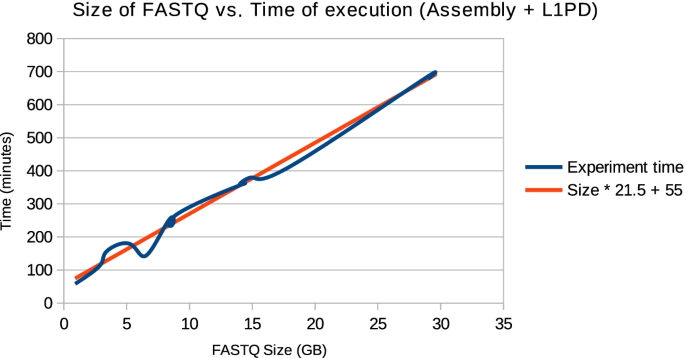
目标基因组组装所需的时间随着包含读取的FASTQ文件的大小而变化,对于大的组装可能是相当大的,如图所示。4.值得注意的是,一旦目标基因组组装完成,L1PD的运行时间是恒定的,如表所示2.L1PD的大部分时间用于标引目标基因组和绘制探针;模式匹配所需的时间可以忽略不计。类似地,内存使用由索引和映射阶段决定。当使用3.1GB的基因组时,索引使用了大约1.8GB的RAM,映射使用了大约2GB的RAM。
这些时间和内存测量,以及L1PD及其应用程序的所有其他运行,都是在一台Dell PowerEdge R740服务器上执行的®至强®黄金6138处理器。每个处理器有40个内核,运行频率为2GHz, L3缓存为27.5MB。该服务器有512GB的DDR4 RAM和5个SSD SATA Mix硬盘驱动器,每个硬盘都有800GB的存储空间,速度最高可达6Gbps。L1PD使用Python编写,系统运行在Ubuntu 18.04.3 LTS上。
与BLASR和MUMmer4的比较
前面我们讨论了用于检测l1的种子-扩展策略的缺点,以BLAST为例。另外两个使用种子-扩展的示例是BLASR和MUMmer4。
BLASR图谱读取长达数千个碱基,“读取和基因组之间的差异主要由插入和删除错误所主导”[21].由于我们的目标是检测l1,为了向BLASR提供所有l1作为查询序列,有必要将L1Base2中的所有序列收集到一个文件中。一旦这项初步工作完成,BLASR就以合理的精度和执行时间检测到了l1。我们的结果是在指定最小相似百分比为100%(使用-minPctSimilarity 100参数)。
MUMmer4是一种基因组校准器,最初设计用于校准细菌基因组。这种方法能够处理任何生物学上真实长度的基因组[22].MUMmer4由两条主要管道组成,一条用于排列核苷酸序列(nucmer)和一种用于排列蛋白质序列(毕业舞会).为BLASR使用而创建的包含所有l1的文件交给了nucmer,以确定如何找到l1。只有13,671个序列被搜索,尽管执行时间合理,但有679,560个结果,大约是l1数量的50倍。
L1PD检测l1只需要16个50-mers,不需要作为输入。我们想知道使用同样的50个mers作为BLASR的输入会有什么结果[21]和MUMmer4 [22].BLASR的结果是516次命中,这是非常少的,因为有16个探针,每个探针都应该在146个orf完整的l1中检测到(\(16 \乘146 = 2,336\)).反过来,MUMmer4没有返回任何结果,无论是设置为查找表示默认值的最大唯一匹配(MUM),还是设置为查找最大精确匹配(MEM)。考虑到BLASR只有在提供L1Base2中所有L1序列作为输入时才能检测到所有L1,本实验清楚地显示了L1PD在信息最少的情况下检测L1的高效。表格3.总结了这些结果,同时附加文件2提供更多详细信息。
L1PD应用程序
接下来,我们将简要描述在L1PD基础上实现的应用程序。
注释
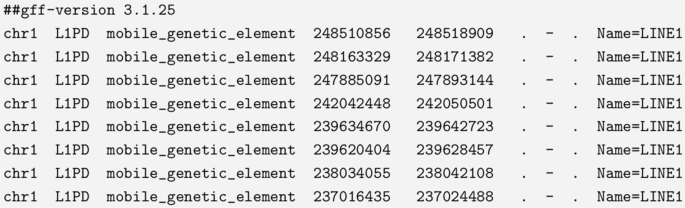
L1PD输出生成为通用特征格式版本3 (GFF3)文件,这是用于基因组注释的格式。GFF3将基因组信息特征存储在9个以制表符分隔的文本列中。在9列中,L1PD填充了以下7列:
序列id(发现LINE-1的染色体)
源(“L1PD”)
类型(“mobile_genetic_element”)
start (LINE-1的起始位置)
end (LINE-1结束位置)
链(“+”表示正向链,“-”表示反向链)
属性(“Name = LINE1”)
在GFF3格式中,链字段前面是分数字段,后面是相位字段。L1PD不计算这些字段,因此,我们将它们留空。由L1PD生成的GFF3文件被GFF3在线验证器视为有效[23].数字5显示了L1PD输出文件的第一行示例。
拷贝数变化
如前所述,目标基因组中l1的拷贝数变异(CNV)可能对疾病的诊断和预后至关重要。CNV的可靠性取决于l1的精确计数,例如L1PD提供的计数。我们将CNV表达为GRCh38基因组中L1拷贝获得或丢失的百分比。我们称之为CNV增益百分比(CNVPG),定义如下:
在哪里程控主题图案算和RPC是引用模式计数。
我们的应用程序使用GCRh38作为参考,但是用户可以用任何感兴趣的基因组来替换它。
我们计算了1000基因组计划中存储为FASTQ文件的基因组的CNVPGs [13].表格4显示得到的一些CNVPG值。正数代表收益,负数代表损失。
LINE-1插入的分布
使用matplotlib Python模块,我们生成了目标基因组和GRCh38DH的每条染色体L1计数的直方图,GRCh38DH是1000基因组计划用于解释诱饵序列、替代单倍型和eb病毒(EBV)的GRCh38基因组版本。
数字6下图为L1PD生成的PNG格式直方图。的x柱状图中的-轴表示参考基因组的染色体,每条柱表示该特定染色体的L1拷贝数。除了CNVPG的可视化之外,这些直方图还可以可视化目标基因组上L1插入位点的分布。
此外,这些直方图有助于L1异常计数的可视化。数字7比较L1副本被随机移除或添加的染色体的L1计数,其中x轴表示检测到L1的染色体中的位置(偏移量)。这种可视化在寻找疾病表型和L1拷贝数之间的相关性时特别有用,就像在肿瘤发展和进展过程中看到的那样。
结论
本文提出了一种替代传统的种子-扩展策略来研究人类基因组中存在的l1。我们认为,模式匹配非常适合于检测具有共同子序列的基因组片段,但在宿主生物的生命中受到截断或片段的增益和损失的影响。该策略将一组探针片段播种到目标基因组上,并在种子中搜索特征模式。通过这种方式,可以避免与种子和扩展方法相关的启发式元素,使用模式匹配代替扩展阶段(种子和模式匹配)。
我们讨论了L1PD的结果,这是一种用于识别目标基因组中L1片段的模式匹配策略的软件实现。我们的结果表明,所选择的探针和相关模式成功检测到所有orf完整和orf2完整的l1。通过调整L1PD的输入参数米,t,\三角洲(\ \),有可能检测到更多的非完整l1,但这增加了假阳性的数量(精度vs.召回率)。
我们还研究了L1PD作为GFF3输出的注释工具,以及L1PD在估计L1拷贝数变化时的使用,以及在给定染色体中L1插入的分布。
我们还利用合成数据评估了L1 pd检测L1拷贝数量变化的能力。我们的结果验证了使用L1 pd作为建立L1拷贝数和L1进化阶段之间可靠相关性的工具,就像在L1相关疾病的进展中所看到的那样。
综上所述,模式匹配策略可以有效地用于L1基因组片段的检测。它在L1PD中的实现在时间和计算空间方面都是一种经济的方法。L1PD算法的当前运行时间几乎与基因组的大小无关。然而,如果需要从存储为FASTQ文件的读取中组装基因组,则管道的性能会受到影响。
用75默探针代替50默探针可提高L1PD的检出率。事实上,正如Phan等人报道的那样,使用75-mers进行mrFAST可导致显着的回忆改善。[24].他们的50分钟测试结果与L1PD获得的记忆力相当。在我们的实现中,我们决定保留50-mers,因为在L1Base2数据库的orf对齐中,具有95%或更多碱基重复列的75-mers比50-mers要少得多。因此,将75-mers更改为75-mers将导致探针数量减少,这反过来又使得很难找到足够强大的模式来自信地消除扩展。通过放宽对探针的一些其他要求,可以解决这个问题。例如,将95%的列相似度降低到更低的百分比。
使用mrFAST是因为它关注结构变化。然而,可以尝试一些最新的校准器,如PuffAligner [25],以了解能否实现改善。此外,L1PD的未来版本可能会包含VCF/BCF模式,这样用户就可以从提供他们自己的. VCF/开始。bcf命令文件。
可用性
L1PD的源代码可在以下地址获得:知识共享署名相似共享4.0许可证https://github.com/juan-lopez/L1PD.该代码由几个shell脚本、一个Python脚本、一个带有探测的FASTA文件以及示例输出文件组成。shell脚本应该在大多数类unix系统下运行。
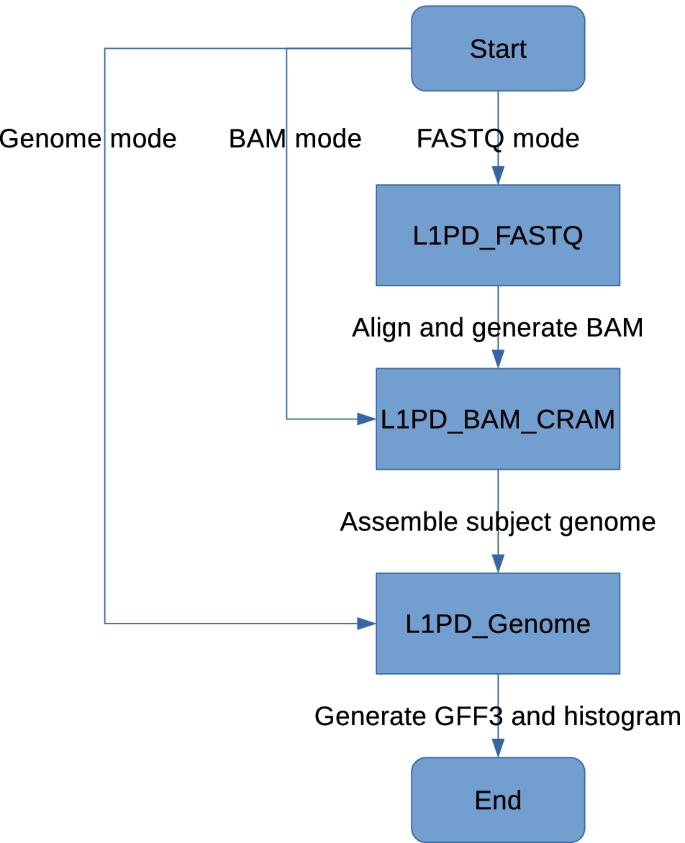
L1PD可以以以下三种模式之一执行:
基因组模式
BAM /补习模式
FASTQ模式
BAM/CRAM模式自动调用Genome模式,FASTQ模式自动调用BAM/CRAM模式,如图所示。8.
软件需求和所需的输入
基因组模式对于总是执行(直接或间接)的Genome模式,必须安装mrFAST,以及Python 3以及Matplotlib和Numpy包。在这种模式下,L1PD唯一需要的输入是受试者基因组,尽管还有其他可选的输入。
BAM /补习模式BAM/CRAM模式需要安装BCFtools(版本1.11或更新)。这种模式所需的输入是一个BAM/CRAM文件和用于比对的相应参考基因组,尽管还有其他可选的输入。
FASTQ模式FASTQ模式需要安装BWA和Samtools(1.11或更新版本)。这种模式所需的输入是成对的FASTQ文件和一个参考基因组,尽管还有其他可选的输入。
FASTQ模式是为那些以FASTQ格式开始读取并且没有完全组装基因组的用户提供的一种选择。然而,用户可以选择使用不同的无引用组装策略,然后在Genome模式下使用组装的数据集。
数据和材料的可用性
L1PD的源代码可在以下地址获得:知识共享署名相似共享4.0许可证https://github.com/juan-lopez/L1PD.
缩写
- CNV:
-
拷贝数变化
- CNVPG:
-
增益拷贝数变化百分比
- EBV:
-
巴尔病毒
- GFF3:
-
通用特性格式版本3
- L1, 1号线:
-
长穿插元素1
- L1PD:
-
LINE-1模式检测
- MEM:
-
最大精确匹配
- 米尔:
-
哺乳动物范围内穿插重复
- 妈妈:
-
最大唯一匹配
- 子:
-
开式阅读架
- sin:
-
短的穿插元素
- 股东价值分析:
-
sin, VNTR和Alu
- UTR:
-
翻译区
- VNTR:
-
可变数串联重复
参考文献
Belancio VP, Deininger PL, Roy-Engel AM。人类基因组中的直线舞蹈:转座因子和疾病。基因组医学。2009;1:7 7。https://doi.org/10.1186/gm97.
Kazazian HH Jr, Moran JV。L1逆转录转座子对人类基因组的影响。Nat Genet, 1998; 19:19-24。https://doi.org/10.1038/ng0598-19.
汉克斯DC, Kazazian HH Jr.逆转录转座子插入在人类疾病中的作用。暴徒的DNA。2016年,七章。https://doi.org/10.1186/s13100-016-0065-9.
Penzkofer T, Dandekar T, T Z. L1Base:从功能注释到活动LINE-1元素预测。核酸Res. 2004; 33:498-500。https://doi.org/10.1093/nar/gki044.
L1Base 2。7 - 9月- 2020年访问。http://l1base.charite.de/
Penzkofer T, Jäger M, Figlerowicz M, Badge R, Mundlos S, Robinson PN, Zemojtel T. L1Base 2:更多逆转录转座活性LINE-1s,更多哺乳动物基因组。核酸决议2016。https://doi.org/10.1093/nar/gkw925.
Hormozdiari F, Hajirasouliha I, Dao P, Hach F, Yorukoglu D, Alkan C, Eichler EE, Cenk Sahinalp S.下一代VariationHunter:转座子插入发现的组合算法。生物信息学,2010,26(12):350 - 7。https://doi.org/10.1093/bioinformatics/btq216.
Lee E, Iskow R, Yang L, Gokcumen O, Haseley P, Luquette LJ III, Lohr JG, Harris CC, Ding L, Wilson RK, Wheeler DA, Gibbs RA, Kucherlapati R, Lee C, Kharchenko PV, Park PJ。癌症基因组图谱研究网络:人类癌症中体细胞反转录转位的景观。科学。2012;337(6097):967 - 71。https://doi.org/10.1126/science.1222077.
王凯,王德安。RetroSeq:基于新一代测序数据的转座因子发现。生物信息学。2012;29(3):389 - 90。https://doi.org/10.1093/bioinformatics/bts697.
吴杰,李W,沃德A,沃克J,孔克尔M,巴茨米高梅。Tangram:一个用于移动元素插入检测的综合工具箱。BMC Genom, 2014。https://doi.org/10.1186/1471-2164-15-795.
Steinbiss . Repeat M. 2021年5月25日访问。http://www.repeatmasker.org/
Altschul S, Gish W, Miller W, Myers E, Lipman D.基本的局部对齐搜索工具。中华分子生物学杂志,1990;215(3):403-10。https://doi.org/10.1016/s0022 - 2836 (05) 80360 - 2.
Clarke L, Fairley S, Zheng-Bradley X, Streeter I, Perry E, Lowy E, Tassé a - m, Flicek P.国际基因组样本资源(IGSR):包含1000个基因组计划数据的全球基因组变异集合。南京大学学报(自然科学版)2016;45(D1): 854-9。https://doi.org/10.1093/nar/gkw829.
Babaian A, Ebou A,等。bioSyntax:计算生物学的语法高亮显示。BMC Bioinform, 2018。https://doi.org/10.1186/s12859-018-2315-y.
李志强,李志强,李志强,等。使用下一代测序的个性化拷贝数和分段复制图。Nat Genet, 2009; 41:1061-7。https://doi.org/10.1038/ng.437.
李志强,李志强,李志强,李志强。使用fastthash加速读映射。BMC genome . 2013;14(增刊1):13。
van Rijsbergen CJ。评估。见:信息检索,第2版。Butterworth-Heinemann:格拉斯哥,苏格兰;1979,第112-140页。
李华,杜斌。基于Burrows-Wheeler变换的快速精确短读对齐。生物信息学,2009,25(14):1754 - 60。https://doi.org/10.1093/bioinformatics/btp324.
Li H, Handsaker B, Wysoker A, Fennell T, ruj, Homer N, marg, Abecasis G, Durbin R, GPDP子群。序列对齐/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。https://doi.org/10.1093/bioinformatics/btp352.
Cock PJA, Antao T, Chang JT, Chapman BA, Cox CJ, Dalke A, Friedberg I, Hamelryck T, Kauff F, Wilczynski B, de Hoon MJL。Biopython:用于计算分子生物学和生物信息学的免费Python工具。生物信息学,2009,25(11):1422 - 3。https://doi.org/10.1093/bioinformatics/btp163.
Chaisson MJ, Tesler G.利用基本局部比对逐次细化(BLASR)绘制单分子测序reads:应用与理论。BMC Bioinform, 2012。https://doi.org/10.1186/1471-2105-13-238.
Marçais杨晓东,张晓明,张晓明,等。MUMmer4:一个快速和通用的基因组比对系统。PLOS计算生物学,2018。https://doi.org/10.1371/journal.pcbi.1005944.
斯坦比斯S. GFF3在线验证器。7 - 9月- 2020年访问。http://genometools.org/cgi-bin/gff3validator.cgi
潘伟,高胜,陈强,等。基因组的复杂性如何解释将读取数据与基因组对齐的困难。BMC Bioinform 2015。https://doi.org/10.1186/1471-2105-16-S17-S3.
Almodaresi F, Zakeri M, Patro R. PuffAligner:基于河豚鱼指数的快速,高效和准确的校准器。生物信息学。2021;37(22):4048 - 55。https://doi.org/10.1093/bioinformatics/btab408.
确认
作者要感谢德克萨斯a&m大学的助理研究科学家Chris Blazier,他建议将BLASR和MUMmer进行比较。本文部分得到了位于阿雷西博的波多黎各大学的资助。
资金
这项工作得到了美国国立卫生研究院拨款2P20GM103475-16的部分支持。
作者信息
作者及隶属关系
贡献
进行研究以准确检测人类基因组中的LINE-1的想法最初是由KSR提出的。模式匹配策略最初是由JS作为NIH INBRE 2P20GM103475-16拨款的子项目提出的。JOL开发并实现了该方法。AC合作开发了这个项目。所有作者都参与了研究结果的解释和这篇手稿的写作。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
所有的方法都是按照相关的指导方针和规定进行的。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
洛佩兹,j.o.,塞格尔,J.,查莫罗,A。et al。人类基因组中LINE-1s的高精度检测模式匹配。BMC生物信息学23375(2022)。https://doi.org/10.1186/s12859-022-04907-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04907-4
关键字
- 1号线
- 人造石铺地面
- K-mer
- 探针