摘要
背景
准确预测药物靶标结合亲和力(DTA)在药物发现中具有重要作用。大多数用于预测DTA的计算方法使用机器学习模型,特别是深度神经网络,并且依赖于大规模标记数据。然而,仅基于相对有限的标记药物靶标数据,很难从数千万种化合物和数十万种蛋白质中学习到足够的特征表示。存在大量未知药物,这些药物从未出现在标记的药物靶标数据中。这是生物医学领域的一种非流通问题。最近的一些研究采用自监督预训练任务来学习氨基酸序列的结构信息,以增强蛋白质的特征表示。然而,预训练与DTA预测之间的任务差距带来了灾难性遗忘问题,阻碍了特征表示在DTA预测中的充分应用,严重影响了模型对未知药物发现的泛化能力。
结果
为了解决这些问题,我们将预训练和多任务学习相结合,提出了一种新的面向未知药物发现的DTA预测模型GeneralizedDTA。我们引入自我监督的蛋白质和药物预训练任务,从蛋白质的氨基酸序列和药物化合物的分子图中学习更丰富的结构信息,以缓解基于深度神经网络编码导致的高方差问题,加快预测模型在小尺度标记数据上的收敛速度。我们还开发了一个具有双重适应机制的多任务学习框架,以缩小预训练和预测之间的任务差距,防止过拟合,提高DTA预测模型对未知药物发现的泛化能力。为了验证模型的有效性,我们构建了一个未知药物数据集来模拟未知药物发现的场景。实验结果表明,与现有的DTA预测模型相比,我们的模型在未知药物的DTA预测中具有更高的泛化能力。
结论
我们模型的优势主要在于两类预训练任务和多任务学习框架,可以从大规模的未标记数据中学习到更丰富的蛋白质和药物的结构信息,然后将其有效地整合到下游预测任务中,从而在未知药物发现中获得高质量的DTA预测。
背景
传统的湿式实验室药物发现效率非常低[1,2]。它通常花费10到17年的时间和数十亿美元用于研究和实验过程[3.]。这种低效的流程显然难以满足新冠肺炎等快速发展疾病的需求。为了提高药物发现的效率,用计算机预测药物-靶标相互作用(DTI)越来越受到人们的关注。2,4,5,6,7]。这些计算DTI预测方法不仅成本低,而且可以大大加快药物开发的进程[8]。
预测药物-靶标结合亲和力[9]是一种特殊的DTI预测任务。与传统基于二值分类的DTI预测不同,DTA预测可以获得药物与靶点之间的定量结合亲和力,更详细地描述药物与靶点的相互作用。相关研究主要采用机器学习模型实现编码和解码两阶段的建模过程。编码过程从药物和各种目标(如蛋白质)中学习特征表示。解码过程根据这些特征表示来预测绑定关联。早期的研究通常采用浅层机器学习模型来学习DTA预测的特征表示。SimBoost [10]通过协同滤波计算药物化合物与靶点之间的亲和相似度,并将相似度作为特征向量预测DTA。KronRLS [11]使用基于核的方法生成药物的分子描述符。随着深度学习的迅速发展,深度神经网络在DTA预测中得到了广泛的应用,尤其是在编码过程中。DeepDTA [12]首次将深度学习引入DTA预测,使用卷积神经网络(CNN)生成药物和蛋白质的一维表示。GraphDTA [13]使用开源化学信息学软件RDKit代替化合物串构建药物化合物的分子图,并利用图神经网络学习药物化合物的特征向量。MGraphDTA [14]建立了一个具有27个图卷积层的超深度GNN,以同时捕获化合物的局部和全局结构。马特\ \ _ \ ()DTI [15通过关系感知的自我注意块对药物化合物原子之间的相关性进行编码,并通过多头注意块对药物表征和目标表征的相互作用进行建模。DeepNC [16]分别通过GNN层和1-D卷积网络层学习药物和靶标的特征。MINN-DTI [17将交互变压器模块与改进的通信信息传递神经网络(CMPNN)相结合,以更好地捕捉药物和靶标之间的双向影响。除了药物和蛋白质的特征编码外,特征聚集也引起了人们的关注。FusionDTA [18利用一种新的多头线性注意机制,基于注意权重聚合全局信息。
上述研究均基于标记药物靶标数据集,如Davis [19]和Kiba [20.]。与数以千万计的化合物和数十万种蛋白质相比,标记的药物靶标数据相对有限。戴维斯数据集[19]只包含72种药物和442个靶点。KEGG数据集[21]总共只有4797个药物靶标对。然而,ZINC15数据库[22]含有超过2.3亿种待售化合物。仅基于相对较小的标记药物靶标数据,很难学习涵盖所有药物和化合物的特征表示。针对这一问题,Hu等[23]对大量未标记的数据进行蛋白质预训练任务,获得具有增强氨基酸序列结构信息的鲁棒蛋白质编码模型,然后在解码过程中对编码模型进行微调,即DTA预测建模过程,拟合相对较小的标记药物靶标数据。由于增强了结构信息,他们的DTA预测模型取得了很好的结果。
然而,Hu等人的模型仅利用蛋白质预训练任务获得氨基酸序列的结构信息,忽略了药物化合物分子图的结构信息。更重要的是,预训练和DTA预测之间存在任务差距。蛋白质预训练的目标是根据氨基酸序列的上下文信息准确预测被掩盖的氨基酸,而DTA预测的目标是准确计算药物化合物与蛋白质的结合亲和力。Hu等人采用顺序结构将预训练任务与DTA预测任务相结合[23]。两者之间的任务差距会导致灾难性的遗忘问题[24]。随着微调迭代次数的增加,下游预测模型越来越关注标记的药物靶标训练数据中频繁出现的药物和蛋白质,导致对那些从未出现在标记的药物靶标数据中的未知药物的预测结果不佳。这是生物医学中的一种分布外问题[j]。25]。DTA预测模型泛化能力较差[26关于未知药物的发现。当标记的数据明显小于未标记的预训练数据时,这个问题尤其严重。
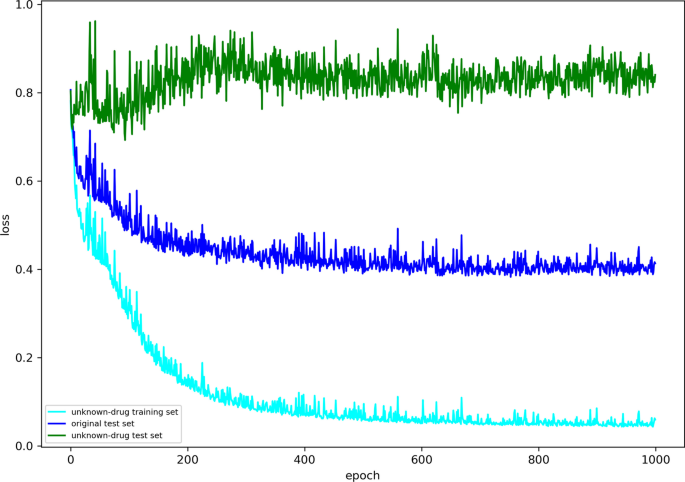
然而,现有的DTI和DTA预测研究并没有特别关注这些未知药物。据我们所知,未知药物发现中模型泛化能力差的问题尚未得到研究。为了证明这个问题的存在,我们使用Davis数据集对未知药物发现执行DTA预测任务。原始训练集和测试集的划分参考Öztürk等人的工作[13]。我们随机选择了20个\ \ % \ ()对原训练集中的药物,共有14种药物,作为新药。从原始训练集中删除所有对应的药物-目标对,构建未知药物训练集。从原始测试集中提取相应的5178个药物靶标对,构建未知药物测试集。未知药物发现中的DTA预测任务是在未知药物训练集和测试集上完成的。使用GraphDTA [12]迭代1000次,结果如下:
数字1给出了损失函数在1000次迭代中的收敛曲线。横轴表示迭代次数,纵轴表示损失函数值。从图中可以看出,在前200次迭代中,未知药物训练集和原始测试集上的损失显著减小,未知药物测试集上的损失在0.85处反复波动,没有下降趋势。这表明GraphDTA是过拟合的,对未知药物发现缺乏足够的泛化能力。有必要对这个问题进行专门研究。
在以往的研究中,可以通过数据增强、特征去除等手段来干预模型的过拟合。对于DTA预测任务,由于需要增加标记的药物靶标数据,数据增强成本太高。特征去除会降低模型的准确性,偏离预训练特征增强的初衷。基于以上观察,本研究提出了一种将自监督预训练与多任务学习相结合的新的DTA预测模型——GeneralizedDTA。主要贡献可总结如下:
- (1)
首先,本研究在DTA预测任务中引入了蛋白质和药物的预训练任务。利用这两种预训练任务,首次学习到蛋白质氨基酸序列和药物化合物分子图谱的结构信息,并将其整合到DTA预测任务中。
- (2)
其次,本研究建立了具有双重适应机制的多任务学习模型,以缓解预训练参数的灾难性遗忘问题。采用基于maml的更新策略,对预训练参数进行少量梯度更新,然后利用更新后的参数对整个模型进行下游DTA预测任务的训练,以加快收敛速度,防止模型陷入局部最优。
- (3)
第三,构建一组未知药物数据集,模拟未知药物发现场景,并对这些数据集进行对比实验。实验结果表明,与现有的DTA预测模型相比,我们的模型的泛化能力有了显著提高。它可以更好地适应于未知药物发现中的DTA预测。
方法
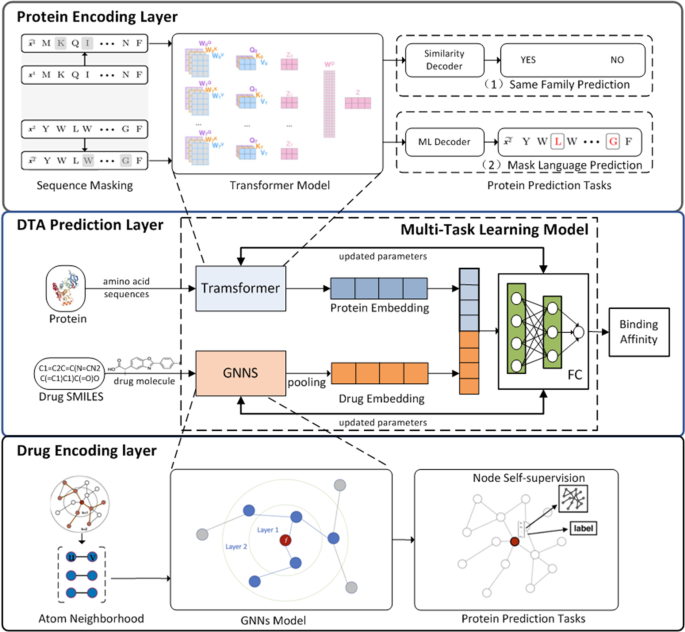
为了实现未知药物发现中的DTA预测,本研究提出了自监督预训练与多任务学习相结合的概化DTA模型。采用两种蛋白质预训练任务来学习氨基酸序列的结构信息。设计了一种新的药物预训练任务来学习药物化合物分子图的结构信息。为了缓解预训练参数的灾难性遗忘问题,提出了一种具有双重适应机制的多任务学习框架,以防止预测模型陷入过拟合。数字2给出了广义DTA的模型体系结构,包括四个模块:蛋白质编码层、药物编码层、DTA预测层和多任务学习框架。
蛋白质编码层
蛋白质编码层利用蛋白质预训练任务编码蛋白质的氨基酸序列作为载体。灵感来自BERT [27],本研究采用以多头注意力为编码器的变压器模型接收氨基酸序列。给定一个氨基酸序列\(t=\left[t_{1}, \ldots, t_{n}\right]\)在哪里\(t_{i} \in \{21\)氨基酸类型\ \} \ (),变压器模型将其转换为\(z=\left[z_{1}, \ldots, z_{n}\right]\)如下:
在哪里\(问\ R ^ {d_ {1} \ * d_ {2}}, K \ R ^ {d_ {1} \ * d_ {2}}, V在R ^ \ {d_ {1} \ * d_ {2}} \)是注意力的参数,n是正面的个数,\(W^{0}\ in R^{d_{1}} \乘以d_{1}\)正面的重量是和\ \√{d_ {k}} \)为q的维数。自关注函数同时对所有键的每个查询的点积进行计算,并除以softmax函数得到值[28]。它可以简化为参数化函数Transformer (\子弹(\ \)),并设置参数\θ(\ \):
本研究基于转换模型,采用两个预训练任务获取蛋白质氨基酸序列的结构信息。
掩码语言建模(MLM)任务[28这个任务随机筛选一些氨基酸并预测它们的类型。给定一个被掩盖的氨基酸序列t和一个被掩盖的氨基酸组\ (m = \ \ {m_ {1}, m_ {2}, \ ldots m_ {N} \右\}\),传销解码器计算的日志概率t如下:
在哪里\(F C(\子弹)\)是一个具有参数的全连接神经网络(FC)\(θ_ {1}\ \)和\ (m ^{\ '} = \左\ {m_ {1} ^ {\ '}, m_ {2} ^ {\ '} \ ldots m_ {N} ^{\ '} \右\}\)表示整个屏蔽氨基酸集的预测氨基酸集。然后将对数似然函数作为MLM任务的评价指标:
通过上述MLM任务,转换模型可以有效地学习蛋白质氨基酸序列的双向上下文表示。
同族预测(SFP)任务[29,30.]:该任务使模型能够确定两个蛋白质是否属于同一家族。为了使用SFP任务对变压器模型进行预训练,本研究选择了两个氨基酸序列\ (t ^ {1} \)和\ (t ^ {2} \)从Pfam数据集。采用随机抽样,保证来自同一类和不同类的概率相同。瞄准蛋白质对\(左\ \ langle \ mathrm {t} ^ {1}, \ mathrm {t} ^{2} \ \纠正\),有辍学的FC [31]来计算它们的相似度值:
在哪里\(\theta _{2} \in {\mathbb {R}}^{|z| \times 2}\)为FC的参数,左\ (z_p = \ [z_ {1} ^ {1}, \ cdots z_ _ {1} {1} ^ {1}, z_ {1} ^ {2}, \ cdots z_ {n_{2}} ^{2} \右]z \ {\ mathbb {R}} ^ {z | | \ * 1} \)向量表示的是\(左\ \ langle \ mathrm {t} ^ {1}, \ mathrm {t} ^{2} \ \纠正\)和\({\hat{c}} \in {\mathbb {R}}^{2 \times 1}\)为预测的相似性值,即蛋白质对属于同一蛋白质家族的概率。SFP任务训练模型以最小化交叉熵损失,该交叉熵损失旨在处理概率上的预测误差。因此,本研究采用对数似然函数来测量SFP损耗:
由于变压器模型被要求对来自同一家族的蛋白质产生更高的相似性值,SFP任务使变压器模型能够更好地吸收蛋白质氨基酸序列的全局结构信息。
药物编码层
药物编码层通过一种全新的药物预训练任务,将药物化合物的分子图编码为载体。它采用GCN [32从药物化合物的分子图中挖掘潜在的关系。
给定药物化合物的分子图\ ({\ mathcal {G}} = ({\ mathcal {V}}, {\ mathcal {E}}, {\ mathcal {X}}, {\ mathcal {Z}}) \)在哪里\ ({\ mathcal {V}} \)是化学原子集,\ ({\ mathcal {E}} \)化学键是固定的,\ ({\ mathcal {X}} \在{\ mathbb {R}} ^{| \ν| \ * d_ {v}} \)和\ ({\ mathcal {Z}} \在{\ mathbb {R}} ^ {| \ varepsilon | \ * d_ {e}} \)分别是原子和键的特性集。GCN主要涉及每层每个原子的“更新”和“聚合”两个关键计算。它们可以表示为参数化函数\(\Psi (\bullet)\)通过参数\ψ(\ \):
在哪里\(u, v \in {\mathcal {v}}\)是两个化学原子,\ (z_ {u v} \)为化学键的特征向量(u,v),\ (\ mathrm {h} _ {v} ^ {0} = \ mathrm {x} _ {v} \在{\ mathcal {x}} \)是GCN的输入,表示原子的特征v,\ (\ mathrm {h} _ {v} ^ {l} \)表示原子的特征v在GCN的第1层,\ ({\ mathcal{一}}\)是药物化合物的邻接矩阵\ ({\ mathcal {G}} \),\ ({\ mathcal {N}} _ {v} \)邻域原子是原子的集合吗v。
以便得到药物化合物的表示形式\ ({\ mathcal {G}} \),使用最后一层GCN上的POOLING函数将分子图变换为向量:
在哪里\ (h_ {{\ mathcal {G}}} \)载体表示药物化合物吗\ ({\ mathcal {G}} \), POOLING是一个简单的池化函数,类似于max或mean-pooling [33,34]。为简单起见,我们将GCN表示为:
本研究在GCN模型的基础上,设计了一种新的预训练任务来学习药物化合物分子图的结构信息。
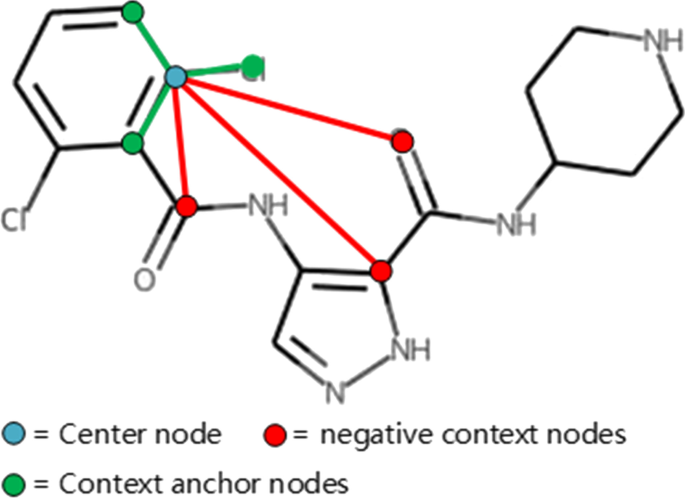
药物预训练(DP)任务:这项新任务旨在通过鼓励药物化合物分子图中相邻化学原子生成相似的嵌入来提高药物的表示学习能力[35]。聚合是GCN中每一层的关键计算。在化合物级聚集中,相邻的化学原子根据式()聚集它们的信息。10) [36,37]。对于每个化学原子\(v \in {\mathcal {v}}\), GCN的代表是\ (\ mathrm {h} _ {v} \)和\(\Psi (\bullet)\)在Eq. (10).因此,如图1所示。3.,给定一个随机的原子键u作为中心节点,自监督损失函数[38]来实现DP任务,即鼓励相邻化学原子的相似嵌入:
在哪里v是否直接连接到中心节点的上下文锚节点u,\ (v ^ {\ '} \)是没有直接连接到的否定上下文节点吗u,\ψ(\ \)为GCN参数,σ\ (\ \)是s型函数。通过5层GCN,每个原子包埋几乎吸收了分子图中所有的小局部结构[39,40]。
DTA预测层
DTA预测层是将药物化合物与蛋白质结合,预测其结合亲和力。本研究采用FC进行DTA预测。对于给定的药物-蛋白质对\(\left\ rangle {\mathcal {G}}, t\right\rangle\)在哪里\ ({\ mathcal {G}} \)药物化合物的分子图和t是氨基酸序列,对应药物化合物的载体\ ({\ varvec {h}} _ {\ mathcal {G}} \)蛋白质载体\ (z_p \)可由药物编码层和蛋白质编码层获得。然后,预测它们的结合亲和力的过程\({\帽子{y}} \)如下所示:
在哪里\γ(\ \)为全连接层参数,Concat (\子弹(\ \))表示输入是的连接向量\ ({\ varvec {h}} _ {G} \)和\ (z_p \)。
DTA预测任务训练模型最小化损失函数。本研究采用均方误差(mean squared error, MSE)作为损失函数:
在哪里\({\帽子{y}} \)药物-蛋白对的预测结合亲和力和y是真实值,\(\theta, \psi, \gamma)组合为模型参数。
具有双重适应机制的多任务学习框架
本研究采用多任务学习将编码器即预训练任务与解码器即DTA预测任务连接起来,防止在较小监督样本下由于局部最优性而导致的过拟合。为了使模型整体偏倚于主任务DTA预测,本研究采用了MAML的更新策略[41]。
将药物预训练任务定义为查询集。对于这个任务,我们调整了先验参数\ψ(\ \)具有一个或几个梯度下降步骤的化合物级聚合。设置学习率为\α(\ \)为双重适应。新的先验参数\(\psi ^{\prime}\)可获得如下:
然后是FC参数\γ(\ \)在DTA预测层中,定义为支持集,更新如下:
之后,通过多任务学习的整体损失函数的反向传播来更新所有参数。我们将总体损失函数定义为:
在哪里\(\lambda _{\text{atom}}\)手动设置的是药物预训练任务损失函数的权值。本研究采用梯度下降法更新所有可学习参数。在预训练药物任务之前,我们记录原始模型参数,并将预训练中第一次更新的参数(查询集)作为后续DTA预测的先验参数。将DTA预测与药物预训练任务的综合损失函数作为双适应的目标函数。后续原始参数通过多任务学习框架进行更新。与冻结策略不同的是,更新后的模型参数是原始参数,而不是先验参数。
双重自适应机制需要在预训练任务中保存所有可学习的参数。对于多头变形金刚学习来说,这将带来训练时间的巨大增加。此外,本研究主要针对未知药物,将药物预训练任务引入DTA预测。因此,本研究的多任务学习框架只是利用上述双重适应机制将药物预训练任务与DTA预测任务结合起来。
结果
数据准备
本研究对以下两个数据集进行了预训练任务:
蛋白质预训练数据集:Pfam数据集[42]用于蛋白质预训练。它是由欧洲生物信息学研究所利用基于UniProt的序列数据库制作的。根据蛋白质序列相似性,将超过21M个氨基酸序列聚为16479个家族。本研究对该数据集进行了两个蛋白质预训练任务,以学习氨基酸序列的结构信息。
药物预训练数据集:ZINC15数据库[22]用于药物预训练。它是由加州大学的欧文和休伊切特实验室提供的。在本研究中,通过药物预训练任务,使用200万种未标记化合物来学习药物化合物分子图的结构信息。
戴维斯夫妇[19]和Kiba [20.]进行绩效评价。Davis的数据集包括30056对药物靶标,涉及442种蛋白质和68种化合物。Kiba数据集包括118254对药物靶标,涉及229种蛋白质和2068种化合物。它们的结合亲和力由相关抑制剂用各自的解离常数值表示。构建了一组未知药物数据集,用于模拟未知药物发现的场景。这个过程包括以下两个步骤:
未知药物化合物/蛋白质的选择:本研究根据相似度选择未知药物化合物和蛋白质。指[43],我们对所有药物化合物进行基于子结构特征的k-means算法,并选择异常值作为未知药物化合物。指[25],我们根据Pfam家族选择了未知的蛋白质。从最小的42个家族中选择蛋白质作为未知蛋白质。
未知数据集构建:提取含有未知化合物或未知蛋白的药物靶标对作为未知测试集(Unknown - tes)。从原始训练集中移除相应的药物-靶标对[12],剩余数据用于构建未知训练集(unknown- trs)。
数据分布如表所示1。同样,我们可以从Kiba中获得未知药物数据集,如表所示2。在构建未知药物数据集后,我们从药物预训练数据集中删除了所有未知药物化合物,以避免数据泄漏。
模型参数
采用多任务学习框架和双重适应机制,先单独进行蛋白质预训练任务,然后同时进行药物预训练任务和DTA预测任务。对于蛋白质编码,设置氨基酸载体的维数为20,自注意头像数为12,隐藏层数为12,隐藏层的维数为768。对于药物编码,设置GCN的层数为5,隐藏层的维度大小为300。对于DTA预测,FC层数设置为3。对于多任务学习,学习率设为0.001,药物预训练权值分别设为0、0.5、1.0和2.0。
基线的方法
为了证明模型的有效性,本研究将提出的广义dta与以下基线方法进行了比较:
DeepDTA [12]:它使用CNN和池化架构来捕捉蛋白质和药物之间潜在的相互作用特征。研究表明,采用较少参数的CNN网络可以用于变压器过拟合测试。因此,本研究采用三层卷积对DeepDTA进行药物和蛋白质编码,核大小分别设为4、6、8。
GraphDTA [13:它将药品的SMILES字符串表示为短ASCII字符串。在本研究中,GraphDTA的药物编码采用三层图卷积,各层特征维数分别设置为78156312。这种增量参数设计可以增强原子间的信息传递。
SAGDTA [44]:利用药物分子图的自注意机制,获得药物的有效表征。在本研究中,分子图中每个原子节点的特征和SAG使用了3块的分层池架构,该架构已被证明可以更好地吸收全局信息。
MGraphDTA [14]:采用基于化学直觉的深度多尺度图神经网络进行DTA预测。构建了具有27个图卷积层的超深度GNN,以同时捕获化合物的局部和全局结构。在本研究中,学习比率和嵌入大小分别为5e-4和128。
评价指标
本研究采用MSE和r平方(\ (R ^ {2} \)) [45]来评估模型的预测结果。MSE和\ (R ^ {2} \)是定义良好的度量,用于度量回归任务中拟合线的接近程度。它们的计算方法如下:
在哪里\({\帽子{y}} _{我}\)绑定亲和度的真实值是多少我-药物-靶标对;\({\帽子{y}} \)是对应的预测值,和酒吧\ ({\ {y}} \)是所有绑定亲和力的真实值的平均值。
预测药物靶点结合亲和力的性能评价
表3.和4给出实验结果。可以看出,SAGNet和我们的模型具有\(\lambda _{\text{atom}}=0\)在两个数据集中表现最差。这表明没有附加辅助约束的深度网络在处理未知数据时表现较差。我们的模型\(\lambda _{\text{atom}}=0\),其中\(\lambda _{\text{atom}}=0\)表示药物预训练与DTA预测之间的不结合,MSE最大。这表明药物预训练与DTA预测之间由于灾难性遗忘而存在过拟合。有必要开发一个多任务学习框架来绑定预训练模型和预测模型。GraphDTA [13]取得了比DeepDTA更好的性能,说明基于药物化合物分子图的结构信息对DTA预测有价值。MGraphDTA [14]在四种基线方法中取得了最好的效果。这再次证明了化合物结构信息的重要性,这也是本研究引入基于图的药物预训练任务的重要动机。我们的模型\(\lambda _{\text{atom}}=0.5\)和\(\lambda _{\text{atom}}=1.0\)在Davis数据集和Kiba数据集的所有评估指标方面分别取得了最好的性能。这表明我们的模型采用了一种新的药物预训练任务,并通过多任务学习框架将其与DTA预测任务相结合,在未知药物发现方面具有更好的泛化能力。但是,不同的数据集可能需要不同的优化权重。其原因可以归结为不同数据集的亲和度测量方法不同。
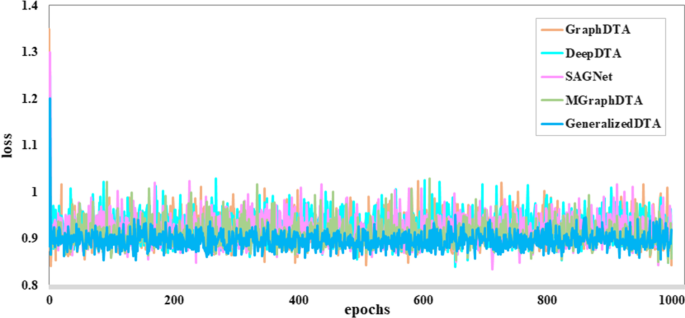
数字4给出了Davis给出的未知te的收敛性分析。如图所示,本文提出的GeneralizedDTA可以有效地收敛于来自Davis的未知te,具有最高的泛化能力。
讨论
消融实验
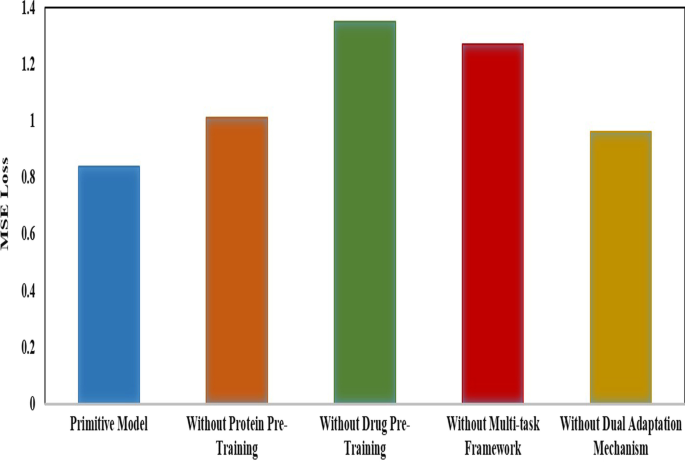
提出的广义dta结合了预训练和多任务学习。它涉及蛋白质预训练、药物预训练、多任务框架和双重适应机制四个核心组成部分。为了分析它们的有效性,我们设计了一项消融研究,包括4种消融变异,不进行蛋白质预训练,不进行药物预训练,不进行多任务框架,不进行双重适应机制。没有双重适应机制的变体是先完成预训练,然后将预训练好的组件转移到DTA中。对Davis的未知药物数据集进行了实验。
数字5给出了实验结果。如图所示,我们的方法优于所有的变体。这表明四个分量都能有效地改善DTA预测。药物预训练的效果最大,蛋白质预训练的效果较小。这表明药物化合物的结构信息在DTA预测中比蛋白质的结构信息更重要。数字5同时也表明,在多任务框架的约束下,深度学习模型的效果可以得到显著提高。双自适应机制可以防止深度学习模型参数更新中的局部最优性,提高DTA预测性能。
预训练模型的对比分析
预训练是广义dta的核心组成部分。本研究采用基于转化蛋白的蛋白质预训练和基于gcn的药物预训练。目前,有几种最先进的蛋白质预训练模型和药物预训练模型:
ESM (46]:它是一种蛋白质预训练模型,通过掩模语言建模和同源信息相关建模,使用具有自监督任务的非常大的深度模型框架。
DISAE [47]:它也是一种蛋白质预训练模型,在不了解结构和功能的情况下,利用所有蛋白质序列及其多序列比对来捕捉蛋白质之间的功能关系。
ContextPred [48]:它是一种药物预训练模型,探索节点级自监督任务和样本子图中的图结构分布,以预测其周围的图结构。
格罗弗(49]:它也是一种药物预训练模型,利用基于局部随机行走的目标,通过节点、边和图级的自监督任务来学习丰富的结构和语义信息。
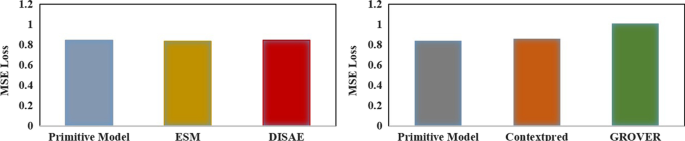
为了评估GeneralizedDTA的预训练,本研究使用上述预训练模型分别替换GeneralizedDTA的预训练成分。对Davis的未知药物数据集进行了实验。
数字6给出了实验结果。如图所示,对于蛋白质预训练,我们基于转换器的蛋白质预训练得到的DTA预测结果与基于ESM和DISAE的预测结果相差不大。这表明这些最先进的蛋白质预训练模型,如ESM,可以略微提高DTA预测,但并不显著。考虑到计算资源的需求,我们的蛋白质预训练是合适的,特别是在低资源环境下。
我们基于gcn的药物预训练采用节点级自监督任务。随机屏蔽节点和边缘属性[35],可以训练GCN模型生成能够区分原子相似性的图嵌入。基于这种图嵌入,可以有效地提高下游DTA预测模型的能力。如图所示。6基于我们基于gcn的药物预训练的DTA预测结果与基于ContextPred的预测结果相似,但明显优于GROVER。由于ContextPred也采用了节点级自监督任务,这表明我们基于gcn的药物预训练中节点级自适应周边邻居比GROVER中的随机游走策略更适合DTA预测。原因可能是随机漫步策略过于关注下游的不相关信息。
我们的模型的结果\(\lambda _{\text{atom}}=0\)在表3.结果表明,如果将预训练与预测解绑定,由于缺乏约束,模型对现有药物的学习过于精细,失去了对未知药物的预测能力。因此,本研究开发了一个具有双重适应机制的多任务学习框架,将药物预训练和DTA预测结合起来。在我们的双重适应机制中,GCN的参数并不是每次都是固定的。这有助于避免因标记数据集小而陷入局部优化。我们还使用预训练任务的损失函数作为DTA预测任务的正则项,进一步缓解模型的过拟合。因此,具有双重适应机制的多任务学习框架是提高模型对未知药物DTA预测泛化能力的最关键因素。以上对预训练模型的对比分析也表明,仅仅更换预训练模型并不能显著提高DTA预测效果。考虑到计算复杂度,本研究采用基于电流互感器的蛋白质预训练和基于gcn的药物预训练。
结论
深入研究基准数据集Davis,我们注意到之前的DTA预测研究并没有考虑模型在未知药物发现中的泛化能力。为了解决这一挑战,本研究提出了一种新的DTA预测模型,称为generizeddta。我们引入两个蛋白质预训练任务和一个全新的药物预训练任务,学习更丰富的蛋白质和药物的结构信息,加速模型在小尺度标记数据上的收敛。我们还开发了具有双重适应机制的多任务学习框架,以防止预测模型陷入过拟合,提高模型在未知药物发现中的泛化能力。一组新的未知药物数据集的对比实验验证了我们的模型在未知药物发现中的DTA预测的有效性。
数据和材料的可用性
源代码在GitHub存储库中是公开的https://github.com/Frank-39/GeneralizeDTA。
参考文献
吴敏,李小林,郭长奎。使用化学基因组学方法的药物-靶标相互作用的计算预测:一项实证调查。生物通报,2018;20:1337-57。
Manoochehri HE, Nourani M.基于半二部图模型和深度学习的药物-靶标相互作用预测。生物信息学报。2020;21(4):1-16。
穆拉德:新药的开发花费了26亿美元。中国医药科学,2014;13(12):877。
杨建平,刘建平。基于局部局部模型的药物-靶标相互作用的监督预测。生物信息学。2009;25(18):2397 - 403。
张建军,张建军,张建军,等。基于深度潜在因子的药物相互作用预测模型。2019.p . 1254 - 1258。
彭军,李军,尚霞。基于特征表示学习和深度神经网络的药物-靶标相互作用预测方法。生物信息学学报。2020;21(13):1-13。
王春春,赵颖,陈鑫。药物通路关联预测:从实验结果到计算模型。生物通报;2021;22(3):061。
罗勇,赵翔,周军,杨军,张勇,匡伟,彭军,陈磊,曾军。基于异构信息的药物-靶标相互作用预测与计算药物重新定位的网络集成方法。生态学报,2017;8(1):1 - 13。
陈鑫,闫春春,张翔,张翔,戴飞,尹军,张勇。药物-靶点相互作用预测:数据库、web服务器和计算模型。生物通报,2016;17(4):696-712。
He T, Heidemeyer M, Ban F, Cherkasov A, Ester M. Simboost:使用梯度增强机器预测药物靶标结合亲和力的跨读方法。化学信息学报,2017;9(1):1 - 14。
Corsello SM, Bittker JA, Liu Z, Gould J, McCarren P, Hirschman JE, Johnston SE, Vrcic A, Wong B, Khan M, Asiedu J, Narayan R, Mader CC, Subramanian A, Golub TR.药物再利用中心:下一代药物库和信息资源。中华医学杂志,2017;23(4):405-8。
Öztürk H, Özgür A, Ozkirimli E. Deepdta:深度药物靶标结合亲和力预测。生物信息学。2018;34(17):821 - 9。
阮涛,李海,李德明,冯涛。基于图神经网络的药物结合亲和力预测研究。生物信息学。2020;37(8):1140 - 7。
杨忠,钟伟,赵磊,陈春春。基于深度多尺度图神经网络的可解释药物-靶点结合亲和力预测。化学学报。2022;13:816。
曾艳,陈霞,罗艳,李霞,彭丹。基于多注意块的药物-靶点结合深度亲和性预测。生物通报;2021;22(5):117。
Tran HNT, Thomas JJ, Malim NHAH。Deepnc:一个基于图神经网络的药物-靶标相互作用预测框架。PeerJ。2022; 10:13163。
李峰,张震,关健,周森。基于相互作用神经网络的有效药物-靶标相互作用预测。生物信息学btac377 2022;
袁伟,陈刚,陈永成。基于注意力的特征聚合和知识精馏的药物-靶点结合亲和力预测。生物通报。2022;23:1-13。
李建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军,吴建军。生物工程学报,2011;29(11):1046-51。
唐杰,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛,许涛。化学学报,2014;54(3):735-43。
Kanehisa M, Goto S, Hattori M,青木下KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M.从基因组学到化学基因组学:KEGG的新进展。核酸学报,2006;34(增刊1):354-7。
斯特林T,欧文JJ。锌15配体的发现。化学学报,2015;55(11):2324-37。
胡峰,胡勇,张军,王东,尹鹏。基于变压器和图嵌入的结构增强蛋白-药物相互作用预测,2020;10 - 10。
Kirkpatrick J, Pascanu R, Rabinowitz N, Veness J, Desjardins G, Rusu AA, Milan K, Quan J, Ramalho T, Grabska-Barwinska A, Hassabis D, Clopath C, Kumaran D, Hadsell R。科学通报,2017;35(6):521 - 526。
蔡涛,谢玲,陈敏,刘勇,何东,张生,Mura C, Bourne PE,谢玲。基于门户学习的暗化学基因组学空间探索:应用于靶向不可药物基因组和新冠病毒抗感染多药理学研究。2021;arXiv预印本arXiv: 2111.14283
M.机器学习中的Out of distribution generalization。纽约大学博士论文;2020.
Devlin J, Chang M-W, Lee K, Toutanova K. Bert:基于深度双向变换的语言理解预训练。参见:计算语言学协会北美分会2019年年会论文集:人类语言技术(NAACL-HLT 2019)。2019.第4171 - 4186页。
刘建军,刘建军。基于结构信息的蛋白质序列嵌入研究。参见:第七届国际学习表征会议论文集(ICLR 2019) 2019;
李斌,李波,尹松。基于结构信息的深度双向蛋白质序列表征的预训练。IEEE Access. 2021; 9:123912-26。
Elofsson A, Sonnhammer E.序列和结构蛋白结构域家族的比较作为结构基因组学的基础。生物信息学,1999;15(6):480 - 500。
Srivastava N, Hinton G, Krizhevsky A, Sutskever I, Salakhutdinov R. Dropout:一种防止神经网络过度拟合的简单方法。[J] .数学学报,2014;15(1):1929-58。
张建军,张建军。基于图像卷积网络的半监督分类。见:第五届国际学习表征会议论文集(ICLR 2017) 2017。
张建军,张建军。扩散卷积神经网络。第30届神经信息处理系统国际会议论文集(NIPS 2016)。2016.2001 - 9页。
Duvenaud D, Maclaurin D, Aguilera-Iparraguirre J, Gómez-Bombarelli R, Hirzel T, Aspuru-Guzik A, Adams RP。基于图的卷积网络学习分子指纹。第29届神经信息处理系统国际会议论文集(NIPS 2015)。2015.2215 - 23页。
唐杰,曲敏,王敏,张敏,闫杰,梅强。线:大规模信息网络嵌入。参见:第24届万维网国际会议论文集(WWW ' 15)。2015.p . 1067 - 1077
陈凯,陈建军,陈建军。基于分布式表征的词和短语组合。In:神经信息处理系统的进展。2013.P. 3111 - 3119
应荣,何锐,陈凯,Eksombatchai P, Hamilton WL, Leskovec J.基于图卷积神经网络的网络推荐系统。第24届ACM SIGKDD知识发现与数据挖掘国际会议论文集。2018.P. 974 - 983。
李建军,李建军,李建军,等。大型图的归纳表示学习。第31届神经信息处理系统国际会议论文集。2017.P. 1025 - 1035。
白毅,丁宏,乔毅,Marinovic A,顾凯,陈涛,孙勇,王伟。保持图邻近度的无监督归纳全图嵌入。见:第七届学习表征国际会议(ICLR 2019)论文集。2019.
刘建军,刘建军,刘建军,刘建军。深度图信息挖掘。见:第七届学习表征国际会议(ICLR 2019)论文集(海报)。2019.
李建军,李建军。基于模型不可知元学习的深度网络快速适应。第34届机器学习国际会议论文集(PMLR 2017)。2017.第1126 - 1135页。
Finn RD, Bateman A, Clements J, Penelope Coggill RYE, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer ELL, Tate J, Punta M. Pfam:蛋白质家族数据库。核酸学报,2014;42(D1): 222-30。
金正日年代,陈J,程T, Gindulyte,他J,他年代,李问,鞋匠英航Thiessen PA, Yu B, et al。Pubchem 2019更新:改进了对化学数据的访问。核酸学报,2019;47(D1): 1102-9。
张松,姜敏,王松,王鑫,魏铮,李铮。基于自注意图网络的sagg - data:药物靶标亲和力预测。中国生物医学工程杂志,2011;22(6):893。
Cameron AC, Windmeijer FA。一些常见的非线性回归模型拟合优度的r平方度量。[J] .经济研究。1997;37(2):329 - 342。
Rives A, Meier J, Sercu T, Goyal S, Lin Z, Liu J, Guo D, Ott M, Zitnick CL, Ma J,等。生物结构和功能是通过将无监督学习扩展到2.5亿个蛋白质序列而产生的。自然科学进展,2021;118(15):e2016239118。
蔡涛,林辉,Abbu KA,邱勇,Nussinov R,谢磊。msa正则化蛋白序列转换预测全基因组化学-蛋白相互作用:在染色体去孤儿化中的应用。中国生物医学工程学报,2011;31(4):1570-82。
胡伟,刘斌,Gomes J, Zitnik M, Liang P, Pande V, Leskovec J.图神经网络预训练策略。2019;arXiv预印本arXiv: 1905.12265
荣勇,卞勇,徐涛,谢伟,魏勇,黄伟,黄军。大规模分子数据的自监督图变换。神经网络信息处理系统。2020;33:12559-71。
资金
国家重点研发计划项目(批准号:2020YFB2104402)和北京市自然科学基金项目(4222022)资助。
作者信息
作者及单位
贡献
所有作者都对研究的构思和设计做出了贡献。数据收集、编码和分析由石成玉和陈建辉完成。手稿的初稿是由林少甫和石成玉撰写的。所有作者都对手稿以前的版本作了评论。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
林淑娟,史晨,陈杰。基于预训练和多任务学习的广义dta:预测未知药物发现的药物-靶标结合亲和力。BMC生物信息学23, 367(2022)。https://doi.org/10.1186/s12859-022-04905-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04905-6
关键字
- DTA预测
- 训练的任务
- 多任务学习
- 双重适应机制