摘要
背景
反合成预测是由反应产物推断反应物的任务,对设计目标产物的合成路线具有重要意义。产品分子通常用一些描述符表示,如简化分子输入线输入规范(SMILES)或分子指纹,以建立预测模型。然而,现有模型大多只利用一个分子描述子,将分子描述子作为一个整体来考虑,而没有进一步挖掘多尺度特征,不能充分、精细地利用分子和分子描述子特征。
结果
我们提出了一个新的模型来解决上述问题。首先,我们建立了一种新的基于卷积神经网络的特征提取网络,利用不同大小的滤波器从分子描述子中提取多尺度特征;然后,利用双分支特征提取层融合多个分子描述子的多尺度特征,在没有专家知识的情况下进行逆合成预测。与基准USPTO-50k化学数据集上的其他模型的比较结果表明,我们的模型在top-1、top-3、top-5和top-10精度方面超过了最先进的模型7.4%、10.8%、11.7%和12.2%。由于代谢反应中的化合物比化学反应中的化合物更难被特征化,在生物反合成预测领域尚无相关工作,我们利用著名的MetaNetX代谢数据集进一步测试了我们的模型在生物反合成预测任务中的可行性,top-1、top-3、top-5和top-10的准确率分别为45.2%、67.0%、73.6%和82.2%。
结论
在USPTO-50k上的比较结果表明,我们提出的模型优于现有的最先进的模型。在MetaNetX数据集上的评价结果表明,用于反合成预测的模型也可以用于生物反合成预测。
简介
在有机化学和药物开发领域,经常需要找到一系列的反应物来合成目标化合物。它起源于托蒎酮的合成[1],后来被正式定义为逆合成预测[2并已成为有机化学的基本问题之一。逆合成预测问题的巨大搜索空间使得传统方法(物理有机、量子化学等)难以解决这类问题。几十年来,研究人员已经开始使用计算机技术来辅助逆合成预测[3.].由于机器学习在计算机视觉和文本分类等领域取得了重大进展,研究人员开始将机器学习应用于逆合成预测。
为此,人们提出了几种预测模型,如RetroSim [4], NeuralSym [5], Seq2Seq [6],及[7]等,利用机器学习来辅助预测任务。现有的基于机器学习的逆向合成预测模型大致可以分为两类:基于模板的模型和无模板的模型。第一类中的模型利用反应模板来辅助预测过程,而第二类中的模型将反合成预测视为序列到序列的转换问题。
现有的大多数模型主要使用分子描述符作为一个整体进行反合成预测。例如,分子指纹(Extended Connectivity prints, ECFP)将一个化合物分子转化为一串以存在或不存在某种类型分子片段为特征的二元向量,通常用于比较分子指纹的相似性以选择合适的模板,或者直接输入深度神经网络(DNN)作为特征来预测模板。对于其他描述符,例如SMILES,它利用简单的原子符号、键符号和语言规则来描述分子的三维结构,它经常用于seq2seq模型以获得产物到反应物的直接转换。然而,这些现有模型要么没有充分利用分子的多个描述子,导致预测精度低,要么通过建立复杂模型来提高预测精度,导致时间消耗大。如上所述,SMILES侧重于全局信息,而分子指纹侧重于分子结构信息,这两个描述符都是结构化的表示,描述符已经包含了分子的高维特征,如官能团特征。这些描述符中的任何一个缺失都会导致分子特征的缺失。此外,这些描述符的特性也没有得到充分利用,例如多尺度特性。
因此,在本文中,我们利用几个分子描述子,利用CNN提取这些描述子的多尺度特征[8]并利用这些特征进行逆合成预测。具体而言,本文的主要贡献如下:
提出了一种新的基于cnn的多尺度特征提取网络。提议的网络连接了几个不同大小的滤波器。通过这种设计考虑,所提出的网络能够自适应地提取分子描述子的多尺度特征。
我们提出了一个基于上述网络的端到端模型,命名为CNN-TMN,用于逆合成预测。CNN-TMN由两个分支特征提取层组成,用于提取不同分子描述符的可伸缩特征,并将这些特征用于逆合成预测。
我们验证了CNN-TMN对标准USPTO-50k的优势,这些优势来自专利数据库[9],得到Top-1和Top-10的准确率分别为61.1%和87.7%。我们还在MetaNetX数据集上验证了CNN-TMN,作为生物还原合成预测领域的第一次尝试,并为代谢反应数据集提供了基线。
相关工作
在本文中,反应特指两个或两个以上分子碰撞并相互反应生成一个新分子的合成反应。比如反应\(A + B \xrightarrow []{}C\)表示分子A和B合成了一个新的分子C。A和B称为反应物,C称为产物。反合成是将目标产物分解成构件(反应物)的过程。反合成预测的目的是预测能产生目标产物的反应。现有的方法主要分为两类:无模板方法和基于模板的方法。我们首先介绍无模板方法。Template-free方法”部分;然后介绍了基于模板的相关方法。基于模板的方法”一节。
Template-free方法
无模板法通常用已知的反应建立模型来预测目标产物的反应物。然后,所建立的模型可用于直接将目标产物(用分子描述符如序列和图表示)转换为反应物。第一个无模板模型是Seq2Seq,它是基于递归神经网络(RNN) [6].之后,Karpov等人建立了一个基于transformer的方法[10],获得了比Seq2Seq更好的预测性能。Tetko等人在研究Transformer在不同训练场景下的效果的基础上,提出使用数据增强策略对模型进行训练,从而进一步提高模型的性能[11].由于化学分子通常以分子图的形式表示,一些研究者开始利用图的信息来提高预测精度。例如,Seo等。[12]利用序列和图来提高Transformer的性能。Shi等。[13利用分子图信息将逆合成预测作为图到图问题的转换。与之前的无模板模型不同,Hasic等人利用分子指纹直接从目标分子的结构中泛化知识,而不需要额外的信息。由于缺乏模板的指导,预测模型无法揭示反应物与产物之间的反应关系,降低了预测结果的可靠性。
基于模板的方法
在基于模板的方法中,反应根据其原子映射信息被组织成一组模板,以描述反应物与相应产物之间的转化关系。这些模板既可以由人类专家手工制作,也可以通过开发RDKit等工具包从大量已知反应中自动提取。14].因此,基于模板方法的反合成预测的主要目标是为目标产物找到合适的模板,因为从找到的模板中很容易获得相应的反应。基于相似模板产生相似产品的假设,Coley等人提出了一种基于相似度的方法,称为RetroSim,该方法将分子相似性作为一种有效的指标来选择目标产品的模板[4].显然,这种基于相似性的方法非常简单,易于实现。但是,其性能对采用的相似度量很敏感。Segler等人认为逆综合预测问题是一个多分类问题,每个类对应一个模板,并建立了一个基于MLP(多层感知)的模型NeuralSym来预测目标产品的模板[5].该方法无需任何背景知识,可通过一系列隐藏层自动学习分子ECFP指纹与模板之间的非线性关系,易于实现。然而,它有两个缺点。首先,在NeuralSym中缺乏卷积层,导致它无法学习到与模板相关的有利于预测的特征。其次,ECFP指纹的分子表征只能描述分子中结构单元的存在或不存在,无法捕捉到单元-单元相互作用和详细的结构信息。最近,一些研究人员提出建立基于图学习的预测模型。例如,Dai等人提出了一种基于图神经网络(GNNs)的条件图模型,命名为GLN,用于预测目标产品模板的条件概率,隐式地考虑了相应反应的化学可行性和策略[15];Somnath等人将逆合成预测问题作为识别可用于合成目标产物的前体分子的问题,并基于前体分子的图拓扑在化学反应过程中基本不变的思想,提出了基于图的方法GraphRetro [7].与其他方法相比,基于图的方法将反应的领域知识嵌入到图模型中,因此可以获得更好的性能。但它们主要依赖于子图同构的运算,扩展性较差。此外,对领域知识的严重依赖使得初学者很难建立一个健壮的图模型。
该方法
我们的方法属于基于模板的方法。类似于[5],我们也将每个模板视为一个类,并相应地将逆综合预测任务视为一个多分类问题。不同于[5],设计了一个复杂的特征提取网络来学习有利于分类的多尺度特征。在介绍我们方法的细节之前,我们先给出问题的定义。
问题定义
对于一个反应米反应物\ (R_i: S_{我1}+ \ cdots + S_ {i, j} + \ cdots S_{我}\ longrightarrow P_i \),我们用下面的四重组合来表示\ (R_i \):
在哪里\ (\ {S_ {i, j} \} \)反应物的集合是\ (R_i \),米是反应物的总数,\ (P_i \)是\ (R_i \),\ (T_i \)是否从中提取模板\ (R_i \),\(为C_i \)班级标签是\ (T_i \).在我们的工作中,\(S_{i,j} (j=1,\dots,M)\),\ (P_i \)而且\ (T_i \)都表示为SMILE序列。为了方便起见,我们设\(为C_i \)作为正整数。
让\ ((P_i为C_i) \)对表示一个训练样本,问题的目标是训练一个多分类模型\θ(\ \)随着分布(p(C| p, \theta)\)使用一组训练样本,其中p表示输入乘积的概率P属于带class标签的模板C.
CNN-TMN模型的构建
基于cnn的特征提取网络
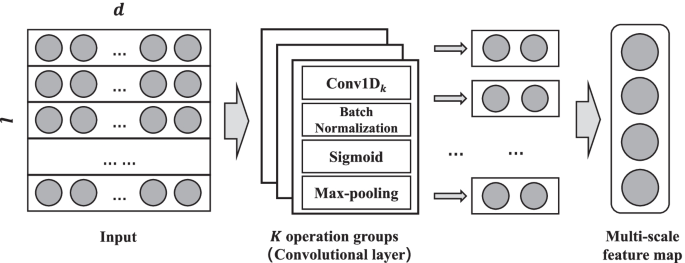
在本节中,我们将介绍所提出的特征提取网络。我们的网络的主要思想是利用多个不同大小的滤波器来提取可伸缩的特征,并将它们整合起来,得到最终的多尺度特征。换句话说,我们的网络也可以看作是的串联K简单的操作组,其中包含卷积、max-pooling、批量归一化和激活操作。所提出的特征提取网络结构如图所示。1.该网络的输入为单热矩阵,输出为多尺度特征图。
我们网络中使用的过滤器都是一维过滤器[8],称为Conv1D。让(X\in R^{l\ * d}\)为网络的输入,为输入序列的单热矩阵,其中l是序列的长度,d为矩阵的维数,等于Conv1D的输入通道。的X将被送入K不同的操作组得到最终的多尺度特征图。在每个操作组中,操作是按顺序进行的。首先,输入之间的点积X和过滤F,得到Conv1D后提取的特征图。然后,对提取的特征图进行批量归一化[16],以减少内部协变量移位,通过调节数据分布来提高泛化能力,提高训练速度。对于规范化的特征映射,我们将应用激活函数。这里,我们使用sigmoid函数[17]来映射[0,1]中的特征。之后,max-pooling [18]操作对特征图进行下采样。最后,将不同操作组输出的特征图拼接在一起,得到最终的多比例尺特征图。
操作组的总数与不同大小的过滤器的数量相同,由一个参数决定一步.设滤波器的最小尺寸为最小值,最大值为马克斯,步长为一步的大小我-th滤波器入我-th运算组计算为:\(F_i = min +(i-1)\times step\),在那里\(F_i < max\).
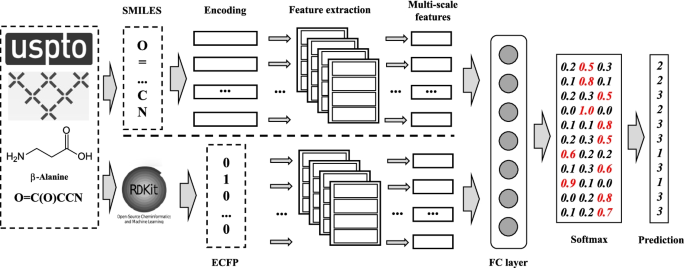
CNN-TMN模型
在本节中,我们将详细介绍CNN-TMN。CNN-TMN概述如图所示。2.CNN-TMN的思想是利用基于所提出的网络的双分支特征提取层,提取分子描述子的多尺度特征,并将其整合得到最终的预测结果。在本文中,我们选择分子描述符作为分子指纹(特别是ECFP)和SMILES。下面介绍用于提取上述ECFP和SMILES多尺度特征的两个不同分支的具体细节。
对于ECFP的特征提取,网络的输入是ECFP,输出是它的多尺度特征图。本文中,ECFP由二进制向量构成:\(X=\{x_1,x_2,{\ldots},x_i,\ldots,x_L\}\),在那里\ (x_i \ \ {0,1 \} \),l为ECFP的最大长度。由于ECFP是一维二元向量,可以直接作为特征提取网络的输入。所有的输入通道K过滤器设置为1。过滤器的大小设置为32到2048一步的32,即过滤器的大小为\(\{32, 64, 96, 2048年\ ldots \} \),共64个过滤器。特征提取后,我们将得到ECFP的多尺度特征。
在提取SMILES多尺度特征前,采用单热编码对SMILES序列进行编码。首先,集合这个词令牌介绍了。的令牌由数据集中不同的字符组成,USPTO-50k和MetaNetX的长度分别为40和48。对于一个长度序列l时,单热矩阵的形状为\ (L长度(令牌)\ \倍).SMILES序列的长度是由数据集的分布决定的,这将在"数据集分割策略”一节。编码后,类似于ECFP的特征提取,将单热矩阵输入特征提取网络,得到分子SMILES的多尺度特征。与ECFP相比,在SMILES中,由于单热矩阵的维数为40,因此输入通道为全部K过滤器设置为40。所有过滤器的大小设置为5到l与一步5,即提取SMILES特征时滤波器的大小为\(\{5,10,15, \ldots,L\}\),其中过滤器的最大大小小于l.
损失函数
对于损失函数,我们使用交叉熵损失函数:
在哪里米而且N分别是标签和样品的总数,\ (y_ {ic} \)表示一个符号函数,如果样本的实标号我和c,然后\ (y_ {ic} = 1 \),\ (p_ {ic} \)表示预测标签的概率c.
实验
我们进行了两种实验来测试CNN-TMN。首先,我们利用基准化学反应数据集USPTO-50k设计了一系列对比实验,综合评估了CNN-TNM的逆合成预测性能。9].由于尚无关于生物还原合成预测的公开报道。其次,我们尝试使用广泛使用的代谢反应数据集MetaNetX [19],以研究其可行性。
数据集和数据预处理
数据集
USPTO-50k数据集在以前的逆合成预测工作中被用作基准数据集[6,10].它目前包含50,016个条目,每个条目对应一个由SMILES序列表示的原子映射反应。MetaNetX代谢反应数据集包含30,986个独特的代谢反应。与USPTO-50k相同,所有反应记为SMILES序列。表中列出了两个数据集的概要1.
数据预处理
由于原始数据集是由反应SMILES组成的,我们需要对每个反应SMILES序列进行处理,以提取每个反应物和产物,并生成相应的模板及其类标签。首先,我们将每个反应SMILES按照符号“>”切片为反应物和生成物。对于多产物反应,我们把它们分成多个单产物反应。然后,我们使用RDKit[14],提取所有反应的反应模板,形成模板集。考虑到多个反应可能具有相同的模板,我们将多余的模板从模板集中去除。对于集合中的每个唯一模板,我们给它赋一个正整数作为类标签。最后,我们将类标签与相应的产品相关联来构造数据集\ (D = {(P_i为C_i)} \)(\ (P_i \)表示乘积,和\(为C_i \)表示它的类标签)。
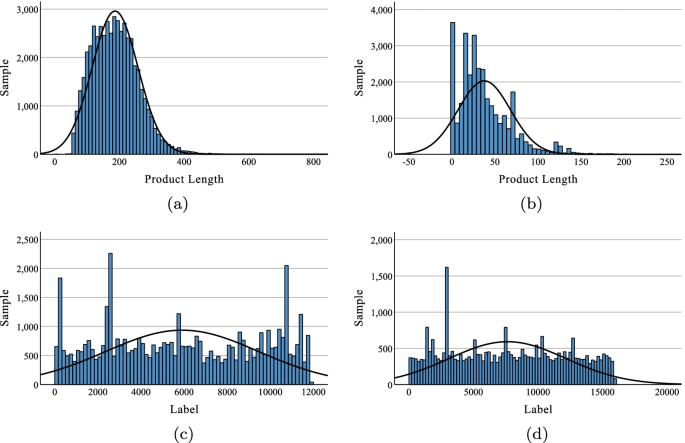
我们统计分析了不同类别的产品长度和产品数量的分布,如图所示。3..从这个图中我们可以看到,USPTO-50k中大部分的产物序列都小于300,而MetaNetX中大部分的产物序列都小于100。如在“CNN-TMN模型Section,我们需要将产品的长度设置为相同的长度l当执行单热编码时。如果产品的长度小于l,我们将插入零,并不可避免地引入噪声。更大的l是,含有噪声的产品序列越多。因此,根据分析结果,我们设lUSPTO-50k和MetaNetX分别为300和100美元。此外,我们发现不同类别的总数非常大(USPTO-50k为11856个,MetaNetX为15939个),而每个类别的产物数量却很少(USPTO-50k平均约为5个,MetaNetX平均为2个),这导致了逆向合成预测问题的困难。
实验装置
基线
为了评估CNN-TMN,我们参考了11个比较基线,包括5个基于模板的模型和6个无模板模型。具体的:
基于模板的:RetroSim[4];NeuralSym[5];GLN[15];循证医学[20.];GraphRetro[7].
Template-Free:Seq2Seq[6];变压器[10];它g2g[13];Tetko的[11];侠盗猎车手[12];基本的[21].
评价指标
我们使用广泛使用的Top-k(\(k= 1,3,5,10 \))精确匹配精度作为我们之前工作的评估指标[4,5,6].该指标比较预测的SMILES序列是否与真实值序列相同。
实现细节
CNN-TMN是在PyTorch中实现的[22].分子的ECFP和反应模板由开源化学信息学软件RDKit提取[14].ECFP和SMILES特性中的滤波器大小分别设置为32到2048,步长为32,5到200,步长为5。所有分子序列的长度都设置为300,进行一次性编码。CNN-TMN训练了20个epoch,批大小为128,学习率为0.001,Adam [23]优化器在单个NVIDIA RTX 2080Ti GPU上。我们训练CNN-TMN大约需要一个小时。
数据集分割策略
为了训练CNN-TMN,我们使用了该领域常用的分裂策略[6],训练集/验证集/测试集随机分为80%/10%/10%,称为“平原”。此外,由于我们将逆综合预测问题视为一个多分类问题,分裂策略会对模型的最终预测性能产生影响。[中使用的分割策略6会导致标签失衡。因此,我们设计了一种新的分割策略“Aug”作为数据增强。新的分割策略确保训练集中的每个标签至少出现一次,并且分割比例与之前的工作相同。
UPSPTO-50k数据集评价结果
不同型号的性能比较
本节将本文提出的模型与USPTO-50k基准数据集上的11个逆合成模型进行比较,实验结果如表所示2.所有比较模型都使用相同的分割策略。脚注1
在采用合理的分割策略后,CNN-TMN的预测精度比基于模板的最先进模型GraphRetro提高了7.6%,比无模板模型Tetko的预测精度提高了7.4%。此外,随着的增加,预测精度的性能也有所提高k,尤其是当\ (k = 10 \).在比较的模型中,NeuralSym与CNN-TMN相似,同样将逆综合预测问题视为多分类问题。CNN-TMN的准确率比它提高了10.6%,正确拆分后提高了22.6%。实验结果证明了CNN-TMN在反合成预测方面的有效性。
如在“数据集分割策略节中,分裂策略对CNN-TMN的性能有很大的影响。当分裂策略不合理时,CNN-TMN的预测性能下降较大,Top-1精度下降12%。在[6]时,对数据集进行随机分裂,而在本文中,我们采用了一种新的分割策略,该策略在“数据集分割策略”一节。为了探究不同的分裂策略对预测精度的影响,我们选择了与我们相似的NeuralSym进行不同分裂策略下的性能比较,结果如表所示3..
表中的结果3.结果表明,分割策略对CNN-TMN和NeuralSym的性能都有非常显著的影响。主要原因是随机分裂会导致类的不平衡,如果一个特定的标签没有包含在训练集中,对该类的预测精度会大大降低。经过合理的分割,两者的准确率都有了明显的提高。采用合理的分裂策略后,NeuralSym的预测准确率提高了6.9%,而CNN-TMN的预测准确率提高了12%。上述实验表明,我们的分裂策略可以显著提高这类模型的预测精度。
消融实验
消融研究旨在验证使用多个描述符和所提出的特征提取网络的有效性。首先,我们探讨了使用不同分子描述符的有效性,结果总结在表中4.检查\(\对号\)表中表示CNN-TMN使用特定的分子描述符。例如,第一行表示CNN-TMN仅使用ECFP进行预测。
通过只使用ECFP描述符,如第一行所示,它实现了47.2%的Top-1精度和80.0%的Top-10精度。通过仅使用分子的SMILES描述符,我们实现了53.8%的Top-1准确度和84.7的Top-10准确度。通过进一步融合两个描述符,我们获得了61.1%的最终Top-1精度,如最后一行所示。上述实验结果表明,使用多个分子描述符可以显著提高预测的准确性。
然后,我们验证了所提出的特征提取网络在提取分子描述子多尺度特征方面的有效性。结果如表所示5.正如我们在表格中看到的5结果表明,本文提出的网络能够有效提取分子描述子的多尺度特征,预测精度较基线平均提高26.65%。此外,对于两种不同的分子描述符,对准确度的影响是不同的,总体而言,使用SMILES的准确度比使用ECFP的准确度高1.6%。主要原因是ECFP描述子在表示上是一维向量,与SMILES相比,ECFP的特征更稀疏,这导致多尺度特征提取网络在提取两个分子描述子时表现不同。
为了进一步验证所提出的特征提取网络的有效性,我们将网络替换为常用的CNN架构TextCNN [8],在文本分类领域常用,网络的其余部分保持不变。实验结果如表所示6.与TextCNN相比,CNN-TMNl有明显的改进,主要是因为我们使用了不同大小的滤波器来提取不同尺度的特征,而在TextCNN中,最大的滤波器只有5个。通过使用大卷积核,我们能够捕捉序列的不同长度依赖关系,这可以更好地提高模型的预测精度。
的影响一步关于性能
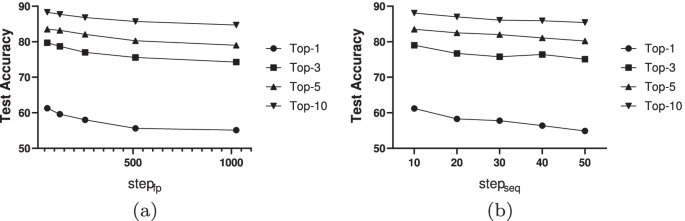
在CNN-TMN中,我们有一个参数一步用于确定SMILES和ECFP特征提取中滤波器的大小。本节将详细讨论此参数的影响。用于提取ECFP的过滤器\ (step_ {fp} \)和微笑\ (step_ {seq} \),则设置为\ \{64128256512、1024 \}\)而且\(\{10年,20年,30、40、50 \}\),分别。结果如图所示。4.
如图所示。4,均为两位数,精度较高的可采用较小的一步,反之亦然。一个更大的一步将导致粗粒度的特性,而较小的一步可以使提取的特征更加细粒度。即使指纹是稀疏的,也可以通过使用不同大小的滤波器提取特征。但是,缺点比较小一步滤波器数量的增加会使网络的体系结构更加复杂,从而增加计算成本。这一结果支持了假设,即通过使用不同大小的滤波器,我们可以很好地利用CNN提取序列的多尺度特征。
模型复杂性分析
如上所述,有一个参数一步控制特征提取网络中操作组的数量。以研究不同的效果一步在模型尺寸上,我们增加了不同条件下模型参数数量的统计实验一步.由于提取不同分子描述子的步骤不同,在不同条件下模型参数的数量也不同一步分别列出,统计结果见表7.
从表中的统计结果可以看出,CNN-TMN中参数个数与参数负相关一步,且均随的增加而减小一步.同时,结合图中的结果。4,可以发现的大小一步与CNN-TMN的准确率呈负相关。主要原因是我们用的时候比较小一步, CNN-TMN可以利用更多的参数来表征分子描述符,从而提高预测精度。
生物还原合成预测的可行性研究
由于化学反应简单,反合成预测主要集中在化学反应预测上,但在生物系统中,代谢反应比化学反应更复杂,据我们所知,目前还没有关于代谢反应数据集(如MetaNetX)的发表工作尝试。为了验证CNN-TMN在生物还原合成预测中的性能,我们将CNN-TMN应用于MetaNetX代谢反应数据集。生物还原合成预测比较模型为MLP,只有三层:输入层、隐藏层和输出层。输入层是ECFP,而输出层是模板的标签。结果如表所示8.
表中的结果表明,与化学反合成预测相比,生物反合成预测的性能下降了15.9%。如图所示。3.MetaNetX数据集的产品长度分布通常比USPTO-50k短,而且在标签数量方面,MetaNetX数据集的产品特征更少,在MetaNetX中更难预测。然而,CNN-TMN仍然比MLP高出1.6%。这也证明了CNN-TMN的有效性。同时,上述结果为生物还原合成预测提供了基础。
结论与讨论
结论
在本文中,我们提出了一个新的端到端模型,称为CNN-TMN,用于逆合成预测。CNN-TMN利用新设计的基于cnn的特征提取网络来提取分子描述符的多尺度特征。具体来说,在我们的网络中,我们只关注序列的低级特征,这与传统的CNN提取深度特征不同。通过使用不同大小的过滤器,我们可以提取可伸缩的特征。在CNN-TMN中,我们使用双分支特征提取层提取一个分子的多个分子描述子的多尺度特征,然后将它们连接在一起,得到最终的融合特征,用于逆合成预测。实验结果表明,本文提出的特征提取网络能够有效提取分子描述子的多尺度特征,且CNN-TMN在USPTO-50k上的预测精度明显高于其他现有模型,证明了CNN-TMN的有效性。此外,我们将CNN-TMN应用于生物还原合成预测,并在MetaNetX数据集上提供了基线。
讨论
本文提出的CNN-TMN充分考虑了多个分子描述符,可以更充分地利用分子特征。现有模型只考虑分子序列描述符,如SMILES [6,10,12],分子图[7,13]或分子指纹[5].“实验”消融实验部分表明,利用几种分子描述符可以显著提高反合成预测的准确性。根据分子描述子的特点,设计了适合的特征提取网络。在特征提取网络中,我们使用了多个不同大小的滤波器,而不是固定大小的滤波器,这种方法可以更好地捕获序列的长依赖关系。表5而且6验证了我们的特征提取网络的优越性。我们的CNN-TMN是一个基于模板的模型,即对于特定的产品,预测一个可以应用于该产品的模板。如果预测的模板与实际情况不匹配,则预测将被认为是失败的,但有可能将预测的模板应用于产物以获得潜在的反应物。与无模板模型预测的错误反应物相比,基于模板模型预测的错误反应物在化学原理方面更具解释性。
然而,由于我们将逆合成预测问题视为一个多分类问题,且类数过多,因此它比其他模型对训练集的影响更敏感。实验结果如表所示5,如果训练集中的标签不平衡,类似模型的预测精度会低得多,平均Top-k精度下降11.4%。为了更好地预测,一种使类别更加平衡的分割策略,例如“数据集部分,需要考虑。
数据和材料的可用性
在当前研究中使用的数据集和代码可在代码和数据集中获得。
笔记
-
ems是预印本,未正式出版。
参考文献
Robinson R. lxiii - tropinone的合成物。化学学报,1917;11:762 - 8。
科里EJ。化学合成的逻辑:复杂碳源分子的多步合成(诺贝尔演讲)。化学与工程学报。2001;30(5):455-65。
李志强,李志强。复杂有机合成的计算机辅助设计。科学。1969;166(3902):178 - 92。
Coley CW, Rogers L, Green WH, Jensen KF。基于分子相似性的计算机辅助反合成。中国生物医学工程学报,2017;3(12):1237-45。
Segler MH, Waller MP。用于反合成和反应预测的神经符号机器学习。化学通报,2017;23(25):5966-71。
刘波,Ramsundar B, Kawthekar P, Shi J, Gomes J, Luu Nguyen Q, Ho S, Sloane J, Wender P, Pande V.基于神经序列到序列模型的反合成反应预测。中国生物医学工程学报,2017;3(10):1103-13。
Somnath VR, Bunne C, Coley C, Krause A, Barzilay R.逆向合成预测的学习图模型。神经网络信息处理系统。2021;34:9405-15。
用于句子分类的卷积神经网络。见:2014年自然语言处理(EMNLP)经验方法会议论文集。多哈:计算语言学协会;2014.1746 - 51页。https://doi.org/10.3115/v1/D14-1181.
从文献中提取化学结构和反应。博士论文,剑桥大学,2012。
王志强,王志强,王志强等。一种新型的反合成变压器模型。在:人工神经网络国际会议。施普林格;2019.817 - 30页。
Tetko IV, Karpov P, Van Deursen R, Godin G.用于直接和单步反合成的最先进的增强nlp变压器模型。网络学报。2020;11(1):1 - 11。
徐世伟,宋云云,杨建勇,裴s,李华,申杰,黄世杰,杨娥。Gta:反合成的截断注意图。Proc aai Conf Artif Intell. 2021;35(1): 531-9。
史超,徐敏,郭华,张敏,唐娟。一种基于图到图的逆合成预测框架。在:国际机器学习会议。PMLR;2020.8818 - 27页。
Landrum G,等。Rdkit:开源化学信息学。2006.
戴华,李超,戴波,宋林。基于条件图逻辑网络的逆综合预测。2020.arXiv预印本arXiv: 2001.01408.
Ioffe S, Szegedy C.批归一化:通过减少内部协变量移位加速深度网络训练。在:国际机器学习会议。PMLR;2015.448 - 56页。
韩杰,Moraga C. sigmoid函数参数对反向传播学习速度的影响。见:人工神经网络国际研讨会。施普林格;1995.p . 195 - 201。
Ciresan DC, Meier U, Masci J, Gambardella LM, Schmidhuber J.用于图像分类的灵活,高性能卷积神经网络。见:第22届人工智能国际联合会议,2011。
Moretti S, Martin O, Van Du Tran T, Bridge A, Morgat A, Pagni M. Metanetx/mnxref-调和代谢产物和生化反应,将基因组尺度代谢网络整合在一起。核酸研究,2016;44(D1): 523-6。
孙锐,戴华,李林,Kearnes S,戴波。基于能量的逆合成观点。2020.arXiv预印本arXiv: 2007.13437.
王志强,王志强,王志强,等。基于分子子结构指纹识别的单步反合成预测。《化学学报》,2018;29(2):641-52。
Paszke A, Gross S, Massa F, Lerer A, Bradbury J, Chanan G, Killeen T, Lin Z, Gimelshein N, Antiga L,等。Pytorch:命令式的高性能深度学习库。计算机应用。2019;32:8026-37。
金马平,巴杰。亚当:一种随机优化方法。2014.arXiv预印本arXiv: 1412.6980.
确认
我们要感谢编辑和审稿人对这项工作的宝贵意见,这有助于提高本文的质量。
资金
国家重点研发计划项目(No. 2019YFA0904303)资助。
作者信息
作者及隶属关系
贡献
所有作者都参与了研究的设计。框架结构设计:JL和FY。数据提取:XLZ和QZ。代码编写:FY和ZHY。起草稿:FY和JL。对稿件的关键修改,修改,最终稿件的批准:所有作者。监督:杰。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
杨飞,刘杰,张琼。et al。基于cnn双分支多尺度特征提取网络的逆综合预测。BMC生物信息学23, 362(2022)。https://doi.org/10.1186/s12859-022-04904-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04904-7
关键字
- Retrosynthesis预测
- 卷积神经网络
- 机器学习
- 多尺度特性