摘要
背景
尽管细胞低温电子断层扫描技术(CET)最近取得了进展,但由于缺乏注释数据和高结构复杂性,开发用于亚分子分辨率大分子鉴定的自动化工具仍然具有挑战性。迄今为止,为这个问题构建的深度学习方法的范围仅限于传统的卷积神经网络(cnn)。识别不同类型和大小的大分子是一项冗长而耗时的任务。在本文中,我们采用基于胶囊的架构来自动完成大分子识别任务,我们称之为3D-UCaps。该体系结构由三个部分组成:特征提取器、胶囊编码器和CNN解码器。特征提取器将输入子层析图的体素强度转换为局部特征的活动。编码器是一个三维胶囊网络(CapsNet),它采用局部特征来生成输入的低维表示。然后,三维CNN解码器通过上采样从给定的表示重构子层析图。
结果
我们对合成数据和实验数据进行了二元和多类定位和识别任务。我们观察到3D-UNet和3D-UCaps有一个\ (f - \)测试数据得分大多在60%和70%以上。在这两种网络体系结构中,我们都观察到网络的性能下降了至少40%\ (f \)-在识别非常小的颗粒(PDB条目3GL1)与大颗粒(PDB条目4D8Q)时得分。在实验数据的多类识别任务中,3D-UCaps具有一定的识别率\ (f \)-测试数据得分为91%,而3D-UNet为64%。更好的\ (f \)与3D-UNet相比,3D-UCaps的-score得到了更高的精度评分。我们推测这是由于编码器中使用的胶囊网络。为了进一步研究基于capsnet的编码器架构的影响,我们进行了消融研究,并发现\ (f \)-score随着网络深度的增加而提高,这与之前报道的3D-UNet的结果相反。为了呈现一个可重复的工作,源代码、训练模型、数据以及可视化结果都是公开的。
结论
定量和定性结果表明,3D-UCaps成功地完成了包括大分子识别和定位在内的各种下游任务,并且至少可以与CNN架构竞争这项任务。考虑到胶囊层提取了分子的存在概率和方向,这种架构有可能导致比3D-UNet更好地解释数据的表示。
介绍
了解单细胞内的生物过程需要对天然结构和大分子复合物的空间分布有详细的了解。尽管细胞低温电子断层扫描(CET)的最新进展使研究人员能够以亚分子分辨率对这些复杂结构进行三维可视化,并接近其原始状态,但缺乏注释数据阻碍了他们开发这些结构的自动分割工具。在拥挤的细胞环境中,使用像深度学习(DL)模型这样的全自动方法来定位和识别大分子结构还没有得到充分的探索。数字1说明了识别这种复杂结构的问题。基本上,大部分建议的架构[1,2,3.]局限于卷积神经网络(CNN),它需要大规模的训练数据集,并通过耗时的用户干预进行标注。
目前大分子(粒子)定位和鉴定的标准方法是模板匹配(TM) [4]。在TM中,使用所需分子的模板密度图扫描整个层析图,并为体积中的每个体素计算相互关联分数。相关度最高的分数被认为是可能的粒子位置,在这些位置上可以提取子体积,用于下游任务,如分类和子断层图平均。模板匹配的缺点是计算复杂度高,难以识别具有相似结构的粒子。
计算机视觉的许多研究领域,如医学成像,已经被深度学习方法彻底改变了。最近,不同的CNN架构提出了几种全自动二维方法用于大分子识别。第一个提出的方法是DeepPicker,它是一个有7层的二维CNN [5]。该方法包括四个预处理步骤,即对二维显微照片进行评分、清洗、过滤和分类。然后,使用显微照片训练CNN模型,网络的结果再次经过上述四步预处理阶段。后来,DeepCryoPicker [3.]被提出有13层,并使用从2D显微照片中提取的小子区域进行训练。在后期处理中,将子区域拼接在一起以生成完整的显微照片。其他监督算法包括DeepEM [6],黄玉[7]及crYOLO [8]。前者使用正无标记学习技术训练二维CNN,用于检测非球状不对称粒子。所有这些先前的模型都是为单颗粒分析而开发的,其中纯化的蛋白质用透射电子显微镜成像,产生大量的二维图像,每个图像包含数千个颗粒。然而,在CET中,可能的目标颗粒需要在真实细胞拥挤的三维环境中以及从有限数量的断层扫描中识别出来。
在最近的一项工作中,Moebel等人提出了DeepFinder模型[2],采用3D-UNet架构[9]用于在CET图像中进行大分子识别。该模型是使用以单个分子为中心的层析图的三维子体积来训练的。为了验证他们的模型,作者使用了合成的SHREC ' 19数据集[10], 10张断层图加上3个实验数据集,分别为50张、4张和5张断层图。他们报告说,该模型在多类识别场景中的表现优于二元分类。与以往的研究工作类似[1,3.,他们研究\ (f {1} \)根据训练集中粒子的数量对进化进行评分,并报告当断层图中注释粒子的数量过低时性能会下降。此外,还指出,使用有限的数据量进行大分子的二元识别是具有挑战性的。其他3D大分子识别算法包括Che等人开发的算法。[1]基于深层小接受野[11]和CB3D [12)模型。众所周知,CNN架构需要大量数据。尽管数据采集技术取得了突破,但由于标注过程耗时,制备一个以合理数量的粒子作为样本的CET数据集来调整网络权值仍然是该领域的瓶颈。
受到先前有希望的结果的激励[1,2],我们采用了一种基于CapsNet的新的3D dl架构[13],这是已知的数据效率[14,15]。CapsNet的3D版本由Zhao等人提出。[16]。后来,Nguyen等人[17用5层3D CapsNet代替3D- unet的编码器部分,进行MRI图像分割。这个模型被称为3D- ucaps,它有三个主要组成部分:一个特征提取器,一个具有五层3D胶囊的编码器,以及一个由五层3D卷积组成的解码器。在这里,我们修改3D-UCaps结构来进行大分子识别和分割。我们的实验和可视化结果表明,该模型通过学习编码器提取的粒子的纹理和几何形状等一般特征,成功地完成了二元和多类大分子的识别。
本研究的主要贡献有两个方面:首先,引入了一种更深层的神经网络架构,成功地对测试数据进行了多类和二元分子识别。其次,在数据量有限的情况下,使用CapsNet架构改进多类和二元分类。在这里,如果数据包含少于10,000个带注释的粒子,则认为数据是有限的。
本文的其余部分组织如下:在第一部分中,我们简要说明了用于大分子鉴定和定位的数据集和模型。在下一节中,我们将通过各种实验来评估该模型。接下来是数字和视觉结果。最后,我们讨论了研究的局限性和未来的工作。
材料和方法
数据集
我们使用了两组数据来进行实验。第一个是实验数据集,由四张断层图组成,每张断层图分别标注了大肠杆菌的70S核糖体和嗜酸乳杆菌的20S蛋白酶体两个大分子。数字2说明在我们的研究中使用的粒子的密度图。
层析图的原始体素尺寸为\(3712 \times 3712 \times 1392\)体素大小为1.1 Å。对于这里报道的所有实验,层析图被降采样到\(410 \times 410 \times 154\)体素产生各向同性体素大小为10 Å。需要下采样来增加感受野,这样神经网络甚至可以完整地看到大的大分子。除了下采样,低通高斯滤波器[18应用于断层扫描。的角度范围内获得的倾斜序列重建层析图\(\pm 60^{\circ}\)与一个\(3 ^{\保监会}\)增量。通过模板匹配,再进行多ref分类,得到专家标注[19]停止,停止[20.]。表格1显示实验数据集中所有层析图上粒子类的分布。有关详细信息,请参阅附加文件1。实验数据将应要求提供用于研究目的,并将在不久的将来在EMPIAR数据库中公开提供。
第二个数据集由乌得勒支大学为SHREC ' 19挑战赛发布的10张模拟层析图组成[10]。该数据集包含12种不同大小和形状的不同分子。每个班级每张断层扫描图平均有208个分子。为了对精度和\ (f {1} \)在两个数据集之间的分数,我们选择了三种粒子,覆盖了大、中、小的大分子。模拟层析图的大小为\(512 \乘以512\乘以512\)体素。层析图没有进行降采样或去噪。表格2为SHREC ' 19数据集中每个层析图的粒子数。
方法
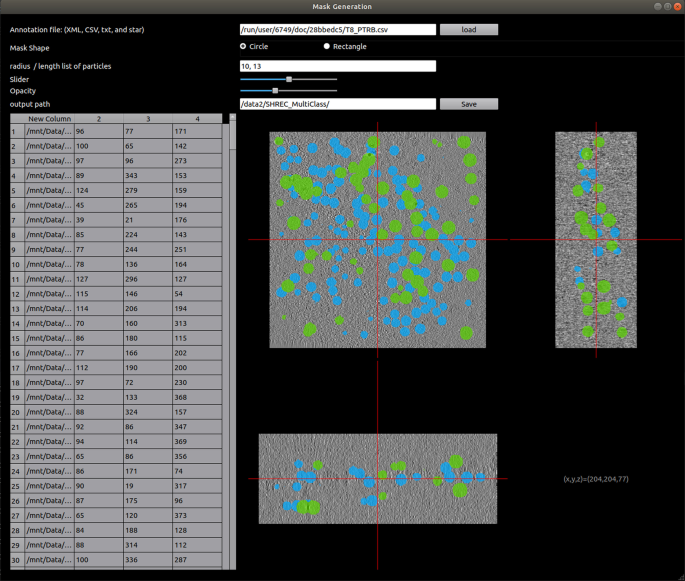
为了训练网络,需要成对的层析图和地面真值标签。在模拟数据中,粒子的精确位置是已知的,而在实验数据中,这样的坐标是由专家提供的。类似于Moebel等人。[2],我们使用球形掩模来生成地面真值标签,即3D球体将每个大分子实例包围在断层图中各自的位置,半径与目标大分子的大小相对应。球形掩模的主要缺点是它们会给ground truth添加一定量的标签噪声。然而,我们选择这种掩码生成策略是为了显示我们的模型对训练数据中的标签噪声的鲁棒性。使用我们开发的GUI生成掩码的示例如图所示。3.。
第一组实验旨在评估所提出的二类和多类粒子的定位和识别方法。为此,我们为不同数量的类生成训练数据集。对于实验数据,我们生成了两个二值子集和一个多类子集,分别表示为PT-BG、RB-BG和PT-RB-BG,其中PT、RB和BG分别代表蛋白酶体、核糖体和背景。如前所述,对于SHREC ' 19数据集,我们只使用了12类高分子中的3类。我们选择了4D8Q、1BXN和3GL1作为大、中、微小颗粒类的代表。这里,我们将这些二值和多类子集分别称为4D8Q-BG、1BXN-BG、3GL1-BG和4D8Q-1BXN-3GL1-BG。通过对训练数据进行6倍交叉验证,我们保留了总粒子的17%进行验证;不使用数据增强。
我们选择了DeepFinder脚注1作为数值比较的模型,这可以被认为是最先进的。DeepFinder基于UNet结构,采用带跳过连接的编码器-解码器范例。编码器是下采样网络组件,使用最大池化和卷积层提取样本的一般特征,而解码器是上采样组件,使用提取的特征,跳过连接和上卷积输出高分辨率标签图。它由两个上下采样层组成,在两个组件中都有大小相同的滤波器\(3 \乘以3\乘以3\)。网络参数个数为\ (903 \ \ sim)K.我们使用上述所有的二元和多类数据集来训练这个网络,用于大分子识别和定位。
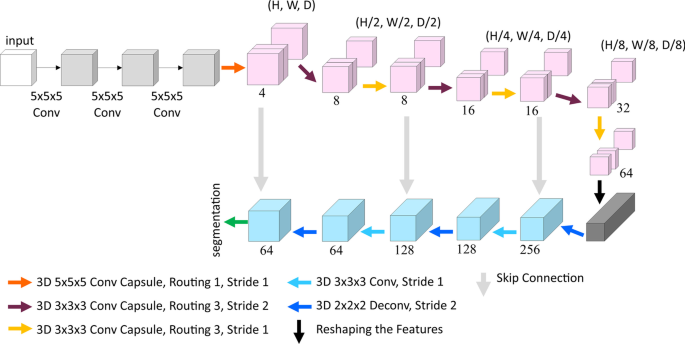
接下来,我们利用3D-UCaps定义了一个网络架构,用于同样的任务,即大分子识别和定位。数字4显示网络的框图,其中包含有关内核大小和层数的更多细节。
类似于DeepFinder, 3D-UCaps脚注2基于具有5层编码器-解码器组件的UNet结构。然而,3D- ucaps的不同之处在于它在编码器部分使用3D CapsNet而不是经典的CNN架构。此外,网络初始化与一个特征提取组件,转换体素强度为局部特征的活动,以便它可以被编码器的主胶囊使用。编码器中的3D胶囊然后提取上下文信息。解码器受益于跳过连接和批处理规范化层。我们将特征提取器中的核的大小设置为\(5 \乘以5\乘以5\)输出是一个大小相等的特征映射h \乘以w \乘以d \乘以64\)。这些输出被重新塑造成网格(h乘以w乘以d),每个代表一个64D特征向量。解码器的卷积层具有一定大小的滤波器\(3 \乘以3\乘以3\)。可训练参数的总数为\ \ (sim 3 m \)。需要注意的是,同样数量参数的等效CNN架构需要更大的数据量才能完全优化。所有训练实验均在Nvidia Quadro RTX 6000 GPU上进行。为了展示一个可重复的工作,我们对这些方法和结果的具体实现是公开的脚注3.,脚注4。
采用这种架构的主要动机是,与训练期间使用的图像相比,当图像中相同物体的方向略有不同时,CNN的性能会下降。换句话说,为了从任何视点检测大分子,我们必须在训练数据中拥有所有这些视点,因为几何关系的先验知识无法在网络体系结构中建模。也就是说,基于cnn的模型无法捕捉实体的空间关系。这主要是由于层层叠叠,从底层特征构建抽象上下文。然而,基于胶囊的架构通过在胶囊内封装多层并捕获实体的内部结构,对几何转换具有鲁棒性。如前所述,使用CET图像进行粒子识别的一个挑战是粒子可以出现在许多不同的方向上。由于CapsNet是视点不变的,它可以处理需要较少的相同粒子的不同视点的识别问题。
cnn使用提取特征的副本和最大池来输出标量特征检测器,这些特征检测器被CapsNet中的矢量输出胶囊和协议路由所取代。在这里,学习到的特征仍然通过覆盖图像更大区域的更高级别胶囊在整个空间中复制。然而,与最大池化不同的是,关于粒子的确切位置的信息不会被丢弃,而是进行位置编码。这是通过路由算法实现的,该算法通过选择哪些胶囊将在下一层被激活来防止指数深度增长。每一层的胶囊应用一组学习过的过滤器来检测粒子的特征和方向。如果胶囊对粒子的存在性非常一致,那么输出将传播到下一层最相关的胶囊。激活胶囊的路线代表了粒子的低水平到高水平的层次结构。这允许架构具有更深或更高数量的下/上采样层。cnn学习到的特征只包含存在信息和纹理特征,这些特征通过较低层次聚合到较高层次。这导致了cnn不能很好地泛化到输入图像的不可见的几何变化的问题。 In CapsNets, fine-grained information is retained through learned features aggregated in higher-level layers. The outputs of each capsule is a high-dimensional vector encoding presence and orientation of the particles. In fact, CapsNets use the非线性压缩函数表示粒子存在的概率。这个函数可以写成:
这里是胶囊的总输入和矢量输出j表示为\ (\ varvec{年代}_j \)和\ (\ varvec {v} _j \),分别。让\ (\ varvec {W} _ {ij} \)是权矩阵和\ (u_i \)是下一层所预测的向量。然后,\ (\ varvec{年代}_j \)为加权和,计算公式为:
在哪里\ (c_ {ij} \)是耦合系数。
同样重要的是,cnn在有限数据量的训练下很难泛化到新的观点[13],而capnet将强度转换为实例化参数的向量,然后应用变换矩阵来克服这个问题。我们稍微修改了结构,将特征提取器中的扩张卷积替换为规则卷积,因为接受野的扩展会使结果恶化。将交叉熵(CE)损失替换为联合Dice和CE损失,以获得更好的分类性能。此外,我们没有使用屏蔽均方误差来处理重建损失,而是使用加权交叉熵,据报道,加权交叉熵在样本不平衡的情况下更成功[2]。
实验结果及分析
实验
两个网络的批处理大小和优化器等超参数设置为相似的值。这里提到的其他参数,如epoch数,是根据网络收敛性来定义的。使用ADAM优化器算法对模型进行计算训练,学习率为0.0001。批量大小设置为24,补丁大小设置为\(64 \times 64\ times 64\)体素。由于大层析图中大分子的尺寸较小,粒子类与背景类之间存在高度的类不平衡。只有1左右\ \ % \ ()其中的体素代表大分子,其余的是背景体素[2]。为了解决这个问题,我们使用大分子位置提取的训练补丁来训练网络。带有提前停止标准的网络训练了300个epoch,并且具有提前停止标准的未训练的3D-UCaps总是在不到300个epoch的时间内收敛。
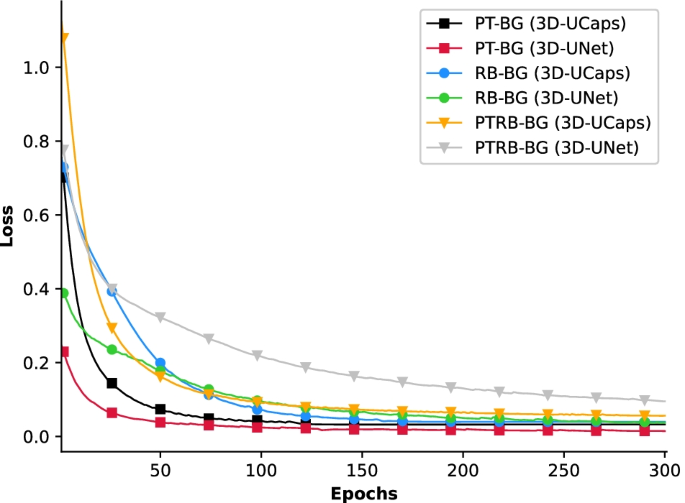
数字5描述了3D-UNet和3D-UCaps在真实数据上的二元和多类训练的学习曲线。两种模型显示出相似的衰减率,然而,3D-UNet在PT-BG数据集上收敛到比3D-UCaps更低的平均损耗(0.03比0.06)。对于PT-RB-BG数据集,3D-UCaps的平均损失显著降低(0.12比0.20)。当使用RB-BG数据集进行训练时,平均损失是相当的。本例对3D-UCaps和3D-UNet进行优化,损失值分别为0.10和0.09。学习曲线表示每个模型的分割质量和泛化能力\ (f \)下一节中表示的分数量化了本地化性能。
为了评估预测的质量\ (f \)分数是常用的。它是基于另外两个衡量标准,精准度和召回率。精度是正确预测的正观测值与总预测的正观测值的比值。它有助于理解模型正确预测为正的频率。当预测假阳性的成本很高时,这个指标很重要。另一方面,当预测假阴性的成本很高时,回忆是一个重要因素。事实上,召回率表示正确预测的正观察值与正类中所有观察值的比率。为了对演出有一个正确的诠释,\ (f \)分数是作为一个同时考虑精确率和召回率的度量标准引入的。这个分数是精确率和召回率的加权平均值,并考虑了假阳性和假阴性。因此,当我们有一个不平衡的类分布时,它是可靠的。这些指标定义如下:
3 d-unet
DeepFinder (3D-UNet)被训练用于多类和二元大分子识别。Moebel等人使用了四种不同的数据集[2]。第四个数据集与我们的真实数据集具有相同数量的层析图和最少数量的注释和类。因此,我们采用了与Moebel等人几乎相同的训练方案。表格3.比较\ (f {1} \)使用真实和模拟数据训练网络在多类和二元大分子识别下的得分、精度和召回。给出了验证和测试数据的结果。验证结果和测试结果之间的较大差距表明存在过拟合问题。
培训3 d-ucaps
同样,3D-UCaps在多类和二元分类场景下对真实和模拟数据进行训练。总的来说,多类场景提供了更好的性能,这与文献中已有的结果一致[2,3.]。类似于3D-UNet,我们提出\ (f {1} \)表中训练3D-UCaps的得分、准确率和召回率4。训练是用实验部分中提到的类似的超参数完成的。
比较与分析
从表3.和4,可以看出,无论用于训练的数据类型(真实或模拟)如何,这两个模型都显示出稳健的性能。然而,关于两者之间的区别\ (f \)通过验证集和测试集的分数,我们可以观察到实验数据比人工数据表现出更大的性能下降。这可能是由于标注过程中产生的标签不正确以及实验数据集的信噪比较低所致。其中,两组的差异分别为0.19和0.13\ (f \)分别为3D-UNet和3D-UCaps的PT-BG数据集验证和测试集得分。
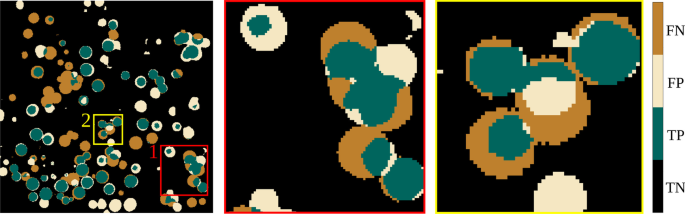
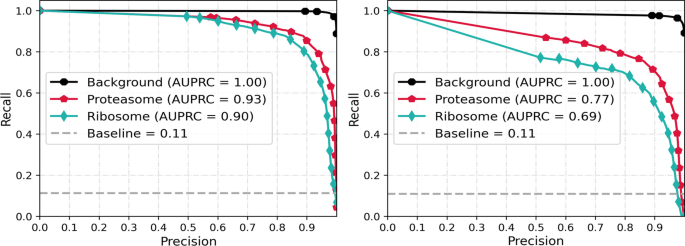
密切\ (f \)验证和测试数据的分数证明了一个成功的学习过程,它依赖于几何形状和纹理等一般特征。一般来说,我们观察到\ (f \)3D-UNet和3D-UCaps的得分接近。然而,我们可以看到,略高\ (f \)3D-UCaps的评分往往与较高的精度评分相吻合(表中粗体)4).当我们观察个体断层图上的像素预测时,这一点可以更明显地看到。为了支持这一观察结果,我们将代表性预测掩模与基础真值掩模重叠,以使用伪颜色评估真阳性(TP)、假阳性(FP)、假阴性(FN)和真阴性(TN)。我们注意到3D-UCaps比3D-UNet预测了更多的TP像素(见图2)。6和7相比之下,3D-UNet预测了更多的FN像素,这导致了整体的降低\ (f \)分数(见图)6和7通过对预测掩模的进一步研究,我们还观察到,在3D-UNet中,通常掩模区域被过度预测(见图1)。7(FP像素的环),而3D-UCaps预测了地面真值掩模的子区域(见图2)。6(FN像素环)。我们推测,与3D-UNet相比,3D-UCaps的精度更高是由于编码器中使用的胶囊网络。
通过说明图中精确度召回曲线(AUPRC)下的面积,可以证明上述解释是正确的。8。AUPRC是不平衡数据的另一个性能指标。在这里,AUPRC曲线的基线被定义为正像素比总像素数的分数。
可视化
数字9给出了实验集中多类场景的测试层析图结果的可视化。在代码实现的材料中提供了其他场景和数据集的验证数据的可视化。对重叠25体素的斑块进行预测,然后缝合生成整个层析图。重要的是要注意,拼接过程自然会在边界周围产生尖锐的边缘和其他伪影,这是通过对这些区域进行平均来解决的。
架构评估
在本节中,我们通过对3D-UCaps架构进行细微的更改来分析模型性能。我们研究了减少层数对性能的影响。Moebel等。[2报告指出,使用两个以上的降采样阶段并不能改善结果。最初,3D-UCaps有5层。为了在3D-UCaps结构中研究这种效果,我们从编码器和解码器中丢弃了3层。然后,我们使用基准参数集在实验数据上训练网络。数字10利用实验数据验证集对5层和2层3D-UCaps的结果进行了比较。实验结果表明,5层网络的性能始终优于2层网络。
消融实验
最初,3D-UCaps在特征提取器组件中使用扩展卷积。扩展卷积通过定义核中值之间的间隔来修改卷积核。使用扩展卷积背后的主要原因是在没有额外计算成本的情况下有更大的接受场。我们进行了两个实验,通过用正则卷积代替扩展卷积来研究扩展卷积的效果。膨胀核的扩大行为似乎是破坏性的,因为减小了\ (f \)多班实验数据得分从91%到86%。同时,增大了编码器中卷积核的核大小\(3 \乘以3\乘以3\)来\(5 \乘以5\乘以5\)性能从91%下降到83%。在小颗粒二元分类的情况下,结果的恶化更为明显。
案例研究
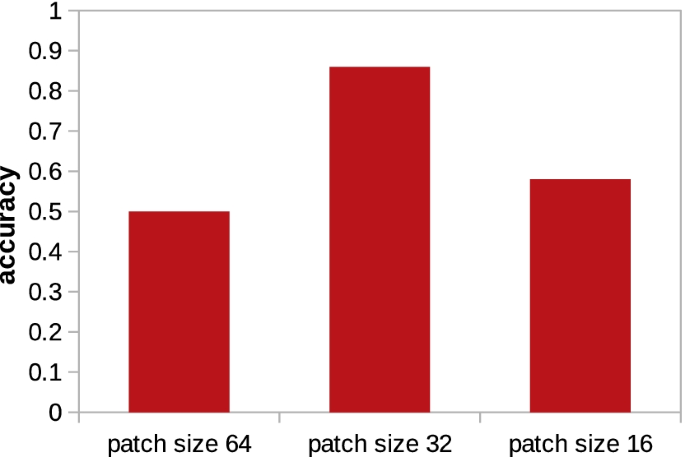
我们还研究了考虑3GL1等微小粒子局域化的模型的性能。这一点尤其重要,因为据报道,在二元分类场景中定位半径小的粒子是极具挑战性的[2]。我们使用大小补丁来训练3D-UCaps\ (64 ^ 3 \)和32 \ (^ 3 \)其中,前者为位于斑块中心半径为13的粒子的合理尺寸。我们观察到,根据探针颗粒选择合适的贴片大小起着重要的作用。从立方体减小补丁大小\ (64 ^ 3 \)体素,32 \ (^ 3 \)大大提高了性能。然而,考虑到网络的内核大小,补丁大小的减小有一定的局限性。有一次,我们将补丁尺寸减小到\ (16 ^ 3 \)时,由于内核较大,网络性能再次下降。减小微小颗粒的贴片尺寸有助于减少训练时输入系统的背景噪声。数字11将3D-UCaps的结果与大小为64、32和16的训练补丁进行比较。
性能
如前所述,通过使用Cuda 11和cuDNN 8,所有训练程序都是使用Nvidia Quadro RTX 6000 GPU实现的。在表5,我们显示了使用模拟数据和实验部分中报告的超参数集训练和测试模型所需的平均训练和推理时间。
讨论
本文提出了一种新的基于胶囊网络的深度学习方法,用于CET中大分子的分割。该网络由U-Net架构组成,其中编码器组件是3D CapsNet,而解码器基于传统的3D CNN,应用体积分割。此外,特征提取器用于初始化编码器的主胶囊。在多类场景下,该网络的性能优于3D-UNet,二值分类的结果也略有改善。
虽然用于训练两种模型的数据量相似,但3D-UCaps中可训练参数的数量大于3D-UNet。这意味着我们实现了一个更深层次的架构,可以用有限的数据进行训练,同时保持准确性和准确性\ (f \)得分。我们的消融研究支持通过在编码器组件中使用胶囊层来实现更高层数的想法。拥有更深层次的体系结构是很重要的,因为低级提取的特征仅仅揭示了被检测对象的抽象属性,比如边缘。然而,它是高级提取的特征,呈现更复杂的概念,如几何形状,局部性和层次结构的对象相对于其邻域。3D-UCaps网络的另一个优点是验证和测试结果之间的差异很小,表明没有发生过拟合。由于胶囊层同时提取了目标的存在概率和方向,因此这种结构在大分子重建或亚分子单元检测等下游任务中具有重要意义。
3D-UCaps模型的一个小缺点是训练的计算时间高,大约是3D-UNet的两倍。然而,一旦预先训练的模型可用,推理时间是合理的。
未来的工作包括降低模型复杂性和训练的计算成本,改进测试数据的分割结果,以及研究其他使用CapsNet的混合架构,其中提取的方向特征起着重要作用。
数据和材料的可用性
实验数据可应要求提供,用于研究目的。有兴趣的读者可以通过khavnekar@biochem.mpg.de与Sagar Khavnekar联系。此外,实验数据将在不久的将来在EMPIAR数据库中公开提供。
参考文献
陈超,林荣,曾鑫,Elmaaroufi K, Galeotti J,徐明。基于深度学习的电子冷冻层析成像大分子结构分类。马赫视觉应用,2018;29(8):1227-36。
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。深度学习提高了三维细胞低温电子断层图中的大分子识别。地理学报,2011;18(11):1386-94。
Al-Azzawi A, Ouadou A, Max H, Duan Y, Tanner JJ, Cheng J. DeepCryoPicker:基于深度神经网络的低温电镜单蛋白颗粒拾取。中国生物医学工程学报,2020;21(1):1 - 38。
李建军,李建军,李建军,等。基于模式识别的蛋白质复合物定位研究。方法细胞生物学杂志,2007;79:615-38。
王峰,龚辉,刘刚,李敏,闫超,夏涛,李翔,曾杰。基于深度学习的低温电镜粒子自动拾取方法。中国生物医学工程学报,2016;35(3):357 - 357。
朱勇,欧阳强,毛勇。基于深度卷积神经网络的低温电子显微镜单粒子识别。中国生物医学工程学报,2017;18(1):1 - 10。
刘建军,刘建军,刘建军,刘建军,刘建军,刘建军。基于卷积神经网络的低温电子显微图像提取技术。自然科学学报,2019;16(11):1153-60。
Wagner T, Merino F, Stabrin M, Moriya T, Antoni C, Apelbaum A, Hagel P, Sitsel O, Raisch T, Prumbaum D,等。SPHIRE-crYOLO是一款快速准确的全自动颗粒拾取器。生物学报,2019;2(1):1 - 13。
[0][0],张建军,张建军,张建军,等。三维U-Net:基于稀疏标注的密集体分割算法。医学图像计算和计算机辅助干预国际会议,pp 424-432(2016)。施普林格
Gubins I, van der Schot G, Veltkamp RC, Förster F,杜晓,曾晓,朱中,常亮,徐明,Moebel E,等。低温电子断层图的分类。SHREC ' 19 Track (2019)
徐敏,柴翔,Muthakana H,梁翔,杨刚,Zeev-Ben-Mordehai T,邢爱平。基于深度学习的电子冷成像中大尺度大分子结构恢复细分方法。生物信息学。2017;33(14):13-22。
Tran D, Bourdev LD, Fergus R, Torresani L, Paluri M. C3D:视频分析的通用特性。CoRR, abs/1412.0767 2(7), 8 (2014)
Sabour S, frost N, Hinton GE。胶囊之间的动态路由。arXiv预印本arXiv: 1710.09829(2017)
马志军,李建军,李建军,等。基于自关注路由的胶囊网络。arXiv预印本arXiv: 2101.12491(2021)
Patrick MK, Adekoya AF, Mighty AA, Edward BY。胶囊网络——一项调查。沙特国王大学计算机信息科学学报,2022;34(1):1295-310。
赵Y, Birdal T,邓H, Tombari f . 3 d点胶囊网络。参见:IEEE/CVF计算机视觉和模式识别会议论文集,pp 1009-1018 (2019)
阮涛,华宝生,黎宁。3D- ucaps:三维胶囊UNET的体积图像分割。医学图像计算和计算机辅助干预国际会议,第548-558(2021)。施普林格
达涛,丁杰,杨玲,祁立坚。一种基于CNN的低温电子显微镜全自动颗粒拾取方法。参见:2018年ACM生物信息学、计算生物学和健康信息学国际会议论文集,633-638页(2018)
Erdmann PS, Hou Z, Klumpe S, Khavnekar S, Beck F, Wilfling F, Plitzko JM, Baumeister W.原位冷冻电子断层扫描显示完整核仁核糖体生物发生的梯度组织。自然科学学报,2011;12(1):5364。https://doi.org/10.1038/s41467-021-25413-w。
wanw, Khavnekar S, Wagner J, Erdmann P, Baumeister W. Stopgap:一种子层析图平均和细化软件包。中国生物医学工程学报,2014;26(增刊1):516 - 516。
致谢
我们感谢所有为本研究提供必要设备和材料的参与者。
资金
由Projekt DEAL支持和组织的开放获取资金。这项工作得到了德国教育和研究部(BMBF)在Forschungscampus MODAL(项目资助3FO18501)的支持。由Projekt DEAL支持和组织的开放获取资金。此外,V.S.由柏林学习与数据基础研究所(BIFOLD)(项目BIFOLD- bzml 01IS18037H)资助。
作者信息
作者及单位
贡献
N.H.开发并实施了所提出的方法,进行了实验,并撰写了大部分手稿。d.b., r.b.和V.S.共同监督这个项目,并帮助修改手稿。s.k., f.b.和R.B.获得数据和/或提供访问权限。D.B.和R.B.获得了项目资金。所有作者已批准稿件并同意将其提交给BMC Bioinformatics。
相应的作者
道德声明
伦理批准并同意参与
不适用
发表同意书
不适用
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1
。关于实验数据集的补充信息。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
哈贾罗拉斯瓦迪,N.,桑卡拉,V.,哈夫内卡尔,S.。et al。利用胶囊网络在低温电子层析图中进行体积大分子鉴定。BMC生物信息学23, 360(2022)。https://doi.org/10.1186/s12859-022-04901-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04901-w
关键字
- 生物医学成像
- 细胞低温电子断层扫描
- 大分子识别
- 深度学习
- 胶囊网络