摘要
背景
控制染色质功能的基因的致病突变已涉及罕见的遗传综合征。这些染色质修饰剂在其影响的表观遗传变化的规模上表现出非凡的多样性,从DNMT1的单碱基对修饰到PRM1/2的全基因组结构变化。DNA甲基化的模式与整个表观遗传尺度上的一系列不同的表观遗传特征相关,使得DNA甲基化对于绘制一般表观遗传失调的区域有价值。然而,现有的方法无法直接从DNA甲基化数据准确地识别整个表观遗传尺度上的差异甲基化区域。
结果
为了解决这个问题,我们开发了DMRscaler,这是一种新的方法,使用迭代窗口程序来捕获从单个碱基对到整个染色体大小不等的差异DNA甲基化(DMRs)区域。我们的基准测试DMRscaler与几个DMR呼叫者的模拟和自然数据进行比较,比较XX和XY外周血样本。DMRscaler是唯一一种准确地在x染色体上调用大小从100 bp到1 Mb (pearson's r = 0.94)到152 Mb的DMRs的方法。然后,我们分析了来自染色质修饰基因突变的罕见疾病队列的甲基化数据NSD1,EZH2,KAT6A在哪里DMRscaler鉴定了涉及发育的新型DMRs跨基因簇。
结论
综上所述,我们的结果表明,DMRscaler是唯一能够捕捉整个表观遗传尺度上DMR特征的大小,并确定驱动人类疾病表观遗传失调的新型共同调节区域。
背景
调节染色质结构和功能的基因对胚胎内复杂发育轨迹的协调至关重要。这些染色质修饰基因的突变在自闭症临床队列中富集[1,2,3.,4]、先天性心脏病[5,6]和全面发育迟缓[3.,5].染色质修饰基因的致病性突变也可导致具有重叠和不同表型特征的特定综合征[7,8,9,10].虽然临床表型通常集中在一组常见的染色质修饰基因周围,但驱动这些表型的潜在分子机制还没有很好地描述。
染色质修饰剂在蛋白质复合物中起作用,结合染色质并形成基因组的物理和化学景观,即表观基因组。基因组中特定染色质修饰物发挥影响的区域对于定义其在发育中的作用至关重要。由染色质修饰剂控制的基因组区域可以高度定位,例如在单个胞嘧啶核苷酸的甲基化中,它调节某些转录因子(tf)的结合亲和力[11,12,13,14,15],或者它可以扩展到更广泛的染色质景观,就像在精子发生过程中发生的PRM1/2介导的基因组压实[16,17]或Xist将x染色体凝聚在具有多个x染色体副本的细胞中[18,19,20.].在局部和全局之间存在着从几十千碱基到许多兆碱基的中间尺度上的表观遗传特征的多样性。这些特征包括多梳抑制结构域(PRDs) [21,22,23]和拓扑学相关域[24]和共同调节的基因簇。这些中等大小的特征协调整个基因组发育中的高阶模式事件,如PRD调控Hox分割模式[25],或将嗅觉受体基因簇组织成TADs [26]与成员嗅觉受体基因相互依赖的表观遗传调节[27,28].全面了解染色质修饰剂需要了解其对表观遗传景观影响的规模。
虽然对于许多表观遗传特征之间的相互作用,因果关系的方向仍然是一个悬而未决的问题,但DNA甲基化(DNAme)的变化通常与表观遗传尺度范围内其他表观遗传特征的变化有关。DNAme是一个甲基与单个胞嘧啶核苷酸的共价加成,通常在CpG二核苷酸的背景下[29].而DNAme则直接改变了一组DNA结合蛋白的结合亲和力[11,12,13,14,15],它也与更高阶的表观遗传特征有关。在启动子和增强子上,DNAme与基因活性呈负相关[30.,31].在几十到几百个千碱基的prd中,DNA甲基化被多梳抑制复合物的拮抗作用所消耗[32,33],因此,PRDs上多梳活性的变化往往与差异甲基化有关[33].从DNAme模式可以可靠地预测活性和非活性染色质的兆级结构域[34],而在结肠癌中,据报道,DNAme的变化与这些兆酶大小的非活性域重叠[35].
表型变异和遗传异质性可以使罕见综合征的诊断具有挑战性。更具有挑战性的是解释在罕见疾病患者全基因组测序研究中发现的罕见遗传变异的临床意义。在缺乏明确的功能数据的情况下,这些遗传变异被注释为意义未知的变异(VUSs)。区分致病变异和良性变异的一种方法是从同一基因中已知致病突变的患者中识别差分DNAme的共同模式,这是一种甲基化特征[9,10,36].DNAme签名的存在表明,常见的表观遗传标记与特定基因的致病突变有关。然而,将观察到的DNAme变化与导致疾病的表观遗传机制直接联系起来仍然是一个开放的挑战。
尽管已知差异DNA甲基化特征的尺度存在多样性,但没有现有的方法可以从全基因组甲基化数据中识别差异甲基化(DMRs)的全尺度区域。相反,现有的方法被设计用于在单基因或增强子的范围内识别DMRs,这为理解完整的表观遗传结构提供了重要但不完整的信息。这在使用DNA甲基化在更广泛的表观遗传学背景下理解共调控基因和区域的动态方面留下了空白。
这里我们描述一种方法,DMRscaler这种方法可以准确地识别出不同甲基化的区域,这些区域可以跨越几个碱基对,也可以跨越那些存在于更大范围内的、跨越全球DNA甲基化图景中许多兆碱基序列的区域。我们通过模拟大小从100 bp到1 Mb的DMRs,并测试其相对于现有方法的性能,演示了我们的差分甲基化调用器的动态范围。此外,我们使用真实的甲基化数据来测试DNA甲基化的性别差异DMRscaler在最高水平上,它将x染色体称为一个单一的差异甲基化特征,同时仍然称为常染色体上的小的基因水平DMRs。最后,我们表明,染色质修饰基因的致病突变与大型和高度保守的基因簇的差异甲基化有关,如HOX而且PCDH基因簇。通过连接本地和全球,DMRscaler可以提供一个更广泛的观点,差异DNA甲基化结构。
实现
DMRscaler的主要动机是能够可靠而准确地识别可能存在于显著不同尺度上的差异甲基化区域。在DNA甲基化数据中,DNA甲基化被测量为样本中所有细胞基因组中给定CpG位点上的胞嘧啶甲基化的比例。这个比例是该位点的β (beta)值,β = 0表示完全未甲基化,β = 1表示完全甲基化。整个基因组中所有CpGs的β值分布遵循双峰分布(附加文件1:图S1)。DMRscaler将一组CpG探针作为输入,这些探针具有染色体、基因组位置和预计算的个体CpG水平显著性p值,以及所需的I型错误控制水平的个体CpG水平p值截止阈值。期望的I型误差控制的p值截止值的估计应该在单个CpG水平上进行,以避免识别代表与感兴趣条件不相关的CpG相关块的DMRs。估计该截断值的一种方法是用DMRscaler包,是重复个体CpG水平显著性测试与病例和对照标签的排列,并比较来自这些随机排列的CpG显著性值分布与真实的病例-对照分区,但也可以使用其他方法。通过处理p值输入,DMRscaler为用户提供了选择最适合其实验设计的统计测试的灵活性。
为了识别以差异甲基化CpGs(即差异甲基化区域或DMRs)为特征的区域,DMRscaler使用滑动窗口方案(图2)。1A、B)。窗口由相邻cpg的计数来定义,而不是由基因组区域的跨度来定义。使用相邻cpg的计数进行窗口定义DMRscaler不可知的CpG密度。这允许DMRscaler扫描低CpG覆盖区域,如异染色质,可能会错过使用CpG之间的距离参数。该方法的未来迭代可能允许指定固定的基因组间隔宽度来定义DMRs。然而,这里的一个限制是,它取决于货币DNA甲基化芯片上所包含的甲基化位点的选择。
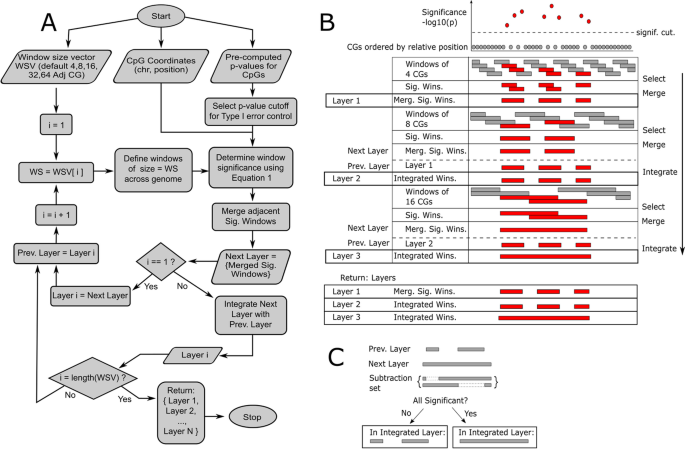
的轮廓DMRscaler方法。一个的决策树流程图DMRscaler.首先指定窗口大小向量,其中列出了用于识别每一层中的DMRs的窗口大小,以及CpG探测的坐标,其中包含为每个CpG预先计算的p值。此外,应使用这些预先计算的p值进行I型错误控制,方法是设置p值截止值,以确保识别出的DMRs与感兴趣的条件相关,具有预期的I型错误率。窗口显著性由公式(1).相邻的重要窗口合并形成Next_Layer.对于第一次迭代,返回Layer_1设置为这个Next_Layer,对于后续迭代返回Layer_i设置为积分的结果Next_Layer与Prev_Layer.层的集成在1C中描述。Prev_Layer更新为Layer_i在进行迭代i + 1之前。在生成最大窗口大小的层之后,将返回算法每次迭代的结果列表。B算法图形描述。顶部显示了按位置排序的cpg表示,并与一个显著值相关联。窗口被放置在有序的cpg之上,如果窗口得分显著,则选择窗口。然后合并相邻的窗口。如果一个Prev_layer已分配,然后进行集成。C集成过程。为每一个Next_LayerDMR,所有重叠Prev_LayerDMRs被鉴定出来。减法集是通过单独减去每个重叠部分而生成的Prev_LayerDMR来自Next_LayerDMR。减法涉及从Next_Layer.如果减法集的所有元素在使用窗口评分函数重评分时都是显著的,则Prev_Layer而且Next_Layer中合并区域Integrated_Layer,否则Prev_LayerDMRs用于Integrated_Layer.这个程序保证了更广泛Next_LayerDMRs只包括在没有单的情况下Prev_Layer该区域的重要性由DMR负责Next_Layer
区域显著性被认为是在一个窗口内通过随机机会获得CpG等级集或更极端等级集的概率,前提是该窗口中最显著的CpG已经被抽取出来。零假设是,一个窗口内CpG的等级与从CpG等级的完整集合中随机抽取所期望的相同或不那么极端,因为该窗口中最重要的CpG已被抽取。超几何检验序列的乘积用于确定函数所描述的每个窗口的区域范围显著性
窗口中的CpG,在排除了窗口中最重要的重叠CpG后,从最小到最大的顺序是从\(i = 1\)来\(i = m\).变量定义如下:
超几何累积分布函数,\ (hyper_{提供}\),则设置返回获得的可能性\ (k_{我}\)或者更多的成功案例\ (n_{我}\)从庞大的人口中抽取\ (N_{我}\)哪里有\ (K_{我}\)成功案例总数。在每一步\(i = 1\)来\(i = m\)时,函数决定拥有的概率\ (k_{我}\)或更多等级的cpg\ (K_{我}\)或更高\ (n_{我}\)随机抽取\ (N_{我}\)论文认定。变量在每一步都会更新,以说明在给定前一步中包含的信息的情况下可能性是如何变化的。对变量的更新使每个超几何测试的结果相互独立。然后,这些独立检验的乘积给出了区域显著性值,该值有效地表示了在给定大小的窗口内,通过随机机会将cpg与观测到的等级或更极端的等级相关联的概率。这实际上是将DMRs定义为通过FDR或FWER对照CpGs的邻接具有统计上显著相关性的区域。值得注意的是,这一程序意味着,虽然区域显著性与个人CpG水平显著性相关,但这两个显著性指标是不同的。例如,如果一个地区的最显著个体水平CpG显著值为p= 0.01,但区域显著性值为p= 1e−12,则该区域几乎可以肯定由真正通过相邻关联的CpG组成(例如,在某些方面作为相对于所有测量的CpG集合的单位进行调节),然而,该区域的差异甲基化是否真正与感兴趣的生物条件相关应基于单个CpG水平显著性。
由于可以测试显著性的窗口几乎与数据集中包含的cpg数量一样多,因此必须考虑多次测试,以避免过多的第一类错误。为了做到这一点,DMRscaler提供了使用Bonferroni校正程序来控制家族错误率的选项,或Benjamini-Yekutieli程序[37来控制错误发现率。用户提供了一个区域范围的显著性阈值,低于该阈值的区域被认为是显著的。这两个过程的实现使执行的测试数量等于低于用户指定的单个CpG水平p值截止点的测量探针数量。我们在模拟中观察到保守的FDR控制改变了个体CpG水平FDR阈值和区域显著性阈值(附加文件)1:图S2)。
为了定义在同一分析中可能在规模上发生巨大变化的DMRs,我们实现了滑动窗口过程,在迭代的每一步迭代地增加用于识别DMRs的窗口的大小。迭代的每一步调用的DMRs集合定义为a层用调用该层内DMRs的窗口大小命名的层,并以迭代步骤号为索引。例如,如果首先使用4个相邻cpg的窗口来调用DMRs,则layer_1(或层1)及4 _adjacent_cpg_layer是同义的。准确识别DMR尺度和避免DMR尺寸过度膨胀的一个重要步骤是包含跨这些层集成结果的步骤(图2)。1B,C).该集成过程的工作原理是,该方法从一层迭代到下一层,通过测试在当前层调用的暂时重要窗口在从前一层的每个重叠DMR中分别删除cpg后是否仍然具有统计意义。例如,如果当前层的一个给定窗口有100 cpg,并且暂时被认为是显著的,并且有两个来自前一层的重叠DMR,分别为20 cpg和30 cpg,当前层的DMR只有当从第一个重叠的前一层DMR中删除20个CpG DMR后剩下的80个CpG仍然被认为是一个显著区域时才被保留,并且从第二层重叠的前一层DMR中删除30个CpG DMR后剩下的70个CpG仍然被认为是一个显著区域时才被保留。否则,如果剩下的80 CpG或70 CpG中的任何一个都不重要,那么当前层不认为这100 CpG是DMR,而是将当前层设置为包括前一层的20 CpG和30 CpG DMR,从而从前一层传播这些DMR。
应该注意的是,没有进行额外的多重测试校正来解释用于识别DMRs的层数。由于每一层都依赖于单个cpg的相同基础层来估计显著性,因此跨层测试不是独立的,因此我们的直觉是,只在层内执行FDR或FWER校正是合理的。对这一层集成过程的依赖结构进行更严格的统计核算是我们留下的一个挑战,也是未来的研究方向。
这里将更正式地详细说明这个过程。首先,将最小窗口大小用作识别DMRs和构建的参数层1输出的。需要注意的是,术语“层”用于描述用给定窗口大小参数构造的DMRs的结果集,以表明算法每次迭代的结果之间的关系。每个连续层的构造可以扩展、添加或保留DMRs上一层,因此DMRs之间存在跨层的层次关系。低层中的DMR将始终映射到上层中的一些DMR,以确保这一点层我总是层我+1.定义下一层,对于第二大尺寸的窗口重复以下步骤:覆盖窗口,识别显著富集差异甲基化cpg的窗口,并合并重要窗口(图2)。1一个,B).从第二层开始,一个额外的步骤是集成从上一层执行。这是通过减法来实现的上一层DMR中任何重叠的暂定DMR下一层,并重新测试所有生成的简化DMR集下一层为显著性(图;1C).如果试探性DMR在下一层在每个重叠部分的减法之后仍然是重要的上一层DMR单独,然后试探性下一层DMR在new中被保留集成层.否则,重叠上一层dmr保留不变,取代暂定下一层DMR在集成层.此外,任何上一层的DMR中没有重叠的DMR下一层添加到集成层.通过这种方式,在算法的每次迭代中,DMRs都从上一层到集成层但从未输过。随着算法的进行,上一层是否更新到最新集成层在完成集成的下一步之前下一层.通过使用不断增大的窗口迭代调用DMRs并整合结果,DMRscaler能够识别在规模上变化巨大的dmr。
方法
细胞培养
对于arbolada - tham综合征数据,成纤维细胞系来自先证者和未受影响的父母一方或双方的皮肤穿刺活检。该项目由加州大学洛杉矶分校机构审查委员会#11-001087批准。所有个体水平数据在分析前去识别。通过UCLA病理研究门户创建成纤维细胞培养系,并在DMEM (Gibco™)、10% FBS(热灭活胎牛血清,Thermofisher)、1%非必需氨基酸(Gibco™)和1% PenStrep中建立和培养成纤维细胞系,温度为37℃,温度为5% CO2孵化器。每月对细胞株进行支原体检测。
DNA甲基化研究
在arbolda - tham综合征甲基化研究中,从患者来源的成纤维细胞系中提取DNA。每一行的具体突变在附加文件中给出1:表S1。DNA样本经亚硫酸氢盐转化后,如前所述,在Illumina甲基化EPIC阵列(850 k EPIC阵列)上运行[38]在加州大学洛杉矶分校神经科学基因组学核心生成idat文件。使用MINFI包对产生的idat文件进行QC,并去除重叠snp的探针[39].经QC检测,865,919个CpGs中有852,671个残留,去除性染色体CpGs后,有832,159个残留。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].
数据源
从基因表达综合(GEO)资源下载了对照组、韦弗综合征和索托斯综合征患者的公开外周血甲基化数据集[42,43]注册编号为GSE74432 [10].
模拟
为了演示如何DMRscaler区别于其他方法,我们模拟了不同甲基化区域(DMRs)的大小跨越几个数量级(图。2一个)。
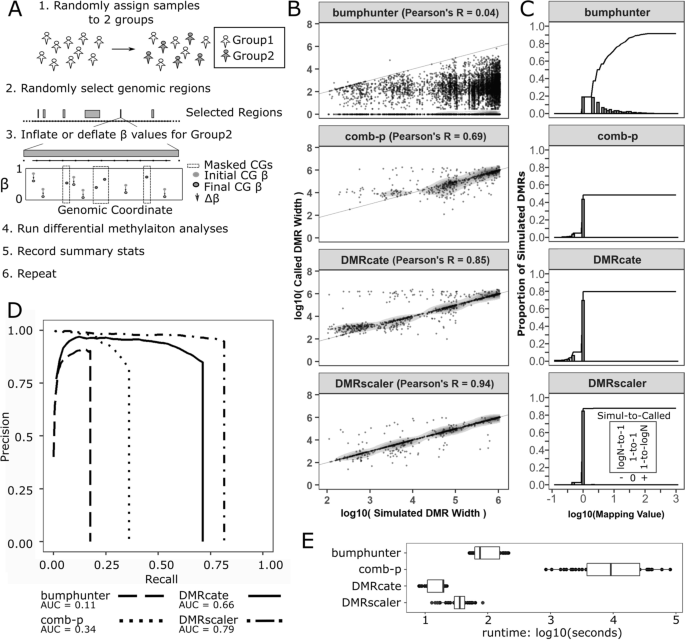
模拟大小在1kb至1mb之间的DMRs,以比较各种方法。一个仿真设计图形描述。首先,样本被随机分为两组。其次,随机选择基因组中不重叠的区域作为DMRs。第三,过选的DMRs中有一组的非掩模CpGs β值被Δβ膨胀或缩小。接下来运行所有的差异甲基化方法,并记录相关的汇总统计数据。重复此过程多次以生成额外的数据点。B模拟DMR宽度v称为DMR宽度以log10刻度绘制。如果两者之间有任何重叠,则在模拟和称为DMRs之间形成对。C映射值图。映射值是为每个模拟DMR计算的,它或者是共享重叠DMR的模拟DMR的数量的倒数,或者是与给定的模拟DMR重叠的所谓DMR的数量的倒数,以更极端的情况为准。日志值> 0表示每个模拟DMR调用多个DMR。值< 0表示多个模拟DMRs重叠单个称为DMR。Value = 0表示每个模拟的DMR调用一个DMR。绘制的线表示模拟DMRs到给定映射值的累积比例。D每种方法的特征级精度-召回率曲线,具体计算方法见方法。E对于每个参数集组合,每个方法在模拟数据集上运行的时间
在Infinium HumanMethylation450 BeadChip (450 K阵列)上从GEO (GSE74432)的53个对照的全血中测量的DNA甲基化被用作模拟的基础[10].以真实数据为基础,以捕捉DNA甲基化阵列数据中存在的自然生物学和技术变异性。使用MINFI包对产生的idat文件进行QC,并去除重叠snp的探针[39].在QC后,485,512个测量的CpGs中有468,162个被保留,去除性染色体后,456,514个测量的CpGs被保留。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].
人工引入DMRs的区域是在整个基因组中随机选择的,但受到以下限制。大小为0.1-1 kb的DMRs要求至少有3个cpg在450 K数组(cpg)上表示,大小为1-10 kb的DMRs要求至少有6个cpg,大小为10-100 kb的DMRs要求至少有9个cpg,大小为0.1-1 Mb的DMRs要求至少有12个cpg。此外,为了避免计数错误,引入的DMR与任何其他引入的DMR间隔至少10 cpg。附加文件中显示了模拟DMR中CpG计数与模拟DMR大小的分布1:图S3。
所有使用的450 k阵列样本都来自对照全血DNA,因此每次运行的模拟样本都被随机分为两组之一,Group1和Group2。每组由8个样本组成,没有从53个样本池中进行替换。
在人为地将DMR引入实际数据矩阵之前,每个DMR中cpg的比例(不包括第一个和最后一个cpg)由噪音参数随机掩蔽,保持其原始β值。这样做是为了模拟真实数据中相邻CpGs甲基化状态的变异性。的值。噪音测试参数分别为0、0.25和0.5,分别对应0%、25%和50%的cpg重叠模拟DMR被遮盖。然后测量Group1和Group2各DMR内CpGs的β均值。具有较大平均β值的组的β值将使所有非掩蔽cpg膨胀到由Δβ参数。模拟是用Δβ分别用0.1、0.2和0.4来模拟小、中、大效应DMRs。如果这导致任何样品的β值大于1,该CpG的β值除以该CpG的最大β值,使值回到0-1的范围。
在将人工DMRs引入数据集后,DMRscaler,bumphunter[44], comb-p[45),DMRcate[46]在数据集上运行并将结果制成表格。DMRscaler使用c(4,8,16,32,64)相邻CpG的window_size_vector进行测试,locs_pval_cutoff对应于个人级别CpGp-value, FDR < 10%, region_signif_cutoff = 0.01,对应于多次测试校正后将区域调用为DMR的区域级显著性阈值,region_signif_method = "benjamini-yekutieli",指定第一类错误控制方法为benjamini-yekutieli过程。BumphunterMaxGap = 1e6,启用黄土平滑。Comb-p用dist = 1e6, step = 5000, seed = 1e-3, region-filter-p = 0.1(附加文件1:图S4)。DMRcate用lambda = 1e6, C = 2000进行检验。选择方法的参数集是为了便于识别更大的DMRs,以便输出更具可比性DMRscaler.
为了对每种方法的性能进行基准测试,使用了几个指标,包括DMRs中差异甲基化的cpg的比例、精密度、召回率、特异性、F1、马修相关系数(MCC)和精确召回曲线下的面积(AUCPR)。这些指标被记录在特征、基对和CpG探针水平上进行分析,其中特征水平评估将每个模拟DMR视为单个正特征,基对水平将每个与模拟DMR重叠的基对视为正特征,CpG探针水平将每个与模拟DMR重叠的CpG探针视为正特征。基对和CpG水平评估是基于真、假阳性和阴性的直接计数。特征水平评估是在Tatbul及其同事提出的测量时间序列精度和查全率问题框架之后进行的,当对单个特征的识别感兴趣时,这通常适用于其他形式的范围数据[47].每个模拟的DMR都被认为是具有真阳性(TP)和假阴性(FN)属性的真实特征。每个所谓的DMR被认为是一个具有TP和假阳性(FP)属性的所谓特征。DMRs是根据所有方法的p值排序的。通过测量精度和召回率,逐步包含下一个最高评分或最显著的称为DMR,生成精度-召回率曲线。第n步测量精密度和查全率为
在哪里\ (TP_{{叫做\ _DMR_{我}}}\)为第i个DMR重叠模拟DMR区域的比例,\ (FP_{{叫做\ _DMR_{我}}}\)为第i个DMR不与模拟DMR区域重叠的比例,模拟\ \ (TP_ {{_DMR_ {j}}} \)为第j个模拟DMR与第1到n个DMRs重叠的比例,称为DMRs,模拟\ \ (FN_ {{_DMR_ {j}}} \)为第j个模拟DMR与第1到n个DMRs重叠的比例,称为DMRs,\ (n \)在第n步中使用的最有效的称为DMRs的数字,和\ \(米)为模拟DMRs的总数。精度和召回率的特征级测量给予每个模拟DMR相同的权重,因此大的模拟DMR不会主导信号。
除了精度和召回率,马利克等人最近对DMR呼叫者进行的基准测试中还包括其他几个指标。[48]来评估方法在仿真中的性能。所有这些指标都是根据所谓的DMRs进行测量的,DMRs具有调整后的区域范围显著性p每种方法均< 0.01。特异性是对真阴性率的衡量,以真阴性占总阴性特征的比例来衡量,并由以下公式计算:
错误发现率(False Discovery Rate)作为精度的倒数,给出错误结果的预期比例,为:
F1是测试准确性的衡量标准,由以下公式给出:
F1的范围从0表示最糟糕的准确性到1表示完美的分类。最后,马太相关系数,它是预测和真实类别标签之间相关性的衡量,由:
MCC值为+ 1表示完全分类,0表示与随机分类等效,−1表示完全错分类。
罕见病数据分析
对于每一个真实的数据分析,DMRscaler,bumphunter, comb-p,DMRcate被用来称为DMRs。DMRscaler使用c(4,8,16,32,64)相邻CpGs的window_size_vector进行测试,locs_pval_cutoff对应于实现FDR < 10%的个体级别CpG p-value, region_signif_cutoff = 0.01对应于多次测试校正后将区域调用为DMR的区域级别显著性阈值,region_signif_method = "benjamini-yekutieli",指定benjamini-yekutieli过程作为I型错误控制方法。DMRcate使用默认参数以及lambda = 1e6, C = 2000进行了测试,以捕获更大的DMRs,以便输出更类似于DMRscaler.Bumphunter采用默认参数,以及启用黄土平滑的MaxGap = 1e6进行测试。
对于性别分析,使用Infinium HumanMethylation450 BeadChip (450 K阵列)从53个GEO (GSE74432)对照的全血(29个女性和24个男性样本)中测量DNA甲基化[10].使用MINFI包对原始idat文件进行QC,并去除重叠snp的探针[39].经过QC, 485,512个测量cpg中有468,162个残留。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].女性和男性样本之间的个体水平差异CpG显著性使用Wilcox检验作为输入DMRscaler而且comb-p.每个方法的原始输出在附加文件中提供2:表S2。
在Arboleda-Tham综合征样本分析中,采用Illumina MethylationEPIC阵列(850 k EPIC阵列)对8例病例和12例对照组进行DNA甲基化检测。使用MINFI包对产生的idat文件进行QC,并去除重叠snp的探针[39].经过QC, 865,919个测得的CpGs中有852,671个保留,去除性染色体后的CpGs有832,159个保留。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].女性和男性样本之间的个体水平差异CpG显著性使用Wilcox检验作为输入DMRscaler而且comb-p.每个方法的原始输出在附加文件中提供3.:表S3。
韦弗分析中,用Infinium HumanMethylation450 BeadChip (450 K阵列)从8名患有糖尿病的患者的全血中测量DNA甲基化EZH2基因突变和来自GEO (GSE74432)的53个对照[10].这一数据来自一项研究,该研究发现了索托斯综合征特有的表观遗传特征NSD1使用韦弗综合征样本作为阴性对照的分类器[10].最近,这些数据被用于识别韦弗综合征特有的表观遗传特征[9].使用MINFI包对原始idat文件进行QC,并去除重叠snp的探针[39].在QC后,485,512个测得的CpGs中有468,162个仍然存在,去除性染色体后,456,514个测得的CpGs仍然存在。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].女性和男性样本之间的个体水平差异CpG显著性使用Wilcox检验作为输入DMRscaler而且comb-p.每个方法的原始输出在附加文件中提供4:表S4。
在Sotos综合征分析中,使用Infinium HumanMethylation450 BeadChip (450 K阵列)从38例Sotos综合征患者的全血中测量DNA甲基化NSD1基因突变和来自GEO (GSE74432)的53个对照[10].使用MINFI包对原始idat文件进行QC,并去除重叠snp的探针[39].这与韦弗综合症数据来自同一项研究[10].在QC后,485,512个测得的CpGs中有468,162个仍然存在,去除性染色体后,456,514个测得的CpGs仍然存在。使用背景校正对单个探针进行预处理和归一化[40]和函数归一化[41].女性和男性样本之间的个体水平差异CpG显著性使用Wilcox检验作为输入DMRscaler而且comb-p.每个方法的原始输出在附加文件中提供5:表S5。
证候DMR重叠分析
为了测试Arboleda-Tham、Sotos和Weaver综合征之间差异甲基化的重叠区域,考虑用于DMR检测的测量cpg的数量被降低采样,仅包括在Infinium HumanMethylation450 BeadChip (450 K阵列)和Illumina MethylationEPIC阵列(850 K EPIC阵列)上测量的cpg。剩下425733个测量的cpg用于调用DMRs。
DMRs之间的重叠和基因集的重叠被计算在综合征之间。基因集重叠被单独考虑,以识别通常可能存在差异甲基化,但由基因的非重叠区域识别的基因,这是直接DMR重叠测量会遗漏的部分,在附加文件中提供了区域水平和基因水平重叠的原始输出6:表S6。
为了测试被确定为属于DMR的CpG是否在综合征之间被富集,即一种综合征中的CpG与DMR的成员关系是否使其或多或少也可能属于另一种综合征中的DMR,我们计算了比值比(or)。OR由2 × 2列联表计算,列联表包括两种综合征中属于DMRs的cpg、属于其中一种而不属于另一种的cpg以及两种综合征中都属于DMRs的cpg。2 × 2列联表中使用的原始计数在附加文件中给出1:表S7。所有综合征对的优势比在附加文件中给出1:表S8。
结果
DMRscaler概述
我们的发展目标DMRscaler就是有一种方法能够准确地识别在整个表观遗传尺度范围内表现出差异甲基化的区域,从小启动子到全染色体尺度特征。实现这一目标的主要瓶颈是差异甲基化区域在相邻cpg之间的甲基化状态表现出显著的变异性。例如,距离0.5 - 1.0 kb的相邻cpg中,近20%的甲基化比例差异大于50%(附加文件)1:无花果。S5, S6)。当试图识别可能跨越更大基因组区域(如基因簇)的DMRs时,这种可变性使得将连续的重要cpg块作为DMRs的琐碎方法无效。解决这一高变异性问题的一种方法是根据相邻CpGs或某些特定的基因组间隔的显著性来平滑差异甲基化位点。然而,平滑方法对用于平滑窗口的带宽参数的选择很敏感。太小的窗口将无法连接较大间隙的特征,太大的窗口将导致过于宽的DMRs。因此,当特征在规模上发生巨大变化时,单靠平滑是不合适的。为了捕获潜在的噪声特征,这些特征可能在大小上有几个数量级的变化,从基对到多兆级,我们需要一种既能抗噪声又能准确确定特征大小的方法。
为了解决确定DMR大小的这些限制,DMRscaler在基因组上使用迭代滑动窗口(图。1A, B),表示为部分有序的测量cpg集合,并在滑动窗口的每次迭代之间实现一个积分步骤(图。1C).每一步的窗口识别了在病例和对照之间具有显著不同甲基化值的CpGs中富集的区域集。通过将CpGs放入窗口,并测试这些窗口以丰富重要的CpGs Eq。1),该算法对相邻cpg甲基化可变性引起的噪声具有鲁棒性。为了解决通过预选窗口大小参数引入的特征大小偏差,DMRscaler使用增加大小的变量参数迭代调用重要的Windows,并将每次迭代的结果与前一次迭代的结果集成在一起。积分步骤(图;1C)用于先前(较低)层(由较小的窗口构建)和当前(较高)层之间,以确定上层中的哪些特征已经由较低层特征充分表示,以及哪些上层特征捕获了较低层特征所错过的统计上显著的关联。如果上层特征捕获了低层中缺失的统计上显著的关联,那么上层特征将被保留,并与任何重叠的低层特征进行解析,否则重叠的下层表示将未经修改地进行。有关更详细的描述,请参见实现。
DMRscaler提供了一个解决方案的问题,识别DMR特征跨整个范围的表观遗传特征大小,无论是在碱基对水平或整个染色体。跨窗口过程迭代的结果集成DMRscaler实现是一种定义DMRs的新机制,可以推广到其他表观遗传特征或一维数据,其中定义感兴趣特征的组件的不连续是预期的,并且感兴趣的特征可能存在于显著不同的尺度上。
DMRscaler与现有方法的比较
我们接下来进行了基准测试DMRscaler三种常用的鉴别差异甲基化区域的方法:bumphunter[44),comb-p[45),而DMRcate[46)(表1).这些方法的一个显著区别是我们的方法,DMRscaler,而且comb-p将预先计算的p值作为while的输入bumphunter而且DMRcate使用t检验确定个体水平的CpG显著性。我们观察到,当样本量较小(每组n = 8)时,由非参数Wilcoxon和t检验确定的差异甲基化显著性之间相关性较差(附加文件)1:图S7)。由于我们的目标之一是开发一种方法,可以在比较罕见疾病数据集的研究中检测DMRs,因此根据实验设计和样本量限制,灵活地选择最合适的个体CpG显著性统计检验是可取的。虽然当样本量足够大(n > 30)或抽样分布近似正态时,t检验是合适的,但小样本中的差异甲基化分析打破了这些假设,因此在我们对罕见病数据集的分析中,使用Wilcoxon检验的灵活性非常重要。
差异甲基化调用者之间的第二个差异比较在这里(表1)在于他们通过建模来识别不同的甲基化区域。简单地说,bumphunter使用线性回归模型来识别在病例和对照条件之间甲基化差异的CpG位点。然后为了检测DMRs,bumphunter识别平滑后高于指定显著性阈值的相邻cpg的延伸。然而,相邻CpGs的甲基化格局是复杂的,高、中、低β值的CpGs混合在一起,使得定义大而连续的差异甲基化区域具有挑战性(附加文件)1:无花果。S5, S4)。Comb-p使用Stouffer-Liptak方法进行p值校正,然后将重要cpg分组在定义的窗口或窗口间隔内经销而且一步参数。DMRcate类似于bumphunter因为它还实现了线性建模(表1).DMRcate在M转换β值上使用高斯平滑函数来识别全基因组数据中的DMRs。这为用户提供了带宽参数lambda和控制参数C的控制,可用于识别较大的差异甲基化区域。然而,行为的DMRcate在较大带宽时,DMRs的定义较差,返回的DMRs大小往往对参数选择很敏感。有关方法的深入回顾,请参阅[48].
设计DMRscaler方法有几个独特的特点,使其能够更准确地识别更大的共调节区域。首先,它处理了甲基化分布在整个基因组的内在可变性,在分配显著性之前,将相邻的CpGs分成窗口。第二,DMRscaler整合了一系列窗口大小定义的窗口层的结果,以考虑在规模上显著不同的区域作为差异甲基化的潜在区域。为了适应各种各样的研究设计和限制,DMRscaler操作于预计算的p-个体水平CpG显著性的值。在这里,由于我们的罕见病队列的样本量小,我们使用Wilcox测试,其他方法生成p-values可以用来模拟协变量的影响。总之,这些特征允许在大动态范围内对差异甲基化区域进行可靠检测,跨越碱基对到兆酶分辨率,并允许在罕见疾病队列中和在其他生物条件(如染色体性别)之间检测差异甲基化的新区域。
DMRscaler在模拟甲基化数据中准确地捕捉从碱基对(bp)到兆酶(Mb)大小的表观遗传特征的尺度
除非另有说明,在以下部分中,DMR假定是最广泛的顶层,即第5层,它使用所有较低的层构建,是DMR特征的最准确表示。
为了将我们的方法与现有的方法进行比较DMRscaler来bumphunter comb-p,而且DMRcate在几个指标上突出了在广泛的模拟DMR大小上调用DMR的行为。这些指标包括:每种方法调用的模拟DMR和模拟DMR之间的相关性,每种方法能够将单个模拟DMRs表示为单个统一特征的映射值或程度,以及每种方法的运行时间。此外,我们使用标准的评价指标,如精密度、召回率、特异性、F1、马修相关系数(MCC)和精密度-召回曲线下面积(AUCPR)来评估方法性能。
我们首先模拟了来自对照血液样本甲基化数据中的DMRs (GSE74432) [10],大小从100bp到1mb不等。2A,详见方法)。在我们的模拟中,我们模拟了在真实数据中观察到的情况,其中相邻的cpg通常具有不同的甲基化状态噪音参数,表示模拟DMRs中cpg的比例,不包括第一个和最后一个cpg,这些cpg在随机选择的放在Group1和Group2的样品中没有差异甲基化。在我们的模拟测试中噪音参数值为0%、25%和50%。的Δβ参数用于模拟DMRs控制差异甲基化程度,其中Δβ为Group1和Group2中样品在无噪声CpGs引入时甲基化比例的差值。模拟运行与Δβ参数设置为值0.1、0.2和0.4分别模拟差异甲基化的小、中、大效应值。使用这些参数的每个组合运行的模拟结果包含在附加文件中1:图S8、S9、S10及附加文件7:表S9。在使用这些参数组合运行的模拟中,方法的相对性能和行为是一致的,因此在主要文本和图形中,我们显示了使用这些参数组合运行的模拟的结果和报告指标噪音= 50%和Δβ= 0.2。
DMRscaler能够准确地将模拟DMRs的大小(pearson 's r = 0.94)相对于bumphunter(pearson 's r = 0.04),comb-p(pearson 's r = 0.69)DMRcate(pearson 's r = 0.85)2B)。DMRscaler在模拟的DMRs和被调用的DMRs之间保持了强的1对1的关系,85%的模拟DMRs被准确地调用DMRscaler比例为1比1,而为19%bumphunter, 44%支持comb-p,而69%的DMRcate(无花果。2C)。
为了测量差异甲基化调用器的性能,我们计算每个测试的AUCPR。AUCPR将被调用的特征的精度度量(被调用的真实特征与所有被调用的特征的比率)和召回率(被调用的真实特征与真实特征总数的比率)结合为一个值,AUCPR = 0表示没有分类,AUCPR = 1表示所有特征的完美分类,没有假阳性。在我们的模拟中,DMRscalerAUCPR为0.79,bumphunterAUCPR值为0.11,comb-p的AUCPR为0.34,而DMRcateAUCPR为0.65(图;2D, AUCPR计算详见方法)。的低AUCPRbumphunter与模拟的DMR区域和所谓的DMR区域之间观察到的低的、轻微的负相关是一致的。2B).这种弱相关性是由于bumphunter严格要求显著差异甲基化cpg是相邻的,以便属于一个共同的DMR,因此将模拟的DMR特征分解为许多更小的特征。的低AUCPRcomb-p是低召回率的结果,功能的大小远远小于经销而且一步参数。属性的较低值经销参数增加了检测较小DMR特征的能力,但以检测较大DMR特征为代价(附加文件1:图S4),在非常大的值时经销运行时变得禁止,特别是当使用更小的步长值时(附加文件1:图S4)。DMRcate有一个相当高的AUCPR,然而,根据带宽参数lambda和控制参数c的选择,在调用DMRs的大小上有一个偏差,具体来说,在1mb和1kb左右有一个多余的DMRs错误调用(图。2B和附加文件1:图S11),这与带宽参数λ(设置为1 Mb)和缩放参数C (λ/C的比值设置为500)的选择有关(附加文件)1:图S12)。我们的数据表明DMRcate能够识别更大的DMR,但所谓的DMR大小对lambda和C参数的参数选择很敏感。的精度-召回曲线的形状支持这一点DMRcate这表明,随着回忆的增加,准确性会有适度的下降DMRcate引起更严重的误报处罚DMRscaler.
虽然使用Wilcoxon检验来生成用于模拟和真实数据分析的个体水平CpG显著性的p值,但我们注意到DMRscaler当使用t检验时,在模拟中具有可比性(附加文件1:图S13)。此外,虽然这些结果集中在顶层的结果DMRscaler,每个较低层的行为显示在附加文件中1:图S14。
比较每种方法在Δβ(0.1、0.2和0.4)和噪声(0%、25%和50%)参数的每种组合上的精度、召回率、特异性、F1、MCC和AUCPR指标,DMRscaler除了特异性,bumphunter始终是表现最好的方法,尽管各方法在特异性上的差异通常很小(表2,附加文件7:表S9)。这些结果进一步证明了这一点DMRscaler在广泛的特征大小范围内准确调用DMRs表现良好。
而DMRscaler与其他方法相比,该方法在大范围尺度上识别DMRs的任务表现良好,在计算时间方面也表现良好,与其他具有较大窗口大小类似参数的时间效率方法相比。平均DMRscaler需要30秒到1分钟才能完成一次运行,bumphunter大约需要1到3分钟,然后DMRcate呼叫DMRs只需要大约10秒。Comb-p,哪个使用了类似滑动窗口的机制DMRscaler,用给定的参数集完成每一次运行需要一个小时。2E)。
仿真结果表明DMRscaler通过调用和模拟DMR尺寸相关性、映射值、精度和召回率来测量,在广泛的DMR特征尺寸范围内,比其他方法更准确地重建DMR特征的尺度。此外,在其他性能测量中,包括特异性,F1, MCC和AUCPR,DMRscaler与其他方法相比,在具有DMRs的模拟数据集上始终表现良好,这些数据集在规模方面变化很大。
46, XX和46,XY个体之间的差异甲基化在经验数据中捕捉了染色体范围和基因特异性的调控特征。
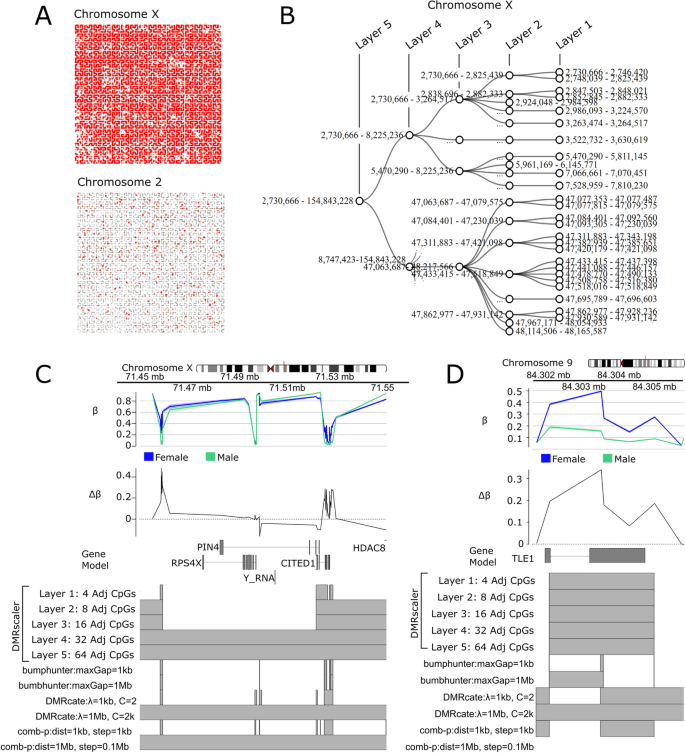
为了在真实世界的DNA甲基化数据中验证我们的假设,我们试图确定我们的方法是否可以捕捉常染色体差异甲基化的小区域以及染色体范围的特征,如x染色体失活。因此,我们的测试用例是女性x失活的自然发生,其中x染色体的一个副本在lncRNA的作用下大部分失活Xist[18,19].这一失活过程与x染色体上男性和女性DNA甲基化的显著染色体范围差异相关(图2)。3.与常染色体(如2号染色体)相比,差异甲基化区域的大小为103 bp至873 bp(图1)。3.A,底部,附加文件2:表S2)。在所有染色体中,DMRs的大小称为DMRscaler跨度为98 bp至152 Mb,与检测到的DMRs的规模相差150万倍DMRscaler.
XX和XY个体间的差异甲基化分析。一个chrX和chr1的希尔伯特曲线。希尔伯特曲线是通过cpg沿着给定染色体的位置排序而构建的。红点为不同甲基化的CpGs, FDR < 0.1。点大小缩放到最大显著性水平(-log10 p值)。B所调用的DMRs的层次关系对角线网络图DMRscaler第4、3、2和1层(分别相当于32个16,8和4个Adj. CpG层)为x染色体。CChrX: 71.4-71.6 Mb。GVIZ轨道堆栈图。顶部轨迹显示每组的平均β值,下一个轨迹显示Δβ,其中Δβ = β女−β男性.在基因模型轨迹下面是DMR轨迹,突出显示每个结果层的DMR区域DMRscaler(1、2、3、4、5层相当于4、8、16、32、64 Adj. CpG层)和每个竞争方法。(D) Chr9:84.302-84.306 Mb.音轨与3C相同
从图中可以直观地看出两性甲基化差异的程度。3.A,我们接下来比较了差异甲基化分析的结果使用DMRscaler,bumphunter,comb-p而且DMRcate.DMRscaler是唯一一种将观察到的差异甲基化整合到横跨98% x染色体的单个DMR的方法(表3.,无花果。3.B、附加文件1:图S15)。即使maxWidth参数设置为1 Mb,Bumphunter在x染色体上报告了1162个独特的DMRs,中位宽度为531 bp (IQR: 1 bp - 1.21 kb)(附加文件)1:图S16,附加文件2:表S2),可能是由于缺乏生成非差异甲基化cpg的机制。标准参数集dist = 1 kb, step = 100 bp,comb-p在x染色体上报告了2390个独特的DMRs,中位宽度为2 bp (IQR: 2 bp - 963 bp)3.,附加文件1:图S16,附加文件2:表S2)。更广泛的参数集dist = 1 Mb, step = 100 kb,comb-p在x染色体上被称为19个唯一的DMRs,中位宽度为3.15 Mb (IQR: 512 kb-8.54 Mb)3.,附加文件1:图S16,附加文件2:表S2)。DMRcate在默认设置下,x染色体上报告了1178个独特的DMRs,中位宽度为1.09 kb (IQR: 616 bp-1.68 kb)。当DMRcate提供了更大的带宽参数(lambda = 1 Mb, C = 2000),它改善了DMRs的巩固,但仍然报告了15个独特的DMRs(中位数宽度:3.95 Mb, IQR: 1.00 Mb - 17.89 Mb)。有关所谓DMR大小的完整分布,请参阅附加文件1:图S17,附加文件2:表S2。
DMRscaler使用不断增大的窗口迭代调用类dmr区域,同时将每次迭代的结果集成到DMRs的下一层。而最顶层是的主要输出DMRscaler,当考虑跨所有层的结果列表时,该过程产生了DMRs的嵌套层次结构,允许对差异甲基化体系结构进行细致入微的观察。在无花果。3.B,这是x染色体的一个子集。在这里,第1层的DMRs被巩固在第2层的DMRs中,然后第2层的DMRs被巩固在第3层的DMRs中,第3层的DMRs巩固到第4层,最终DMRs巩固为一个横跨整个x染色体的单一特征(图2)。3.B)。
虽然整个x染色体可以被认为是一个不同甲基化的特征,但已经确定的是,x染色体上有一小部分基因逃脱了x失活和DNA甲基化[49].当比较女性和男性之间的甲基化时,我们期望x -失活会导致性别之间的甲基化差异,与男性相比,女性整个x染色体上的高甲基化和较小程度的低甲基化[50].因此,我们预计两组之间的Δβ值为零或接近零的区域将富集在由于这些位点相对缺乏差异甲基化而避免x失活的区域。一个这样的区域的例子是两个DMRs的间隙,它持续到第3层和第4层之间的整合,发生在chrX: 71,459,274-71,521,494,这对应于基因RPS4X(无花果。3.C, S18),已知可以避免x失活[51].为了测试DMRs映射中缺口的这种趋势是否在逃避x失活的区域中更普遍地存在,我们对已知逃避x失活的基因重叠的CpGs进行了富集测试,并且在DMRs的每一层中调用CpGs重叠缺口DMRscaler的输出。Balaton等人在2015年的一项研究中报道,已知在x失活中逃逸或沉默的基因的共识被用于富集测试[52].在层1、层2、层3和层4中,分别由相邻的4、8、16和32个cpg的窗口定义,DMRs之间的CpGs重叠间隙与逃脱x失活的CpGs重叠基因之间的优势比为OR = 7.57 (95% CI 6.38-8.99;p-value = 1.04e-134 Fisher精确检验),OR = 7.24(95%可信区间6.07-8.65;p-value = 5.93e-100 Fisher精确检验),OR = 51.99 (95% CI 30.38-90.33;p-value = 4.34e-77 Fisher精确检验),OR = 160.44 (95% CI 25.42-6,396.92;p-value = 5.93e-100 Fisher精确检验)。在第5层未检测到富集,因为整个x染色体巩固为单一特征。Bumphunter同样显示出大量富集,比值比估计在OR≈10-20之间,然而,如前所述,bumphunter不能巩固x染色体上的DMRs,以识别整个x染色体是差异甲基化的。Comb-p而且DMRcate每个人都观察到DMRs与逃避x失活的基因ORs≈1-3之间的差距更小(表1-3)4).这些结果表明,虽然横跨x染色体的顶层DMR与x失活现象最直观地相关,但复杂DMR的层次结构的探索是由DMRscaler可以揭示生物学上有意义的特征,比如x -失活的基因逃逸模式。
x染色体内DMRs的复杂等级关系与常染色体的DMRs形成对比。常染色体上的DMRs几乎没有分支,这意味着这些DMRs在算法的每次迭代中都是稳定的(附加文件)1:图S15)。在chr9: 84,302,344-84,304,414处的一个这样的DMR的基因组视图突出了这种稳定性,其中在算法的第一层被识别为DMR的特征在后续的每次迭代中都是稳定的(图。3.D、附加文件1:图S18)。的基因TLE1与此DMR重叠,之前已被确定为一种在男性和女性之间甲基化差异的常染色体基因[53,54].
不同性别间甲基化差异分析的结果突出了DMRscaler在识别差异甲基化特征,存在于实际数据中显着不同的规模。这种能力DMRscaler现有的方法不能识别较大的DMRs,同时保持较小的DMRs的稳定性,例如DMRcate而且comb-p,或者倾向于将较大的DMR分割为许多较小的特征,如bumphunter.简要分析了由此产生的层次结构DMRscaler的层合并机制揭示了这一点DMRscaler可以捕获DMR中有生物学意义的结构,比如x失活逃逸。这种更完整地表示DMR结构的能力得到了突出体现DMRscaler的潜在价值,可以作为探索不同尺度下表观遗传调控特征之间相互作用的工具。
罕见的染色质修饰剂综合征包含差异甲基化区域,跨越对发育至关重要的基因簇
接下来,我们分析了几种罕见疾病的染色质修饰基因的DNA甲基化数据集,看看是否DMRscaler揭示了新的DMR特征,否则现有方法可能会遗漏这些特征。除非另有说明,在以下部分中,DMR假定是最广泛的顶层,即第5层,它使用所有较低的层构建,是DMR特征的最准确表示。
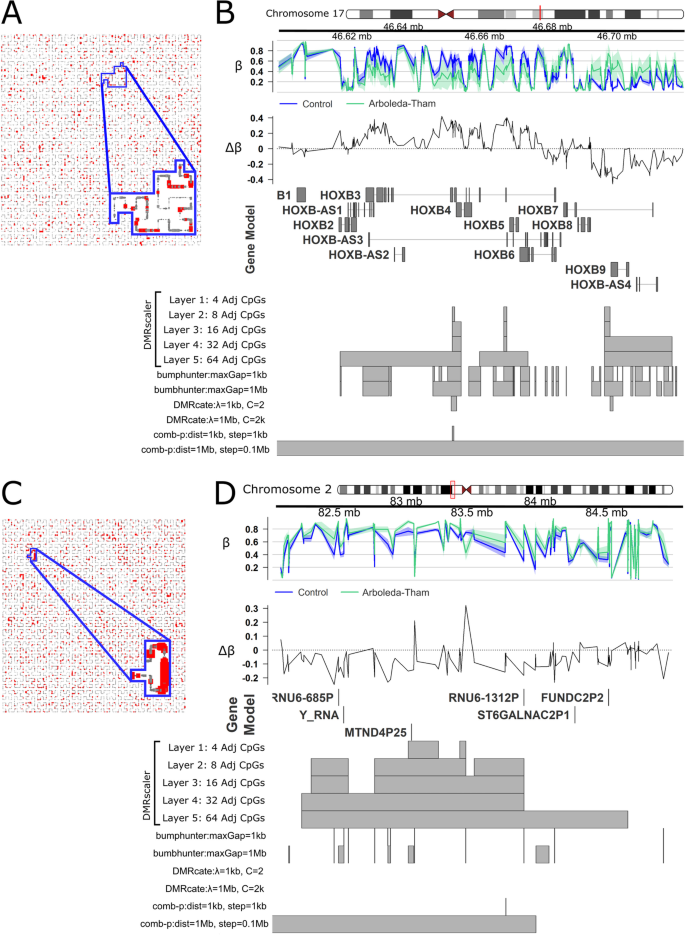
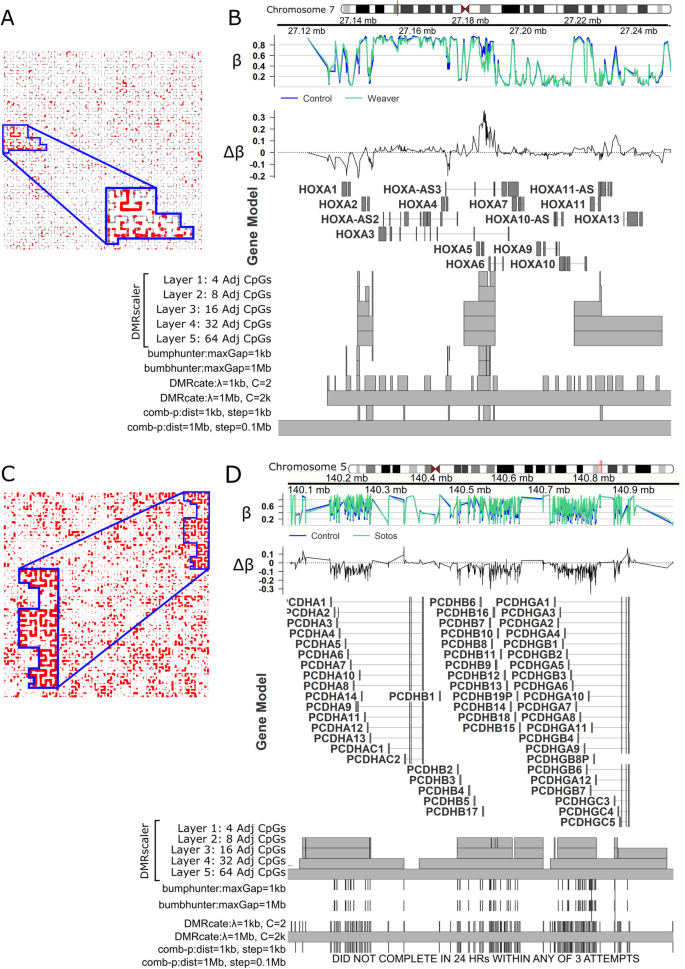
首先,我们比较了Arboleda-Tham综合征患者成纤维细胞的DNA甲基化谱与对照样本。该分析包括20个样本,其中8个患者和12个对照组(附加文件)1:表S1)。所有患者均由Kennedy等人报道。[7].在我们的分析中,DMRscaler鉴定出390个独特的DMRs,中位宽度为144.59 kb (IQR: 21.1 kb - 481.2 kb),总基因组覆盖率为4.9% (151.35 Mb)(附加文件)1:表S10,图S19)。在HOXB鉴定出三个独特的DMRs基因簇。的第一和第二DMRs区域重叠HOXB2, HOXB3而且HOXB4与对照组相比,阿波莱达-谭综合征患者的甲基化程度较低。第二部分与HOXB5和HOXB6在Arboleda-Tham综合征患者中也是低甲基化的。第三个跨度HOXB9与对照组相比,Arboleda-Tham综合征患者的甲基化程度较高。4A,B,附加文件1:图S20)。Bumphunter在这个地区有更多高度碎片化的dmr,包括被遗漏的地区DMRscaler.这可能是由于bumphunter所调用的区域具有很大的方差,Wilcox检验用于预先计算p值comb-p而且DMRscaler比bumphunter使用的t检验更保守。Comb-p1 Mb的大距离参数称整个区域为差异甲基化。DMRs对基因组的相对较大的覆盖范围主要是由跨相对基因稀疏区域识别的多兆级DMRs驱动的,其他方法都无法巩固comb-p使用1mb的距离参数(例如图4C,D,附加文件1:图S20)。
Arboleda-Tham综合征的差异甲基化分析。一个从chr17开始的CpGs希尔伯特曲线,勾勒出chr17:46.59 - 46.73 Mb对应的区域HOXB集群。FDR < 0.1的cpg以红色高亮显示。点大小缩放到最大显著值。Bchr17:46.59 - 46.73 Mb。HOXB集群。GVIZ轨迹叠加图。顶部轨迹显示每组的平均β值,下一个轨迹显示Δβ,其中Δβ = β控制−βArboleda-Tham.在基因模型轨迹下面是DMR轨迹,突出显示每个结果层的DMR区域DMRscaler从每一种竞争方法。CChr2:81.5-84.5 Mb。设计与4A相同。DChr2:81.5-84.5 Mb。音轨与4B相同
韦弗综合征(mim# 277590)是一种罕见的过度生长障碍,是由基因从头突变引起的EZH2一种组蛋白甲基转移酶。将韦弗综合征患者样本与对照组进行比较,DMRscaler共鉴定出226个独特的DMRs,中位数宽度为8.88 kb (IQR: 1.92 kb - 30.04 kb)。这些区域共占基因组的0.40% (12.34 Mb)1:表S11,图S21)。
在HOXA基因簇,DMRscaler确定了与韦弗综合征相关的三种不同的DMRs。第一个跨度HOXA1-HOXA2在韦弗综合征中轻度高甲基化,第二种包括HOX5的最后两个外显子HOX6与对照组相比,韦弗综合征患者的甲基化程度较低。第三个DMR覆盖的第一个外显子HOXA10,还有HOXA11,而且HOXA13.韦弗综合征的第三个DMR通常是弱的高甲基化,在上游有一个小但显著的低甲基化区域HOXA11.其他方法都报告了重叠这些簇的DMRs,但它们要么是碎片化的,要么是过宽的(图2)。5A, B,附加文件1:图S22)。
韦弗(A,B,C)和索托斯综合征(D,E,F)的差异甲基化分析。一个从chr7开始的CpGs希尔伯特曲线,勾勒出chr7:27.1 - 27.3 Mb对应的区域HOXA集群。FDR < 0.1的cpg以红色高亮显示。点大小缩放到最大显著值。Bchr7:27.1 - 27.3 Mb。HOXA集群。GVIZ轨迹叠加图。顶部轨迹显示每组的平均β值,下一个轨迹显示Δβ,其中Δβ = β控制−β韦弗.在基因模型轨迹下面是DMR轨迹,突出显示每个结果层的DMR区域DMRscaler从每一种竞争方法。CChr5:140.1-140.8 MbPCDH集群。设计与4A相同。D.Chr5:140.1-140.8 MbPCDH集群。轨迹与4B相同。除了Δβ = β控制−β索托斯
最后,我们还分析了Sotos综合征(mim# 117550),这是一种由核受体结合SET结构域蛋白1的截断和错义突变引起的过度生长综合征(NSD1)基因(55].DMRscaler分析发现1776个独特的DMRs,中位宽度为555.13 kb (IQR: 156 kb - 1.40 Mb),覆盖了71%的基因组(2.17 Gb)DMRcate其中282个DMRs横跨77%的基因组(附加文件1:表S12,图S23)。我们在32 Adj CpG层发现了三种独特的DMRs,它们跨越了原钙粘蛋白的基因簇。这些DMRs是由突变引起的NSD1覆盖周边Protocadherin(PCDH)基因簇PCDHA,PCDHB,PCDHGB(无花果。5D,E, S22)编码大型跨膜蛋白,这些跨膜蛋白对从细胞信号传递到树突树突树突化等多种过程至关重要[56].一个DMR横跨的第一个外显子PCDHA1-PCDHA12,另一段横跨PCDHB2来PCDHB19P的第3个外显子PCDHGA3-PCDHGA12而且PCDHB1-PCDHGC5.所有的DMRs都涵盖了这些PCDHSotos综合征的聚类相对于对照组高甲基化,尽管值得注意的是,这些聚类的CpG β值高度可变,反映了前面描述的相邻CpG异质性的一个例子(图2)。5D、附加文件1:无花果。S22, s4, s5)。值得注意的是,只有参数λ = 1 Mb和C = 2000的DMRcate也能够调用该区域的DMR,但是它缺乏一种机制来查看内部结构,该结构表明这三个集群中的每一个都被捕获的无差异甲基化cpg区域所分离DMRscaler的分层输出。
这些结果在罕见的染色质修饰剂综合征突出DMRscaler的在识别差异甲基化的模式,存在于更广泛的基因组特征,如基因簇的效用。
差异甲基化重叠区分析
在对每种综合征进行单独分析后,我们询问Arboleda-Tham、Sotos和Weaver综合征之间是否存在共同的甲基化差异区域。在Arboleda-Tham综合征和Sotos综合征之间,我们确定了652个DMR重叠的区域(Arboleda-Tham综合征占总DMRs的77.3%,Sotos综合征占总DMRs的4.7%),以及458个基因与两种综合征的某些DMR重叠(Arboleda-Tham综合征的基因与DMR重叠的比例为11.4%,Sotos综合征的基因与DMR重叠的比例为3.1%)。在Arbolelda-Tham综合征和Weaver综合征之间,我们确定了48个DMR重叠的区域(Arbolelda-Tham综合征占总DMRs的1.3%,Weaver综合征占总DMRs的14.1%),并且在两种综合征中都有39个基因与DMR重叠(Arbolelda-Tham综合征中有13.2%的基因与DMR重叠,Weaver综合征中有5.6%的基因与DMR重叠)。在Sotos综合征和Weaver综合征这两种生长障碍中,我们鉴定出414个区域(Sotos综合征占总DMRs的0.7%,Weaver综合征占总DMRs的91.0%)和282个基因与某些DMR重叠(Sotos综合征中5.9%的基因与DMR重叠,Weaver综合征中93.1%的基因与DMR重叠)(附加文件)6:表S6)。
为了检验重叠的显著性,我们测试了每对综合征之间重叠的优势比(OR)。为了简化分析,使比值比的测量在形式上更接近于DMRscaler方法中,我们只使用测量的cpg计数(详见方法)。从本质上讲,优势比测试的是,在一种综合征的DMRs中发现的CpGs是否富集于另一种综合征的DMRs中发现的CpGs。OR的置信区间(CI)重叠1表示无富集,接近0或大于1表示富集程度较高。用于计算OR的原始重叠计数在(附加文件1:表S7),比值比报告在(附加文件1:表S8)。Sotos和Weaver的优势比最高,OR = 17.16 (95% CI 12.27-23.99,p= 1.9e-32 Fisher 's精确测试)。Sotos和Weaver之间的OR降至OR = 1.55 (95% CI 1.47-1.64,p= 4.6e−56 Fisher 's精确测试)在第5层,这可能是由于第5层Sotos综合征中DMR特征的广泛基因组覆盖。第1层Arboleda-Tham和Weaver的比值比第二高,OR = 9.94 (95% CI 5.83-16.94,p= 4.7e-10 Fisher精确检验),在第5层OR = 3.00时显著性增加,但幅度下降(95% CI 2.71-3.31,p= 3.1e-77费雪精确检验)。Arboleda-Tham与Sotos在Layer 1的OR值较小,OR = 1.72 (95% CI 1.43-2.06,p= 5.6e−8 Fisher's精确测试),并在第5层下降到无意义(附加文件1:表S8)。这些结果显示了这里分析的三种综合征中的每一种的DMRs如何在这里测试的每一种其他综合征的DMRs中的CpGs中富集DMRscaler其中CpG的差异密度需要更高,特别是两种过度生长疾病之间的强烈富集为这些疾病的共同表观遗传效应和潜在的共同促成因素提供了证据。
索托斯和韦弗综合症的一个重叠区域是重叠区域INS,而且INS-IGF2近端,IGF2.这个区域与另一种生长障碍——贝克威-魏德曼综合征(BWS)有关。[57].索托斯综合症的DMR就在上游IGF2和重叠INS而且INS-IGF2(Δβ > 0.2)(附加文件1:图S24A)。韦弗综合征的DMR与IGF2基因重叠,由效应值较小的位点(Δβ ~ 0.05)组成,在韦弗综合征的一个区域高甲基化IGF2基因体与低甲基化进一步上游重叠INS而且INS-IGF2基因。上游的IGF2重叠INS而且INS-IGF2Sotos和Weaver综合征的低甲基化模式是一致的(附加文件1:图S24B,附加文件6:表S6)。
在所有三种症状中,有49个基因重叠了一些DMR,称为DMRscaler.其中包括Pcdhga1, pcdhga2, pcdhga3, pcdhga8, pcdhga10, pcdhgb7, pcdhga11,PCDHGA12,而且PCDHGC3的PCDHG聚类基因,之前仅在索托斯综合征的背景下讨论过。这些基因是值得注意的,因为它们涉及神经发育。的PCDHG如前所述,Sotos的聚类广泛高甲基化(图。5C, D)。在arbolada - tham综合征中,大多数患者的DMR间基因较小PCDHG这个簇中的基因位于5 '末端PCDHGC3.在韦弗综合征中,在同一5 '端延伸处有轻度低甲基化的DMRPCDHGC3Arboleda-Tham横跨到共享的3 '端PCDHG基因。其他评估的方法很少能够识别这一区域PCDHGArboleda-Tham综合征或Weaver综合征的基因簇(附加文件)1:图S25)。
Arboleda-Tham、Sotos和Weaver综合征之间重叠的DMRs和重叠DMRs的基因揭示了许多差异甲基化共享区域和具有差异甲基化模式的共享基因。在任何一种综合征的DMRs中的CpGs在所有三对综合征(Arboleda-Tham:Sotos, Arboleda-Tham:Weaver, Sotos:Weaver)的DMRs中的CpGs都是丰富的,通过优势比来测量。然而,这些数据来自不同的细胞类型(成纤维细胞vs血液),除了由基因突变引起的变化外,还表现出细胞类型的特定变化。这些结果表明,虽然每种综合征都有不同的差异甲基化特征,但在反映共享表型特征的区域也有显著的重叠。
讨论
我们新方法的关键发展,DMRScaler这是对现有方法的重大改进,能够在整个表观遗传尺度范围内准确识别DMRs的大小。
性别间甲基化差异分析DMRscaler结果表明,我们的算法可以处理模拟和真实样本中存在的所有DMR特征。看看XX和XY个体之间的DMRs,DMRscaler能识别出一个小的2.1 kb的DMR重叠在常染色体基因上TLE1这之前被认为是性别甲基化的差异[53],同时还将x染色体的差异甲基化巩固为一个长度为152.13 Mb的DMR,横跨染色体总长度的98%。
此外,DMRscaler提供了DMR的层次定义方法,DMR是通过合并从增加窗口大小构建的层的迭代过程构建的。对跨越x染色体的DMR进行更深入的分析表明,在DMR较低层的间隙中,在上层巩固的间隙中,已知可以逃脱x失活的基因显著富集,例如RPS4X[51,52],这与数据一致,这些数据表明,逃避x失活的区域在两性之间应该具有类似的表观遗传景观[52].这些浓缩结果说明了如何做到这一点DMRscaler,除了提供DMRs的直观表示外,还提供了DMRs的分层定义机制,可用于跨更大的表观基因组特征研究甲基化景观的结构。将这些直观缩放和定义DMR特征的层次映射的行为结合在一起DMRscaler用于在差异甲基化分析中实现比现有方法更大的灵活性和更有意义的结果解释。
最后,考虑到我们的主要兴趣是将这种方法用于罕见疾病研究中较小的样本量,我们进行了测试DMRscaler罕见染色质修饰剂综合征患者的数据集。在染色质修饰基因中含有已知致病突变的标本通常显示表观遗传特征的区域性变化,如DNA甲基化状态[9,10].我们的研究还探讨了由直接控制组蛋白修饰的基因的致病性突变引起的三种综合征。
arbolada - tham综合征(mim# 616268),也被称为KAT6A综合征,是由赖氨酸(K)乙酰转移酶突变引起的遗传综合征KAT6A以全面发育迟缓、智力残疾、语言迟缓或缺失为特征,并具有可变表达性的表型,如先天性心脏缺陷和胃肠道异常[7,58].KAT6A乙酰化组蛋白K3K9, H3K14和H3K23 [59,60,61],但arbolada - tham综合征中受影响的基因组区域尚未得到全面研究。以前,删除KAT6A在模式生物中已经鉴定出HOX基因,包括HOXB集群,作为KAT6A的规管指标[60,62].鉴定出的三种DMRs是由DMRscaler跨越多个基因的HOXB集群(无花果。3.),包含2个基因(HOXB3 HOXB4)发现于KAT6A敲除小鼠模型具有移位的表达域,导致轴向骨架的同源转化[60].突出单基因以外的差异甲基化程度的能力为发生在Arboleda-Tham综合征中的表观遗传变化提供了进一步的背景。
我们研究的一个局限性是病例通常比对照组年龄小。以前的研究已经确定了整体低甲基化与衰老有关[63,64].对于阿波莱达-谭综合征患者相对于对照组的高甲基化区域,我们在分析中不能排除这一潜在的混杂因素。
Weaver综合征和Sotos综合征是罕见的过度生长综合征,在没有测序的情况下很难区分。它们是由基因突变引起的EZH2[65,66),NSD1基因(55),分别。尽管他们共同的过度生长的临床表型,被确定为甲基化差异的基因组区域在这两种综合征之间很大程度上存在分歧,这表明了一个共同而复杂的表型的不同途径。对于韦弗综合症,DMRscaler鉴定了差异甲基化HOXA韦弗综合征相对于对照组的聚类基因。EZH2结合域的全基因组图谱显示EZH2结合HOXA集群(67]和EZH2在套细胞淋巴瘤中的过表达与细胞的高甲基化有关HOXA集群(68].关键的改进在于,与其强调单个基因[9]甲基化差异,DMRscaler能够通过突出这些特征的非随机空间关系作为一对DMRs分别跨越几个基因来证明遗传调控的模块化本质。
此外,DMRscaler在附近发现了一个新发现PCDHA, PCDHB和PCDHG与对照组相比,索托斯综合征患者的聚类普遍高甲基化。原钙粘蛋白家族基因在细胞-细胞粘附中起关键作用,并参与神经回路的复杂模式[56].这些相同的基因PCDHGA / B与对照组皮层组织相比,唐氏综合症患者皮层的聚类也被鉴定为高甲基化[69].从这些结果,我们可以假设的错误调节PCDH大脑发育中的群集可能有助于索托斯综合征的神经发育表型。
值得注意的是,我们观察到在Sotos和Weaver综合征这两种过度生长综合征之间IGFR2地区包括INS而且INS-IGFR2基因也有类似的甲基化差异。印版的正常调节能力丧失IGFR2与另一种过度生长综合征——贝克威-魏德曼综合征(BWS)有关[57].这种共同的差异是否在DNA甲基化的近端IGFR2位点是过度生长表型的表观遗传因子还是过度生长表型的结果值得进一步研究。
大多数真实世界的甲基化数据以简化表示平台的形式出现,该平台查询可能在基因调控中发挥作用的位点上的cpg,如已知的增强子和转录起始位点。虽然在数组中位点之间的距离是可变的,但我们的性染色体结果证明了我们的方法能够在这个简化的表示平台上调用大小显著不同的已建立的DMRs。全基因组亚硫酸氢盐测序(WGBS)为基于阵列的DNA甲基化查询技术提供了另一种选择,提供了更完整的基因组覆盖。虽然WGBS在技术和分析上具有挑战性,并且对于常规使用来说仍然非常昂贵,但DMRscaler对基于数组的数据是不依赖于平台和时间效率的,因此应该很容易移植到WGBS数据的分析中。由于基于阵列的DNA甲基化数据集的广泛可用性,特别是对于罕见疾病队列,我们决定在阵列数据上测试DMRscaler,并将WGBS数据的验证作为未来的方向。
结论
这里我们已经证明了这一点DMRscaler从单个启动子和CpG位点的局部尺度到代表染色体甲基化水平差异的DMR特征的描述是灵活而稳健的。所描述的所有分析都使用共享参数集运行DMRscaler,这对那些寻求探索这些高阶表观遗传特征的研究人员来说是非常有用的,同时也可以描述具有已知生物学意义的局部变化,例如与基因启动子重叠的甲基化变化。重要的是,DMRscaler作为原则的证明。重要的表观遗传特征存在于单个基因范围之外的想法并不新鲜,然而,现有的DNA甲基化分析方法并没有捕捉到这一知识。在这里DMRscaler证明这是可能的计算捕捉这种直觉,并在这样做揭示新颖的生物学见解。
数据和材料的可用性
模拟分析的代码可在https://github.com/leroybondhus/dmrscaler_simulation.用于实际数据分析的代码可在https://github.com/leroybondhus/dmrscaler_real_data.Arboleda-Tham综合征的数据集可以在GEO加入时找到:GSE210484https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE210484.Sotos和Weaver综合征的数据集可以在GEO登录:GSE74432中找到https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE74432.用于比较的方法可在DMRcate [46]:https://www.bioconductor.org/packages/release/bioc/html/DMRcate.html, bumphunter [44]:https://www.bioconductor.org/packages/release/bioc/html/bumphunter.html, comb-p [45]:https://github.com/brentp/combined-pvalues.用于数据可视化的包Hilbert, Curves [71]:https://www.bioconductor.org/packages/release/bioc/html/HilbertCurve.html,基因组范围图[72]:http://bioconductor.org/packages/release/bioc/html/Gviz.html、网络图[73]:https://cran.r-project.org/web/packages/networkD3/index.html.
可用性和需求
项目名称:DMRscaler。项目首页:https://github.com/leroybondhus/DMRscaler.操作系统:独立于平台。编程语言:R [70].其他要求:R版本4.1.0或更高。许可协议:麻省理工学院。对非学者使用的任何限制:无。
缩写
- TF:
-
转录因子
- 珠江三角洲:
-
多梳抑制域
- 泰德:
-
拓扑关联域
- DNAme:
-
DNA甲基化
- CpG:
-
胞嘧啶guinine二核苷酸
- VUS开头:
-
意义不明的变体
- DMR:
-
差异甲基化区
- 罗斯福:
-
错误发现率
- 质量控制:
-
质量控制
- TP:
-
真阳性
- FN:
-
假阴性
- 外交政策:
-
假阳性
- AUCPR:
-
精度召回曲线下面积
参考文献
潘涛D,德莱比E,梅里科D,等。自闭症谱系障碍中基因和细胞通路的收敛失调。胡姆·热内。2014;94:677-94。
Sun W, Poschmann J, Cruz-Herrera Del Rosario R, Parikshak NN, Hajan HS, Kumar V, Ramasamy R, Belgard TG, Elanggovan B, Wong CCY, Mill J, Geschwind DH, Prabhakar S.自闭症谱系障碍的组蛋白乙酰基全关联研究。Cell. 2016;167(1385-1397): e11。
洛佩兹AJ,伍德MA。核小体重构在神经发育和智力障碍中的作用。《神经科学》2015;9:100。
Iossifov I, O 'Roak BJ,桑德斯SJ, Ronemus M,克拉姆N,莱维D, Stessman哈,威瑟斯彭KT,韦弗斯L,帕特森KE,史密斯JD Paeper B, Nickerson哒,Dea J,侗族,冈萨雷斯勒,Mandell JD,鬃毛SM,默撒太,沙利文CA,沃克曼氏金融,Waqar Z,魏L, Willsey AJ, Yamrom B,李YH, Grabowska E, Dalkic E,王Z,标志着年代,安德鲁斯P, Leotta,肯德尔J,在野阵营,Rosenbaum J,马B,罗杰斯L, Troge J, Narzisi G, Yoon年代,沙茨MC,你们K, McCombie WR, Shendure J,为EE,国家兆瓦,从头编码突变对自闭症谱系障碍的贡献。大自然。2014;515:216-21。
马Zaidi,崔M, Wakimoto H, L,江J, Overton JD, Romano-Adesman, Bjornson RD,布莱巴特再保险公司布朗KK, Carriero新泽西,张YH, Deanfield J, DePalma年代,速度KA, Glessner J, Hakonarson H,意大利MJ, Kaltman JR Kaski J,金正日R,克莱恩JK,李T,莱比锡J,洛佩兹,鬃毛SM,米切尔勒,Newburger JW,当着M,同行我,波特G,罗伯茨AE, Sachidanandam R,桑德斯SJ, Seiden HS, MW,萨勃拉曼尼亚年代,Tikhonova IR,王W,沃伯顿D,白色的PS,威廉姆斯IA赵H,塞德曼詹,Brueckner M, Chung WK, Gelb BD, Goldmuntz E, Seidman CE, Lifton RP。先天性心脏病组蛋白修饰基因的新生突变。大自然。2013;498:220-3。
Watkins WS, Hernandez EJ, Wesolowski S, Bisgrove BW, Sunderland RT, Lin E, Lemmon G, Demarest BL, Miller TA, Bernstein D, Brueckner M, Chung WK, Gelb BD, Goldmuntz E, Newburger JW, Seidman CE, Shen Y, Yost HJ, Yandell M, tristini - firouzi M.先天性心脏病的隐性形式具有不同的遗传和表型景观。Nat Commun. 2019;10:4722。
布莱尔肯尼迪J,古迪D, E,钱德勒K, Joss年代,麦凯V,绿色,阿姆斯特朗R,李米,Kamien B, B料斗,谭泰,Yap P,鲜明的Z, Okamoto N, N宅一生,松本N,业务E,墨菲杰,麦考密克E, Hakonarson H,福尔克乔丹,李D,布莱克本P,克利E, Babovic-Vuksanovic D, Schelley年代,哈金斯L,康德,依B, B Cogne布拉德伯里K,威廉姆斯M, Patel C, Heussler H, Duff-Farrier C, Lakeman P, Scurr我Kini U,英语教学,Reijnders M, Schuurs-Hoeijmakers J, Wafik M, Blomhoff, Ruivenkamp卡尔,Nibbeling E, Dingemans AJM, Douine ED, Nelson SF, Arboleda VA, Newbury-Ecob R. KAT6A综合征:76例致病KAT6A变异患者的基因型-表型相关性Genet Med. 2019; 21:850-60。
张LX, Lemire G, Gonzaga-Jauregui C, Molidperee年代,Galaz-Montoya C,刘DS, Verloes, Shillington AG)和泉K,里特,凯娜B, Zackai E、D, Bhoj E, Tarpinian JM, Bedoukian E, Kukolich可,Innes, Ediae顾,索耶SL, Nair公里,Soumya PC, Subbaraman KR, Probst FJ, Bassetti是的,萨顿房车,吉布斯RA,布朗C,布恩点,河中沙洲IA,塔尔塔利亚米,费列罗GB, Niceta M, Dentici ML,无线电FC,克伦B井CF, Coubes C, Laquerriere, Aziza J,等级C, Nampoothiri年代,Mowat D,帕特尔女士,Bracho A, Cammarata-Scalisi F, Gezdirici A, fernando - jaen A, Hauser N, Zarate YA, Bosanko KA, Dieterich K, Carey JC, Chong JX, Nickerson DA, Bamshad MJ, Lee BH, Yang X-J, Lupski JR, Campeau PM。进一步描述KAT6B疾病的临床谱系和致病变异的等位基因系列。Genet Med. 2020; 22:1338-47。
Choufani S, Gibson WT, Turinsky AL, Chung BHY, Wang T, Garg K, Vitriolo A, Cohen ASA, Cyrus S, Goodman S, Chater-Diehl E, Brzezinski J, Brudno M, Ming LH, White SM, Lynch SA, Clericuzio C, Temple IK, Flinter F, McConnell V, Cushing T, Bird LM, Splitt M, Kerr B, Scherer SW, Machado J, Imagawa E, Okamoto N, Matsumoto N, Testa G, Iascone M, Tenconi R, Caluseriu O, Mendoza-Londono R, Chitayat D, Cytrynbaum C, Tatton-Brown K,Weksberg R. EZH2的DNA甲基化特征在功能上分类了三个PRC2复合体基因的序列变异。Am J Hum Genet, 2020; 106:596-610。
Choufani S, Cytrynbaum C, Chung BHY, Turinsky AL, Grafodatskaya D, Chen YA, Cohen ASA, Dupuis L, Butcher DT, Siu MT, Luk HM, Lo IFM, Lam STS, Caluseriu O, Stavropoulos DJ, Reardon W, Mendoza-Londono R, Brudno M, Gibson WT, Chitayat D, Weksberg R. NSD1突变产生全基因组DNA甲基化签名。Nat community . 2015;6:10207。
Wang H, Maurano MT, Qu H, Varley KE, Gertz J, Pauli F, Lee K, Canfield T, Weaver M, Sandstrom R, Thurman RE, Kaul R, Myers RM, Stamatoyannopoulos JA。CTCF占用的广泛可塑性与DNA甲基化有关。Genome res 2012; 22:1680-8。
Wiehle L, Thorn GJ, Raddatz G, Clarkson CT, Rippe K, Lyko F, Breiling A, Teif VB。胚胎干细胞中的DNA(去)甲基化控制ctcf依赖的染色质边界。基因组res 2019;29:75 - 61。
CpG甲基化对YY1和ETS蛋白与Surf-1和Surf-2基因双向启动子的结合有不同的影响。核酸决议1995;23:901-9。
梳子M,古德曼HM。CpG甲基化抑制脑啡肽前基因表达和转录因子AP-2的结合。核酸决议1990;18:3975-82。
Prendergast GC, Lawe D, Ziff EB。Myn (max的鼠同源物)与c-Myc的结合刺激甲基化敏感的DNA结合和ras共转化。细胞。1991;65:395 - 407。
包军,王永华,王永华,等。精子发生过程中组织-鱼精蛋白转变的表观遗传学调控。繁殖。2016;151:r55 - 70。
Brewer LR, Corzett M, Balhorn R.精蛋白诱导的相同DNA分子的冷凝和解冷凝。科学。1999;286:120-3。
Brown CJ, Hendrich BD, Rupert JL, Lafrenière RG, Y兴,Lawrence J, Willard HF。人类XIST基因:分析17 kb非活性x特异性RNA,包含保守重复序列,高度定位于细胞核内。细胞。1992;71:527-42。
克莱姆森CM,麦克尼尔JA,威拉德HF,劳伦斯JB。XIST RNA描绘了间期失活的X染色体:一种参与核/染色体结构的新型RNA的证据。中国生物医学工程学报(英文版),1996;
Disteche CM, Berletch JB。x染色体失活和逃逸。杨晓明。2015;26(4):529 - 529。
Pauler FM, Sloane MA, Huang R, Regha K, Koerner MV, Tamir I, Sommer A, Aszodi A, Jenuwein T, Barlow DP。H3K27me3在沉默基因和基因间区形成BLOCs,并在小鼠常染色体上指定组蛋白条带模式。Genome res 2009; 19:221-33。
Schwartz YB, Kahn TG, Nix DA, Li X-Y, Bourgon R, Biggin M, Pirrotta V.果蝇Polycomb靶点的全基因组分析。Nat Genet, 2006; 38:700-5。
布朗JL,孙M-A,卡西斯JA。在缺少Spps或Pho的情况下,H3K27me3结构域和Polycomb基团蛋白分布的全局变化。美国国家科学研究院。2018; 115: E1839-48。
邵波,李志强,李志强。基因组折叠成拓扑关联结构域的原理。2019年科学通报。https://doi.org/10.1126/sciadv.aaw1668.
王晓明,李志强,李志强,等。小鼠hox基因在体内的表观遗传时间控制。科学。2009;324:1320-3。
狄克逊,杜高金,任B.染色质结构域:染色体组织单位。Mol Cell, 2016; 62:668-80。
Magklara A, Yen A, Colquitt BM, Clowney EJ, Allen W, Markenscoff-Papadimitriou E, Evans ZA, Kheradpour P, Mountoufaris G, Carey C, Barnea G, Kellis M, Lomvardas S.单等位基因嗅觉受体表达的表观遗传特征。细胞。2011;145:555 - 70。
李志刚,李志刚,李志刚,李志刚,李志刚。嗅觉受体表达的表观遗传陷阱。细胞。2013;154:325-36。
辛斯海姆RL。胰脱氧核糖核酸酶II异构体二核苷酸的作用。生物化学学报。1955;215:579-83。
Mcghee JD, Ginder GD。鸡β-珠蛋白基因附近的特定DNA甲基化位点。大自然。1979;280:419-20。
Stadler MB, Murr R, Burger L, Ivanek R, Lienert F, Schöler A, van Nimwegen E, Wirbelauer C, Oakeley EJ, Gaidatzis D, Tiwari VK, Schübeler D. dna结合因子在远端调控区塑造小鼠甲基化。大自然。2011;480:490-5。
Viré E, Brenner C, Deplus R, Blanchon L, Fraga M, Didelot C, Morey L, Van Eynde A, Bernard D, Vanderwinden J-M, Bollen M, Esteller M, Di Croce L, de Launoit Y, Fuks F. Polycomb group蛋白EZH2直接控制DNA甲基化。大自然。2006;439:871-4。
李艳,郑辉,王强,周超,魏林,刘旭,张伟,张颖,杜震,王霞,谢伟。全基因组分析揭示Polycomb在DNA甲基化谷低甲基化中的作用。基因组生物学,2018;19:18。
福汀J-P,汉森KD。利用表观遗传数据中的长程相关性重建Hi-C所揭示的A/B区室。中国生物医学工程学报。2015;16:180。
Berman BP, Weisenberger DJ, Aman JF, Hinoue T, Ramjan Z, Liu Y, Noushmehr H, Lange CPE, van Dijk CM, Tollenaar RAEM, van Den Berg D, Laird PW。结直肠癌中局灶性DNA高甲基化和长程低甲基化区域与核层相关结构域一致。Nat Genet, 2011; 44:40-6。
Butcher DT, Cytrynbaum C, Turinsky AL, Siu MT, Inbar-Feigenberg M, Mendoza-Londono R, Chitayat D, Walker S, Machado J, Caluseriu O, Dupuis L, Grafodatskaya D, Reardon W, Gilbert-Dussardier B, Verloes A, Bilan F, Milunsky JM, Basran R, Papsin B, Stockley TL, Scherer SW, Choufani S, Brudno M, Weksberg R. CHARGE和Kabuki综合征:基因特异性DNA甲基化特征确定了连接这些临床重叠病症的表观遗传机制。胡姆·热内。2017;100:773-88。
张志刚,张志刚。依赖条件下多重测试错误发现率的控制。安统计2001;29:1165-88。
Mansell G, Gorrie-Stone TJ, Bao Y, Kumari M, Schalkwyk LS, Mill J, Hannon E. DNA甲基化研究的指导:来自Illumina EPIC阵列的统计见解。BMC基因组学,2019;20:366。
Aryee MJ, Jaffe AE, Corrada-Bravo H, Ladd-Acosta C, Feinberg AP, Hansen KD, Irizarry RA。Minfi:用于Infinium DNA甲基化微阵列分析的灵活而全面的Bioconductor包。生物信息学。2014;30:1363-9。
特里什TJ Jr,威森伯格DJ,范登伯格D,莱尔德PW,齐格蒙德KD。Illumina Infinium DNA甲基化珠阵列的低水平加工。核酸研究,2013;41:e90。
Fortin J-P, Labbe A, Lemire M, Zanke BW, Hudson TJ, Fertig EJ, Greenwood CM, Hansen KD。450k甲基化阵列数据的功能归一化提高了大型癌症研究的复制。中国生物工程学报。2014;15:503。
埃德加R,多姆拉切夫M,鞭策AE。基因表达综合:NCBI基因表达和杂交阵列数据存储库。核酸Res. 2002; 30:207-10。
Barrett T, Wilhite SE, Ledoux P, Evangelista C, Kim IF, Tomashevsky M, Marshall KA, Phillippy KH, Sherman PM, Holko M.其他:NCBI GEO:功能基因组数据集存档-更新。核酸研究,2012;41:D991-5。
Jaffe AE, Murakami P, Lee H, Leek JT, Fallin MD, Feinberg AP, Irizarry RA。表观遗传流行病学研究中鉴别差异甲基化区域的Bump hunting。国际流行病学杂志2012;41:200-9。
裴德生,杨晓东,杨晓东。用于组合、分析、分组和校正空间相关p值的软件。生物信息学。2012;28:2986-8。
Peters TJ, Buckley MJ, Statham AL, Pidsley R, Samaras K, V Lord R, Clark SJ, Molloy PL.人类基因组中差异甲基化区域的全新鉴定。表观遗传学染色质。2015;8:6。
李志刚,李志刚,李志刚。时间序列的查准性与查准性。arXiv [cs。LG] 2018。
王丽娟,陈晓霞,王磊,马丽丽,陈晓霞。基于监督方法的Illumina甲基化阵列差异甲基化区识别方法研究。生物信息简介。2018;00:1-12。
卡雷尔L,威拉德HF。x -失活谱揭示了女性x连锁基因表达的广泛变异性。大自然。2005;434:400-4。
Sharp AJ, Stathaki E, Migliavacca E, Brahmachary M, Montgomery SB, Dupre Y, Antonarakis SE。人类活跃和非活跃X染色体的DNA甲基化谱。基因组决议2011;21:1592-600。
Fisher EM, Beer-Romero P, Brown LG, Ridley A, McNeil JA, Lawrence JB, Willard HF, Bieber FR, Page DC。人类X和Y染色体上的同源核糖体蛋白基因:逃避X失活和特纳综合征的可能含义。细胞。1990;63:1205-18。
Balaton BP, Cotton AM, Brown CJ。从全基因组研究中推导x连锁基因一致失活状态。生物性别差异。2015;6:35。
刘j, M Morgan, K Hutchison, calhoud。性别对全基因组甲基化影响的研究。科学通报。2010;5:e10028。
Yousefi P, Huen K, Davé V, Barcellos L, Eskenazi B, Holland N.新生儿DNA甲基化的性别差异。BMC Genomics. 2015;16:911。
Douglas J, Hanks S, Temple IK, Davies S, Murray A, Upadhyaya M, Tomkins S, Hughes HE, Cole TRP, Rahman N. NSD1突变是Sotos综合征的主要原因,在一些Weaver综合征病例中发生,但在其他过度生长表型中很少见。胡姆。热内。2003;72:132-43。
Peek SL, Mah KM, Weiner JA。原钙粘蛋白对神经回路形成的调节。中国生物医学工程学报,2017;
Murrell A, Heeson S, Cooper WN, Douglas E, Apostolidou S, Moore GE, Maher ER, Reik W. IGF2基因变异与Beckwith-Wiedemann综合征之间的关联:基因型和表观基因型的相互作用。胡姆·摩尔·热内。2004;13:247-55。
Arboleda VA, Lee H, Dorrani N, Zadeh N, Willis M, Macmurdo CF, Manning MA, Kwan A, Hudgins L, Barthelemy F, Miceli MC, Quintero-Rivera F, Kantarci S, Strom SP, Deignan JL, Grody WW, Vilain E, Nelson SF。KAT6A(一种赖氨酸乙酰转移酶基因)的从头无义突变会导致包括小头畸形和全面发育迟缓在内的综合征。中国科学院学报(自然科学版),2015;
Mishima Y, Miyagi S, Saraya A, Negishi M, Endoh M, Endo TA, Toyoda T, Shinga J, Katsumoto T, Chiba T, Yamaguchi N, Kitabayashi I, Koseki H, Iwama A. Hbo1-Brd1/Brpf2复合体负责H3K14的整体乙酰化,并且是胎儿肝脏红细胞生成所必需的。血。2011;118:2443-53。
Voss AK, Collin C, Dixon MP, Thomas T. Moz和维甲酸协同调节H3K9乙酰化,hox基因表达和片段识别。开发技术。2009;17:674-86。
黄峰,Abmayr SM, Workman JL。KAT6乙酰转移酶的调控及其在细胞周期进展、干细胞维持和人类疾病中的作用分子生物学杂志,2016;36:1900-7。
Miller CT, Maves L, Kimmel CB。Moz调控斑马鱼hox表达及咽段同源性。发展。2004;131:2443 - 61。
肖福辉,王海涛,孔庆鹏。衰老过程中的动态DNA甲基化:年龄相关结果的“先知”。前热内。2019;10:107。
威尔逊VL,琼斯PA。DNA甲基化在衰老过程中会减少,但在不死细胞中不会。科学。1983;220:1055-7。
韦弗DD,格雷厄姆CB,托马斯IT,史密斯DW。一种新的过度生长综合征,骨骼加速成熟,不寻常的相,和喜趾。儿科杂志,1974;84:547-52。
Gibson WT, Hood RL, Zhan SH, Bulman DE, Fejes AP, Moore R, Mungall AJ, Eydoux P, Babul-Hirji R, An J, Marra MA, FORGE Canada Consortium, Chitayat D, Boycott KM, Weaver DD, Jones SJM: EZH2突变引起Weaver综合征。Am J Hum Genet. 2012;90:110 - 118。
Bracken AP, Dietrich N, Pasini D, Hansen KH, Helin K.多梳靶基因的全基因组定位揭示了它们在细胞命运转变中的作用。基因开发,2006;20:1123-36。
Kanduri M, Sander B, Ntoufa S, Papakonstantinou N, Sutton L-A, Stamatopoulos K, Kanduri C, Rosenquist R. EZH2在套细胞淋巴瘤HOX基因表观遗传沉默中的关键作用表观遗传学。2013;8:1280-8。
El Hajj N, Dittrich M, Böck J, Kraus TFJ, Nanda I, Müller T, Seidmann L, Tralau T, Galetzka D, Schneider E, Haaf T。表观遗传学。2016;11:563 - 78。
R核心团队:R:用于统计计算的语言和环境。2020.
顾z, Eils R, Schlesner M. HilbertCurve:用于基因组数据高分辨率可视化的R/Bioconductor包。生物信息学》2016。https://doi.org/10.1093/bioinformatics/btw161.
Hahne F . Ivanek R.利用Gviz和生物导体可视化基因组数据。方法Mol生物学,2016;1418:335-51。
Allaire JJ, Gandrud C, Russell K, Yetman CJ: networkD3: D3 javascript网络图从R. 2017。
确认
我们感谢Arboleda实验室成员对手稿的有益评论和反馈。我们感谢加州大学洛杉矶分校神经科学基因组学核心在处理我们的样本方面的支持和专业知识。
资金
本工作由DP5OD024579支持VA, T32HG002536 GATP(2020-2022)和T32LM012424 (2019-2020) B2K支持Leroy Bondhus。资助机构在这项研究的设计、数据的收集、分析和解释,或手稿的撰写中没有任何作用。
作者信息
作者及隶属关系
贡献
LB和VAA构思并设计了这项研究。LB编写代码,分析数据,制作图表。AW编写了仿真方法的代码,并对数据进行了重叠分析。VAA和LB对结果进行解读并撰写稿件。所有作者均已阅读并批准最终稿出版。
相应的作者
道德声明
伦理批准并同意参与
使用Arboleda-Tham综合征患者的活检样本需获得书面同意。本研究中使用的数据由加州大学洛杉矶分校机构审查委员会批准,研究编号为irb# 11-001087。
发表同意书
不适用。
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1
.补充图S1 - s25、补充表S1、S7、S8、S10-S12
附加文件2。表S2:
差异性别分析。
附加文件3。表S3:
Arboleda-Tham综合征DMRs
附加文件4。表S4:
韦弗综合征DMRs
附加文件5。表S5:
索托斯综合征DMRs
附加文件6表S6:
综合征重叠
附加文件7表S9:
仿真结果
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Bondhus, L., Wei, A. & Arboleda, va .DMRscaler:一种尺度感知的方法来识别跨越碱基对到多兆酶特征的差异DNA甲基化区域。BMC生物信息学23, 364(2022)。https://doi.org/10.1186/s12859-022-04899-1
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04899-1
关键字
- 罕见的疾病
- 表观基因组
- 尺度,DNA甲基化
- 染色质
- Arboleda-Tham综合症
- 索托斯综合症
- 韦弗综合症