摘要
背景
DNA测序成本的降低使我们对遗传变异的了解大大增加。虽然人口规模的项目为基因型-表型关系带来了重要的见解,但在大样本上进行全基因组测序的成本仍然令人望而却步。芯片基因型插入与阵列基因分型相结合是一种成本效益高、准确的常见和不常见变异基因分型方法。代入方法将分型变异的基因型与大型群体特异性参考面板进行比较,并利用连锁不平衡模式估计未分型变异的基因型。最精确的归算方法是基于Li-Stephens隐马尔可夫模型(HMM),该模型将每条染色体的序列视为来自参考面板的单倍型的马赛克。
结果
在这里,我们评估了基于邻近的hmm的准确性,其中每个未类型化的变体都是使用其周围小窗口(小至1厘米)的类型化变体来估算的。最近,基于位置的基因型归算方法被应用于基于机器学习的基因型归算方法中。我们在一组综合基准中评估了基于邻近的hmm参数如何影响输入精度,并表明基于邻近的hmm可以准确地输入常见和不常见的变量。
结论
结果表明,基于位置的基因型归算模型可以有效地用于基因型归算。我们确定的参数设置可以在未来的方法中使用,并且基于邻近的hmm可以用于重构和并行化新的插补方法。基于邻近的HMM实现的源代码可在此公开获得https://github.com/harmancilab/LoHaMMer.
背景
随着DNA测序成本的降低,可用的基因组序列数量也在快速增加[1,2,3.,4]。DNA测序也是RNA测序和ChIP-Sequencing等技术的基础步骤[5]。目前,有数百万个可用的基因组序列,预计还会有更多[6,7,8]。随着基因组数据在临床和转化研究中的应用越来越广泛[9,10],基因数据大小在许多不同的情况下都是可用的,甚至包括来自普通人群的公民科学家[11]。遗传数据广泛地储存在许多地方(包括个人电脑甚至电话),它也很好地进入了娱乐遗传学领域。12]。这是通过广泛绘制种群间的遗传差异图和有效的方法来实现的,这些方法可以筛选大量的数据库来寻找亲属[13]。这些都是由人口规模的项目,如UKBiobank [14]。
遗传数据的主要用途之一是使用全基因组关联研究(GWAS或GWA研究)进行基因型-表型关联[15,16,17,18]。为此,产生了一个大队列,并通过测序对个体进行基因分型。接下来,从所有个体中测量感兴趣的表型(智商、身高、体重指数、血糖水平等)。最后,对所有变异的测量基因型进行了与GWA研究的关联测试,发现大多数变异位于蛋白质编码外显子的基因间区域。因此,有必要使用全基因组测序(WGS)等方法对整个基因组进行基因分型,以确保能够准确检测到因果变异。因果变异是影响编码或非编码元件(例如增强子)并机械地影响疾病表现或导致病例和对照之间显着表型差异的变异。GWA研究通常检测到一种处于连锁不平衡(LD)的变异[19,20.而最可能与这一特征相关的真正的因果变异必须通过精细映射程序加以剖析。然而,这并不具有成本效益,因为必须对大样本进行全基因组测序[21]。为了解决这个问题,基因分型阵列被用于基因分型并降低成本[22]。基因分型阵列被设计为仅对基因组中稀疏的一组变体进行基因分型。然后将这些变体输入到计算机基因型插入算法中[23,24],它推导并“填充”未基因分型(或简称为未分型)的变异。代入算法背后的主要思想是利用整个基因组已知的单倍型结构,并使用在单倍型水平上相关的有型变异的基因型来估计未分型变异的基因型[25]。单倍型结构的出现是因为等位基因在几代之间通过同源染色体之间重组热点的有限数量的交叉遗传[26]。这导致父母和孩子之间作为一个单一的单位遗传了大量的单倍型。尽管保存块的长度(identity-by-descent段[27[])随着关系距离的增加而减少,即使个体之间相隔20-25代,仍然可以检测到[28,29]。代入算法的重点是利用种群中不相关个体之间共享的保守单倍型(即频繁单倍型)。代入方法也用于代入由RNA测序和全外显子组测序鉴定的变异,并用于关联研究中变异的精细定位。
目前最先进的imputation方法,如BEAGLE, Minimac和IMPUTE套件使用隐马尔可夫模型(HMM) [30.,31,32,33,34基于Li和Stephens开发的方法[35,36,37,38,39,40]。HMM将每个单倍型视为一种“状态”,并分析所有通过这些状态的“路径”的概率,这些“路径”产生由该数组类型的等位基因[36]。这样,基于hmm的方法可以使用Li-Stephens单倍型模型施加的概率模型为输入的基因型分配概率。HMM将类型变体和参考面板作为输入,并将参考面板上存在但未被基因分型阵列分型的所有变体归为输入。虽然HMM模型提供了良好的插入准确性,但它们可能在插入罕见变异时失败,因为这些变异至少在罕见的单倍型上表示[41]。然而,随着参考面板尺寸的增加,可以更准确地预测罕见的变异[42]。
在这里,我们将重点放在Li-Stephens基于HMM的输入模型上,并评估“基于邻近的HMM”的性能,即HMM仅在未类化的变体周围的一小段变体上评估路径。虽然有几种方法已经测试了最先进方法的不同参数化,但我们实现了基于邻近的HMM方法,以完全控制参数如何影响imputation。评估归算参数是合理的,因为先前已经表明基因组具有不同的“归算性”[43],也就是说,一些变异更容易推导,而另一些则因为它们周围复杂的单倍型结构而不太容易推导。因此,详细研究基于邻域模型的参数对于调整基于深度学习和安全插值等新的资源密集型方法的参数是有用的。在本研究中,我们重点研究了不同参数对基于附近的插值模型的影响,以及它们如何影响插值的准确性。虽然我们没有明确的目标是为基因组的不同区域生成最佳的基于邻近的代入参数,但我们提供的证据表明,即使没有对基因组进行更广泛的优化,具有相当约束的参数集的基于邻近的模型也可以提供良好的代入精度。这些参数可以作为起点,同时搜索与准确性或其他应用特定考虑因素(例如安全性与性能)相关的特定于邻近的输入参数。
基于位置的方法已经在不同的场景中使用,例如线性输入模型和基于深度学习的输入模型[44,45,46],其中对未类型化变量附近的类型化变量执行输入。此外,IMPUTE和BEAGLE还利用了一个长达40厘米的滑动窗口来节省计算时间。该参数对输入精度的影响没有得到广泛的评估,特别是对于更短的窗口长度。我们通过前向后向算法实现了每个位置的后验概率估计(我们将其称为“前向后向”或“FB”方法)。我们还实现了最大似然HMM路径的推断(称为Viterbi方法),它代表了产生分型变异基因型的参考单倍型的最可能拼接。在这些方法上,我们分析了窗口的大小、目标在窗口内的位置以及窗口上类型变体的数量。我们还通过分析不同的参考种群,报道了种群特异性归算的效果。值得注意的是,我们关注的是分阶段的基因型插入问题,也就是说,我们假设基因型是分阶段的。这是一个合理的假设,因为预相位已被证明可以大大改善估算方法的时间复杂度,同时带来非常小的性能损失[39]。
基于位置的方法的一个主要优点是,它们可以在计算需求方面受到约束,而不需要将整个基因组加载到内存中,也不需要在整个染色体或非常长的片段上运行HMM推理方法。通过这种方式,云上的imputation算法架构可以相应地构建,例如,通过在基因组的不同部分使用不同的模型。在另一方面,最近发展具有隐私意识的基因型插补方法的努力,利用基于邻近的模型进行插补,同时保持基因型数据的机密性[47,48]。这些方法可以潜在地作为基因型插入服务器(如Michigan imputation Server)的替代方法[49],它提供了一种服务。在这些服务器中,基因型数据以普通形式处理,任何人都可以访问基因型数据,这使得当数据来自脆弱或代表性不足的人群时,使用这些服务相当令人担忧。随着遗传隐私日益成为公众讨论的重要话题,有必要开发更有效的归算方法,利用设计隐私原则构建归算服务。虽然已经有人(包括我们的团队)尝试构建安全的插入工具[47,48,50,51]使用可信执行方法和同态加密技术[52],这些方法受计算需求的限制[53]。因此,我们的研究可以告诉这些方法必须考虑和评估的局部参数,以减少计算需求和保持估算精度,同时为遗传数据提供隐私和机密性。基于社区的hmm的实现名为LoHaMMer,可以公开下载https://github.com/harmancilab/LoHaMMer.
结果
简要介绍了基于hmm的插补技术、参数和评估方法。最后给出了插值精度评价。
基于区域的hmm概述
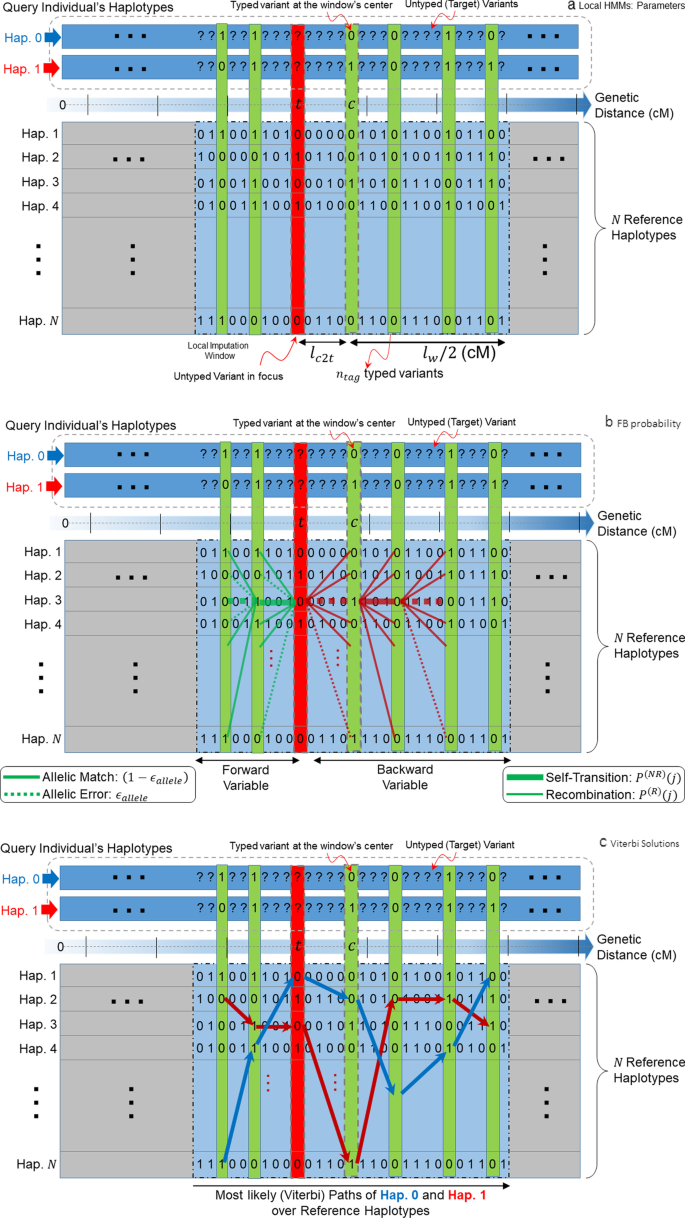
基因型插补总结如图所示。1.基因型插入过程以变异阶段基因型矩阵为输入,\ \ ({G} _{米乘以V} \),个人。在我们评估分阶段代入过程时,\ (G \)使用Eagle等分相算法进行预分相[49(图。1一个)。\ ({G} _ {i, j} \)的阶段性基因型\ ({j} ^ {\ mathrm {th}} \)the的变体我\ ({}^ {\ mathrm {th}} \)个人,也就是说,\ ({G} _ {i, j} ^ {(h)} \ \左\ {\ mathrm {0,1}, \ mathrm {\ varnothing} \右\},le我1 \ \勒米,le V le j 1 \ \ \),在那里\ \ (h)表示基因型的父/母副本,即。\ (h \ \ {\ mathrm {0,1} \} \).\ ({G} _ {i, j} ^ {(h)} = \ mathrm {\ varnothing} \)指示缺失的基因型,该基因型将使用参考面板进行输入。我们用表示未类型化变量的索引集\ ({j} _ {\ varnothing} = \左\ {j | {G} _ {i, j} ^ {(h)} = \ varnothing \右\}\).代入时也取含有参考基因型的基质\ ({H} _ {N \ * V} \)的\ (N \)单倍型相同\ \ (V)的列对应的变体\ (G \).类似于\ (G \),\ ({H} _ {i, j} \ \左\ {\ mathrm{0,1} \右\},le我1 \ \ N, le V le j 1 \ \ \).
一个本地输入设置的说明。查询个体亲本单倍型副本(“Hap”)。0”和“好运。”1 ")显示在顶部的两个矩形中,它们是字符串{0,1}。0和1分别表示对应变异的参考等位基因和备用等位基因。未类型化的变体用“?”表示。来表明他们的等位基因是未知的。遗传距离(以厘米器官为单位)用蓝色箭头表示,用于跟踪中心位置(用' '表示)。c’)和焦点中的目标未类型化变体(用“t”)。参考单倍型显示在下面的框中,其中每一行对应一个单倍型。给定半径的局部窗口\ ({l} _ {w} \)窗口用虚线矩形表示,其中心显示在遗传位置\ (c \)对于位置上的目标变体\ \ (t).已类型化的变体用绿色矩形表示,未类型化的目标用红色矩形表示,其在查询单倍型上的等位基因用问号表示。b“单倍型0”等位基因序列发射的正向和反向变量说明。对于未类型化变体的第三个单倍型,传入路径(前向变量)用绿线表示。每条绿线源于一个单倍型(即状态),表明相应单倍型上的一个等位基因的发射。虚线表示个体单倍型(Hap. 0)和参考单倍型之间的等位基因不匹配。这些路径受到等位基因错误概率(\({\ε}_{等位基因}\))在正向和反向变量。还显示了更左边的不同位置的传入路径,它描述了在隐马尔可夫模型中评估的路径数量的指数增长。目标变体右侧的传出转换用红线表示。与非自转换相比,自转换用较粗的线条表示,以描述它们具有更高的发生概率,即维持单倍型的概率高于产生重组事件的概率。c两条维特比路径显示了与查询个体“Hap”的单倍型对应的单倍型转换。0”和“好运。”1”
Li-Stephens Markov模型
我们的评估使用由标准Li-Stephens模型定义的Markov模型[35],其中每个查询个体的单倍型被建模为“参考单倍型的马赛克”,以便参考单倍型的片段(单倍型上连续的变异等位基因)彼此连接。该模型描述了通过参考单倍型的可能“路径”的概率分布。1b, c)。在该模型中,单倍型之间的转换和单倍型上的错误是概率性的。从最简单的意义上说,单倍型转换和等位基因错误的最小数量可以被认为是描述查询单倍型的最可能路径。基本思路是确定路径上的类型化变异等位基因,并估计未类型化变异等位基因的边际概率:
在这个模型中,参考面板的单倍型对应于Li-Stephens Markov模型的状态。每一种状态(单倍型)都在一个变异位置发出一个等位基因\(1\ j\ V\).此外,变体的状态之间的转换(即单倍型之间的切换)\ (j \)是否只依赖于变异指数之间的遗传距离\ (j \)和\左(j + 1 \右)(\ \).遗传距离衡量的是这两个变异之间发生重组的概率。在马尔可夫模型中,重组对应于状态转换,即状态(即单倍型)转换到新状态。然而,在减数分裂过程中,当同源染色体交叉时,重组发生。重组率的变化取决于基因组上的位置,也就是说,基因组的某些部分比其他部分更容易发生重组。因此,沿着基因组的重组事件的流行程度是根据遗传距离来量化的,遗传距离是以厘米器官(cM)来测量的,cM是两个位点之间重组概率的度量。给定两个变量左(j - 1 \ \ (\) \)和\ (j \),重组概率建模为:
在哪里\ ({P} _ {j} ^{\左(R \右)}\)表示存在重组事件(即马尔可夫链保持在同一状态)的概率,R \ ({} _ {j} \)表示变异在索引处的插值累积遗传距离\ (j \)(确切定义见方法),\({\δR} _ {j} \)表示各变异间的遗传距离左(j - 1 \ \ (\) \)和\ (j \),\ ({N} _ {e} \)表示有效种群大小。值得注意的是,重组的概率只取决于变异的位置,而不是实际的单倍型。这被广泛应用于基于hmm的插值方法中,以减少计算成本。重组不发生的概率可以由\ ({P} _ {j} ^{\左(R \右)}\):
其中所有重组事件都被考虑并从1和\ ({P} _ {j} ^{\左(NR \右)}\)指示索引处的变体之间没有重组\ (\) (j - 1)和\ (j \).由上面的方程得到\ ({P} _ {j} ^{\左(R \右)}\)群体规模越大意味着重组的可能性越大,即有效群体规模越大,随着单倍型(或状态)之间切换的概率增加,重组模式也就越复杂。考虑到查询个体的阶段性基因型,\ ({G} _ {i, j} ^{左(h \) \} \)参考单倍型数据,\ ({H} _ {, j} \)HMM是基于这些方程,利用跃迁概率和发射概率来定义的。跃迁和发射概率表示为
在哪里\({\tau}_{j}\左(b\到a\右)\)表示从单倍型转移的概率\ (b \)来\ \ ()在变指标下\ (j \)从前面的变体at index左(j - 1 \ \ (\) \)和\ ({e} _ {j} \离开({G} _ {i, j} ^{\左(h \右)},一个\)\)表示该等位基因的发射概率\ ({G} _ {i, j} ^{左(h \) \} \)从\ ({} ^ {\ mathrm {th}} \)单体型。发射概率取决于查询个体的等位基因;如果等位基因\ ({} ^ {\ mathrm {th}} \)单倍型匹配查询个体的等位基因,分配高的发射概率,否则分配等位基因错误概率;\({\ε}_{等位基因}\),赋值为发射概率。
利用上述方程和Li-Stephens模型,我们使用两种方法来推断每个类型变体的单倍型状态。
边际状态(单倍型)概率推断
首先,对每个单倍型的后验概率进行估计,并对未分型变异的等位基因进行基于前向后边缘化的估计(图2)。1b).为此,我们使用正向倒向算法[54],这是一种众所周知的动态规划算法,用于有效地计算HMM每一步的状态概率
在哪里\ ({} _ {i, j} ^{左(h \) \} \)表示变量索引下HMM的状态(单倍型)\ (j \)对个人我\ \ ()亲本\ \ (h)(\ (h \ \ {\ mathrm {0,1} \} \)),\ ({G} _{我,[1]}^ {(h)} \)中变异的等位基因序列\ ([1] \)对个人我\ \ ()亲本\ \ (h),\ (P \离开({}_ {i, j} ^ = {(h)}, {G} _{我,[1]}^ {(h)} \) \)是前向变量,它表示发射等位基因序列的概率吗\ ({G} _{我,[1]}^ {(h)} \)假设HMM处于状态\ \ ()在可变位置\ (j \).后向变量的定义类似于后向的等位基因序列的其余部分j变体,即,G \({} _{我[j + 1, V]} ^{\左(h \右)}\).前向和后向变量使用有效的递归关系计算(参见方法)[55,56]。式(6)中的关系由的条件独立性推导而来G \({} _{我[j + 1, V]} ^{\左(h \右)}\)和G \({} _{我[j + 1, V]} ^{\左(h \右)}\),鉴于\ ({} _ {i, j} ^ {(h)} = \).前后变量在每个变量位置\ (j \)对于每个州\ \ (),我们可以估计每个等位基因在每个未分型位置的后验概率:
未分型的变异等位基因\ \ (t)的概率是通过对各州的边际化来估计的\ \ ()对应的单倍型有一个等位基因\ \ (t).正如我们下面所描述的,我们评估了两种不同的单倍型边缘化方法。
最大似然镶嵌单倍型(维特比)
向前-向后算法侧重于在特定变体上边缘化,而Viterbi算法旨在预测沿单倍型最可能的“路径”(图2)。1C)因此一个单倍型是参考单倍型的“马赛克”。从概念上讲,前向-后向算法计算每个变体上每个单倍型的边际概率,而Viterbi分析所有变体的总体概率,以确定最优的镶嵌单倍型。为了计算最可能的单倍型,以单倍型的等位基因序列为条件的状态序列的总体概率最大化。我们把它记为
在哪里\ (P \离开({}_ {[1 V]}, {G} _{我[1 V]} ^{左(h \) \} \) \)表示状态序列的联合概率\ ({} _ {[1 V]} \)以及各变异对应的等位基因序列\(左\ [1,V \] \)为我由状态序列发出的个体。\({\打翻{\ lower0.5em \ hbox{$ \粉碎{\ scriptscriptstyle \微笑}$}}{年代}}_{我[1 V]} ^ {(h)} \)表示使的概率最大化的状态序列我个体的单倍型\ \ (h)(无花果。1c).这个状态序列代表最有可能产生变异等位基因的马赛克单倍型\ ({G} _{我[1 V]} ^{\左(h \右)}= ({G} _{1}我^ {(h)}, {G} _{我2}^ {(h)}, \ ldots {G} _{我,V} ^ {(h)}) \).状态序列可以使用动态规划算法进行推断,即Viterbi算法[57],有效地识别最大似然状态序列,类似于前向算法。
在使用Viterbi算法计算出最可能的状态序列后,我们使用该状态序列上的等位基因分配未类型化变体的等位基因:
在这里,\({\打翻{\ lower0.5em \ hbox{$ \粉碎{\ scriptscriptstyle \微笑}$}}{G}} _ {i, j} ^{左(h \) \} \)表示无型变异最可能的单倍型上的等位基因\ (j \)这是分配给个人的我\ \ ()的单体型\ \ (h).维特比算法不会立即为每个输入的等位基因分配一个分数。我们汇总附近的信息,为输入的等位基因分配一个分数。
推算的局部性参数
我们评估了改变参数对基因型插入准确性的影响。向前向后和基于viterbi的输入算法依次分析变量,同时跟踪每个状态的分数和概率。它们可以使用每条染色体上的所有变异进行,因为LD信息通常局限于单个染色体,而染色体间LD信息虽然可检测,但非常微弱[58]。这些都超出了我们评估的归算方法的范围。采用整条染色体进行代入,使算法能够整合染色体上所有位置的连锁信息。另一方面,在输入无型变异时,连锁信息往往会迅速减少,例如,按血统划分的片段长度(保守单倍型的长度)在代之间迅速减少(25代分离平均有2 cM的保守性)。28])。这些信息可以被整合到向前向后(图2)。1b)和基于viterbi的(图2)。1C)通过滑动窗口进行估算,其中局部窗口外的变量不用于估算。这有助于减少计算需求。例如,BEAGLE使用一个大的滑动窗口(长度为30 cMs),并合并连续的窗口来推断向前和向后变量。在我们的研究中,我们仅在未类型化变量周围的局部窗口上运行前向向后和Viterbi算法,并使用这些“基于邻近的hmm”来估算未类型化变量。例如,如果我们使用一个长度为0.5厘米的局部窗口,那么最可能的状态序列只会在分配的局部附近计算
在哪里R \ ({} _ {k} \)表示变异的插值累积遗传距离\ (k \)(见方法)。在(10),状态序列;\({\打翻{\ lower0.5em \ hbox{$ \粉碎{\ scriptscriptstyle \微笑}$}}{年代}}_{我,\ [k, l \]} ^{左(h \) \} \),仅限于变指标\ ([k, l] \)遗传距离约为0.5 cm。向前向后计算同样局限于基于遗传距离截止点的局部窗口。
我们测试了不同的局部窗口长度,并评估了窗口长度对等位基因插入精度的影响。我们利用一个长度从0.1到1 cMs的滑动窗口,并计算输入精度(参见指标)。另一个重要因素是在局部窗口内定位未类型化的目标变体(图2)。1a).如果将未类型化的变体以局部窗口为中心,预计可以更准确地集成LD信息。然而,“目标到中心距离”对插补精度的影响程度尚不清楚。对于每个未类型化的目标变异,我们首先使用Viterbi和前向-后向方法确定将用于满足局部窗口长度和目标到中心距离标准的类型化变异,并选择种群大小和等位基因概率分配程序。
评估设置和度量
我们使用来自1000基因组计划第三阶段的基因型数据[59]。我们对19、20和22号染色体上的变异进行了广泛的评估,并排除了多等位snv和索引。在这些数据中,22号染色体用于评估不同的参数组合。为了减少参数组合的计算需求,我们将重点放在chr22:25,000,000-35,000,000区域。在评估中,我们随机选择1000个个体作为阶段性参考组,200个个体(已知基因型)进行估计评估。在对参数进行评估后,我们选择了最优的参数集,并在染色体19和20上进行了验证。为了定义分型(标签)变异,我们在Illumina Duo 1 M基因分型平台上提取了基因分型变异的位置[60就像我们之前的研究中使用的一样[47]。这使我们能够在实际的测试用例上进行评估,因为Illumina的阵列被用于几个大型项目,包括HAPMAP项目[60]。我们使用所有映射到与Illumina Duo平台重叠位置的变体作为类型化变体,剩余的变体被分配为未类型化变体。提取变异后,我们使用EAGLE2进行分型[49]。将分阶段类型的变量以不同的参数输入到LoHaMMer中进行imputation。(1)基因型一致性(所有和非参考基因型),(2)基于输入概率的精确召回曲线。我们将实现的基于位置的hmm与BEAGLE进行了比较,BEAGLE被用作基准方法进行插值。根据次要等位基因频率(MAF)的范围和染色体位置,对各变异的归算精度进行分类。
变化局域参数下的插值精度评价
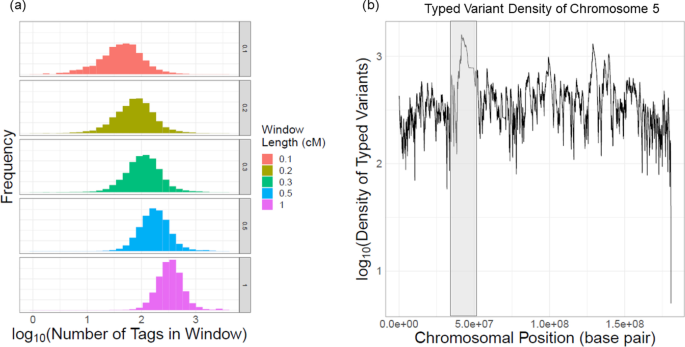
为了评估参数变化时的输入精度,将变量分为“常见”(MAF > 0.05)和“不常见”(MAF < 0.05)变量。我们测试了4个不同参数的影响,包括窗口长度(\ ({l} _ {w} \))、目标到中心距离(\ ({l} _ {c2t} \)),以及窗口中类型变体的数量(\ ({n} _{标签}\)).在这里,我们没有计算所有参数组合,而是为每个参数选择一个范围,并在保持其他参数不变的情况下评估一个参数的影响。我们使用\ \离开({l} _ {w}, {N} _ {e}, {1} _ {c2t}, {N} _{标签}\右)= ({\ mathrm {0.3, 10}} ^ {4}, \ mathrm {0.05, 10000}) \)采用默认参数值。而\ ({n} _{标签}\)设置为10,000的大值,则本地类型变体的数量取决于本地窗口长度(\ ({l} _ {w} \)).数字2A表示不同窗口长度参数下类型变体数量的分布。最长测试窗口长度为1cm(图底部面板)。2A),我们观察到大多数Windows包含少于1000种类型的变体。我们还观察到,沿染色体有大致一致的类型变异覆盖范围(图2)。2b、附加文件1:图S1)。
本地窗口长度(\ ({\ mathbf {l}} _ {\ mathbf {w}}) \)
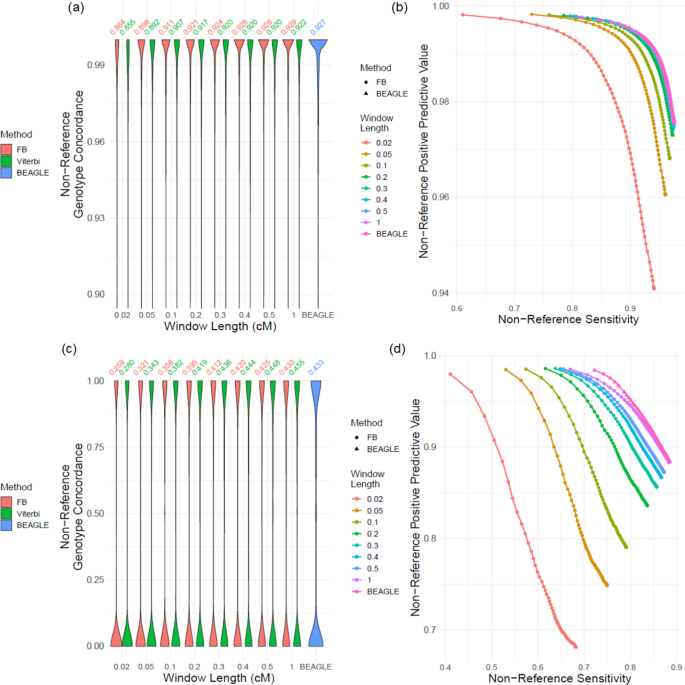
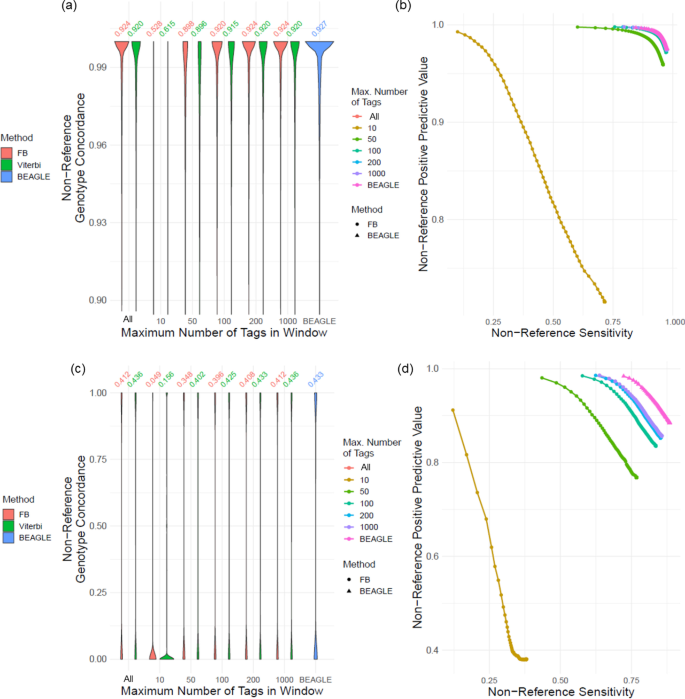
我们首先评估了局部窗口长度对输入精度的影响。我们使用了局部窗口长度\ ({l} _ {w} \ \左\ {0.02,0.05,0.1,0.2,0.3,0.4,0.5,1 \ \}\)厘米。数字3.A、b为不同窗长下常见变异的非参考基因型一致性分布和非参考基因型的查准率曲线。正如预期的那样,精确度随着窗口长度的增加而增加。对于窗长大于0.3 cm的情况,我们观察到向前向后和BEAGLE比较时,非参考基因型一致性增加了约0.5%(图2)。3.a、附加文件1:图S2a)。对于不常见的变体,大于0.5 cm的窗长表现出与BEAGLE非常相似的行为,差异为0.8%(图2)。3.c).不常见变异的精确召回曲线显示,非参考基因型在0.3 cm以上的曲线非常接近(图2)。3.d).这些结果表明\ ({l} _ {w} > 0.3 \)cM是最小窗口长度,具有与BEAGLE相当的精度。对于不常见的变异,我们观察到一致性在高或低的准确性制度(图2)。3.c) BEAGLE和LoHaMMer。所有不常见变异基因型的PR曲线显示出相当稳定的准确性增加模式(附加文件)1:图S2b),对于\ ({l} _ {w} > 0.3 \).
靶心距离(\ ({\ mathbf {l}} _ {\ mathbf {c} 2 \ mathbf {t}} \))
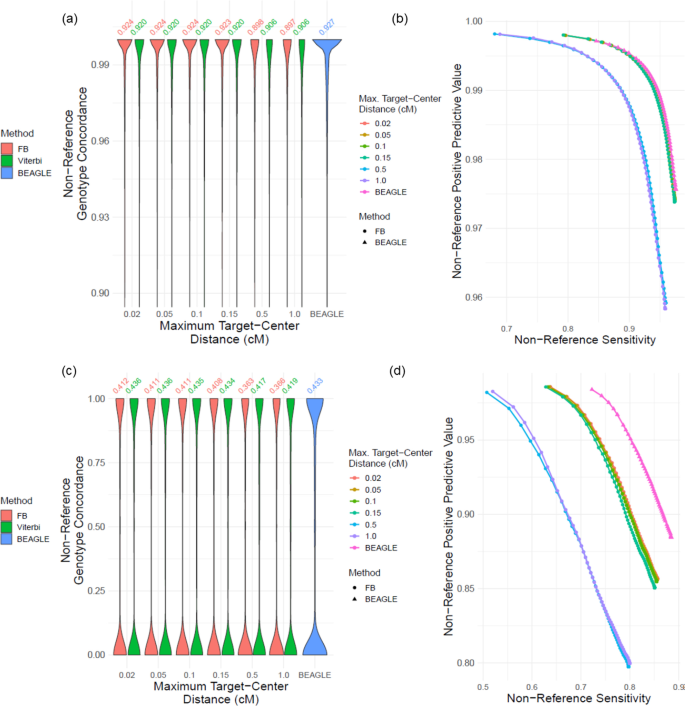
目标变体的定位,l \ ({} _ {\ mathrm {c} 2 \ mathrm {t}} \)(目标中心距离),在插补窗口中是影响插补精度的另一个参数(图2)。1a).我们通过增加目标中心距离值来测试精度的估算;l \ ({} _ {\ mathrm {c} 2 \ mathrm {t}} \中\ {0.02,0.05,0.1,0.15 \}\)厘米。我们使用遗传距离作为这些参数的度量单位,因为它是最自然的选择(方法)。常见变异的非参考基因型一致性显示为不同的中心-目标距离值,表明目标-中心距离的可见影响(图2)。4a, b)。我们观察到,随着l \ ({} _ {\ mathrm {c} 2 \ mathrm {t}} \)增加。这表明来自无型变异两侧的单倍型和LD信息应该是平衡的。为l \ ({} _ {\ mathrm {c} 2 \ mathrm {t}} < 0.15 \)cM,我们观察到基于局部窗口的imputation提供了相当的精度。对于不常见的变异,我们在非参考基因型PR曲线方面观察到类似的模式(图2)。4c, d)。非参考一致性和所有基因型PR曲线也发现了类似的结果1:图S3a)和不常见的变体(附加文件1:图S3b)。
窗口中类型变体的最大数目(\ ({\ mathbf {n}} _ {\ mathbf {t} \ mathbf {} \ mathbf {g}} \))
我们测试的下一组参数是用于输入的类型变量的数量。为此,我们对每个窗口中的类型化变体进行了抽样,使得类型化变体的数量受到类型化变体的最大数量的限制。为此,我们评估了变化的影响\({n}_{tag}\in \{10,50,100,200,1000 \)}。包含小于的窗口中的类型化变体\ ({n} _{标签}\)类型化的变体按原样使用。数字5a、b分别为非参考基因型一致性和PR曲线。为\ ({n} _{标签}\)大于100个变量时,我们观察到,对于常见的变量,准确率会略有提高(附加文件)1:图S4a)。对于不常见的变异,我们观察到基因型的准确性趋于平缓\ ({n} _{标签}= 200 \), PR曲线表现出相似的模式,尽管所有参数选择的imputation精度都相当低(图2)。5c、d、附加文件1: S4b)。对于不常见的变量,在窗口中使用所有类型的变量更合适,因为该参数强烈影响准确性。
参考样本和查询样本之间的祖先不匹配
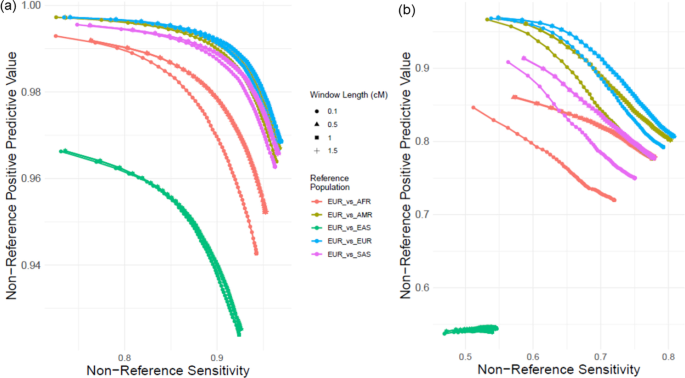
接下来,我们测试了查询个体和参考面板中个体的遗传祖先之间的不匹配如何影响准确性。为此,我们使用欧洲血统的个体(超级种群EUR)作为查询个体。我们使用其他4个超级种群作为参考组:美洲(AMR)、非洲(AFR)、东亚(EAS)和南亚(SAS)。作为基线,我们也使用了欧洲面板作为匹配的参考面板。对于5个查询参考面板对(包括EUR作为参考)中的每一个,我们使用不同的窗口长度参数对22号染色体上常见和不常见的基准变异进行了代入\ ({l} _ {w} = \左\ {0.1 \ mathrm{0.5, 1}, 1.5 \右\}\).数字6显示了不同参考种群和窗长的非参考一致性。正如预期的那样,匹配EUR参考种群的准确性最高,其次是AMR种群,已知AMR种群含有大量EUR混合物[61]。EAS参考面板的插补精度最低。虽然增加窗口长度增加了所有参考种群的准确性,但我们观察到AFR参考种群获得了最高的改进。最近欧洲人和非洲人后裔的混合可能会支持这一点[62]。总之,我们的结果表明,当使用特定人群的参考面板时,参数可能需要重新参数化。
时间和内存要求
我们测试了基于邻近的hmm的时间和内存需求如何随窗口长度参数的增加而增加。我们使用的窗口长度测量了forward-backward和Viterbi方法的时间和内存使用情况\(\{0.02, 0.05, 0.1, 0.2, 0.3, 0.4 0.5, 1.0\}\)厘摩。时间(图2)7a)和内存使用情况(图2)。7两种方法的B)相对于窗长线性增加。一般来说,Viterbi比正向倒推法需要更多的时间。这源于这样一个事实,即Viterbi方法利用了许多低效的分支操作,而这些分支操作是在()中的Viterbi递归中识别最佳路径所必需的。24).我们要强调的是,我们目前的实现是为了便于基准测试而优化的。例如,我们从头开始计算每个窗口的向前和向后变量,这是没有必要的,因为大量的窗口重叠,变量可以被重用。我们在讨论部分讨论了许多优化方法。
19号和20号染色体的准确性
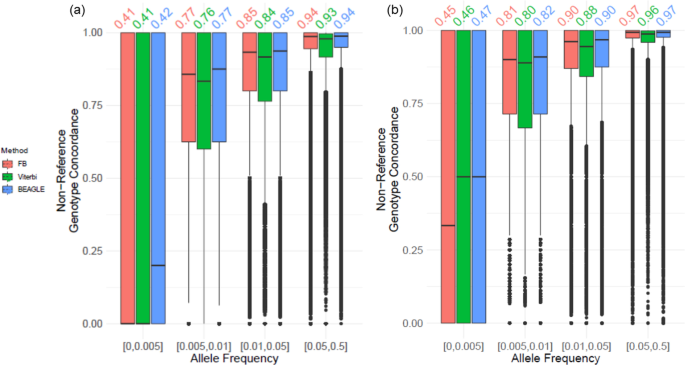
为了在单独的数据集上验证和比较基于邻近度的HMM参数的准确性,我们测试了19和20号染色体变异的参数。我们在Illumina Duo阵列平台上提取了19和20号染色体上的分型变异。在此之后,我们提取了19号染色体27,403型变异中的24,333型和20号染色体28,319型变异中的26,405型。其余的变异(chr19上的768,292个变异和chr20上的742,370个变异)被用作由基于邻近的HMM和BEAGLE输入的未类型变异。我们通过将变体分为4个不同的MAF范围来对MAF进行分类:1)\(\ MAF}\in \left[0,0.005 \right]\)(非常罕见),2)\(\ MAF}\in \left[0.005, 0.01\right]\)(罕见),3)\(\ MAF}\in \left[0.01, 0.05\right]\)(少见),4)\(\ MAF}\in \left[0.05, 0.5\right]\)(共同)。在输入未分型的变异之前,使用Eagle2对基因型进行分期[49]。我们使用参数\ \离开({l} _ {w}, {N} _ {e}, {1} _ {c2t}, {N} _{标签}\右)= ({\ mathrm {0.5, 10}} ^ {3}, 0.02, 1000) \)用于验证准确性。数字8为19号染色体变异的非参考基因型一致性分布(图2)。8a)和20号染色体(图2)。8b).在常见和不常见的MAF范围内的变量的输入与BEAGLE的基线输入具有可比性,基线和基于附近的HMM之间的准确度差异小于1%。罕见变异类别的非参考基因型一致性差异小于2%。这些结果表明,基于邻近的hmm可以潜在地为不常见和常见的变异(即MAF > 1%)提供效用。
讨论
我们分析了使用基于目标变量(即未类型化变量)的局部性计算的hmm输入变量的可行性。只关注非类型化目标的位置有几个优点。首先,计算可以并行化并在更小的规模上执行,而不需要大量的非类型变量。其次,对基于邻域的HMM准确性的评估可以提供对单倍型结构的生物学洞察和归因估计[2]。43]。第三,局部模型可以与基因组的其他部分分离。这样,就可以为其他任务重新设计插补算法。例如,最近发展的具有隐私意识的imputation方法[47,48]广泛使用基于区域的模型。我们的研究结果为安全代入算法的设计提供了见解,以便他们可以适当地选择附近参数,以确保在准确执行代入时使用足够的资源。此外,我们的研究也提供了证据,证明基于hmm的imputation方法可以设计为纯粹的基于邻近的方法。虽然我们没有考虑迭代方法来估计和调整参数,但参数可以使用,例如,期望最大化[63],特别是使用鲍姆-韦尔奇算法[55,56]。
可以引入许多优化来减少基于邻近的hmm的时间和内存使用。例如,可以通过使用单指令多数据(SIMD)操作重新排序向前和向后计算,以简化乘法。此外,我们观察到相邻窗口在计算相邻窗口时基本上重叠。其中许多不需要从头计算,并且可以在相邻的窗口之间重用。由于无型变异(尤其是罕见变异)通常被限制在同一单倍型上,因此彼此非常接近的无型变异(特别是罕见变异)可以联合进行归因。最后,一些彼此接近的类型化变体可能不会为插入提供额外的信息。在计算前向和后向变量时,可以将这些变量视为单个单元。
应该注意的是,默认参数不能提供使用处理非类型化变量局部性的插入hmm所能达到的最佳性能。例如,我们没有评估增加的影响\ ({l} _ {w} \)而类型化变体的最大数量保持不变。这仍然构成一个基于邻近的HMM模型,因为周围类型变体的最大数量是受限的。换句话说,这将保持计算需求不变,但它将使基于邻近的HMM能够评估更大的单倍型块。此外,局部性窗口可以以不同的方式实现。例如,可以根据最小的遗传距离对类型变体进行过滤,即我们可以去除彼此接近且可能仅为imputation提供冗余信息的类型变体。
本文所评估的基于邻近的HMM方法的主要局限性是对于罕见变量的精度较低,特别是对于MAF低于1%的变量。我们的研究结果表明,通过扩展局部窗口以包含更多的变量,可以提高性能。这是合理的,因为较长的窗口比较短的窗口更能准确地分辨出罕见的单倍型。从效用的角度来看,我们观察到大多数下游分析,如全基因组关联研究(GWAS)将阈值设定在远高于1%的范围内[64]。例如,即使是高强度的GWAS研究也会对变异的MAF施加2-5%的阈值,以提供足够的能力来检测表型-基因型关联[65]。此外,即使是最先进的HMM方法也可能无法提供下游分析所必需的低MAF变量的imputation精度。此外,这些罕见的变异往往具有人群特异性[66],使用特定人群的面板可以实现更准确的性能。因此,基于邻近的hmm可用于推算下游任务的MAF值,这些MAF值可用于GWAS等研究。
结论
本研究中参数化的基于位置的hmm可用于并行化和/或局部化计算,而无需执行染色体范围(或非常大规模)的计算。我们假设这些方法可以有效地用于产生基因型imputation,用于下游分析,如GWAS。我们认为,基于位置的hmm的主要用途之一是使用基于位置的方法,这些方法已用于基于机器学习和隐私感知的imputation模型。参数化可以用来指导这些方法中的参数选择。
方法
给出了基于近似的隐马尔可夫模型的Viterbi和前后向估计的计算细节。
描述了非类型化变量局部窗口内的输入hmm
LoHaMMer向前向后计算(图2)1b)和Viterbi(图2)。1C)对类型化变体的估计,也就是说,跟踪只经过类型化变体的单倍型路径。我们假设基因型是阶段性的,基因型矩阵表示为\ ({G} _ {i, j} ^{左(h \) \} \),它代表亲本拷贝上的等位基因\ \ (h)对个人我\ \ ()还有at index的变体\ (j \).父副本有两个值左\ (h \ \ \ {\ mathrm{0,1} \右\}\)表明父亲和母亲的单倍型(反之亦然)。\ ({G} _{我,[1]}^{左(h \) \} \)的等位基因序列我个体的变异在1和之间j,也就是说,\ ({G} _{我,[1]}^{\左(h \右)}= ({G} _{1}我^{\左(h \右)},{G} _{我2}^{\左(h \右)}\点,{G} _ {i, j} ^{\左(h \右)})\).每个变异的等位基因可以有两个值,\ ({G} _ {i, j} ^{左(h \) \} \ \左\ {\ mathrm{0,1} \右\}\),表示参考等位基因和交替等位基因。\ ({} _ {i、k} ^ {(h)} \)表示变量处的HMM状态\ (k \)为我个人。这些状态对应于分阶段参考基因型面板中单倍型的指数,即\ ({} _ {i、k} ^ {(h)} (1, N) \ \).我们用表示未类型化变量的索引\ ({j} _ {\ varnothing} \),即变指标的集合(即< V \ \ (j)),因为基因型缺失。
不同的二次抽样
给定类型化变体(或类型化变体)的最大数量,\ ({\ widehat {n}} _{标签}\), LoHaMMer首先识别当前窗口中的所有变体,该窗口的长度为\ ({l} _ {w} \).考虑到\ ({n} _{标签}> {\ widehat {n}} _{标签}\)是LoHaMMer取每一个变量的总数左(\ \({\压裂{{\ widehat {n}} _{标签}}{{n} _{标签}}\右)}^ {th} \)选择的变体\ ({\ widehat {n}} _{标签}\)在窗户里。如果\ ({n} _{标签}\)小于\ ({\ widehat {n}} _{标签}\),所有类型的变体都用于插入。为了简化表示,我们假设变体索引是基于次采样的变体列表。
类型化和非类型化变异的遗传距离计算R \ (({\ mathbf {}} _ {\ mathbf {k}}) \)
以厘根为单位的遗传距离是一种概率度量,用于衡量减数分裂中两个变异在同一单倍型块中共享的可能性。我们使用遗传距离来定义窗口长度参数(\ ({l} _ {w} \))围绕未类型化的变体。许多先前的研究已经使用估计的人类基因组重组模式生成了遗传图谱作为参考。对于每条染色体,这些图谱列出了从染色体开始到一组用于估计遗传距离的密集标记的累积遗传距离的估计值。用于估计遗传距离的标记不一定与用于推测的类型变异重叠。因此,我们需要插入每个类型变体的遗传距离。给定一个\ ({k} ^ {th} \)类型化(或非类型化)变体,其基因组坐标表示为\ ({pos} _ {k} \)碱基对,我们使用查找表来识别最接近的两个遗传距离标记与基因组坐标\ (l \)和\ \(米),这样\(l\le {pos}_{k}\le m\).作为\ \(米)大于\ (l \),累积遗传距离大于或等于的距离\ (l \),也就是说,\(\Delta \left(m\right)\ge\Delta (l)\),在那里\δ(m) (\ \)表示位于基因组坐标的标记的累积遗传距离\ \(米).我们估计了分型变异的累积遗传距离\ (k \)使用距离的线性插值\ (l \)和\ \(米)基因组距离加权:
这是对每条染色体分别进行的。在对所有类型和非类型变体分配遗传距离后,距离之间的差异,例如,R \(左\ | {}_ {k} - {R} _ {n} \右| \),用于参数基准测试。我们使用来自IMPUTE2网站(https://mathgen.stats.ox.ac.uk/impute/1000GP_Phase3.html).
边际概率估计的正向后算法
前向向后算法依赖于前向和后向变量的计算。考虑到个人我\ \ ()和单体型\ \ (h),正向概率表示为
在哪里\ (P \离开({}_ {i, j} ^ = {(h)}, {G} _{我,[1]}^ {(h)} \) \)表示前向变量,表示所有状态序列和状态序列的排放的总概率\({} _{我,\ [1,j \]} \)发出等位基因序列\ ({G} _{我,[1]}^ {(h)} \)约束条件是最后的状态是变的\ (j \)是\ \ (),也就是说,\ ({} _ {i, j} ^ {(h)} = \).前向变量矩阵可以递归计算[55,56]适用于所有不同的位置和所有的状态
前向变量在哪里\ (j \)是使用forward变量在位置上计算的吗\ (\) (j - 1).边界条件在第一个核苷酸处定义:
这表明第一个变异的状态在所有状态中是均匀分布的,即在启动HMM的单倍型之间不存在偏好。这种边界条件有时通过引入一种称为“起始状态”的特殊状态来描述。
后向概率公式为
(15),\ (P \离开({G} _{我[j + 1, V]} ^{\左(h \右)}|{年代}_ {i, j} ^ {(h)} =一个\)\)表示的向后变量我个体的单倍型\ \ (h),所有状态序列的总概率,\({} _{我,\ [j, V \]} ^ {(h)} \),产生等位基因子序列G \({} _{我[j + 1, V]} ^{\左(h \右)}\)约束条件是第一状态是变的\ (j \)是\ \ (),也就是说,\ ({} _ {i, j} ^ {(h)} = \).后向变量可以使用递归关系[55,56使用下列方式:
为窗口的末端设置向后变量的边界条件:
这表明单倍型均匀分布在等位基因序列的末端。发射和跃迁概率相应地在正向和向后变量的边界处设置为均匀。
计算未分型变异的等位基因和基因型概率
向前和向后变量用于推断在未类型化变异中观察到等位基因0和1的概率。估计索引处未类型化变体的等位基因概率\(j\in {j}_{\varnothing}\), LoHaMMer标识最接近该变体的两个连续类型变体\ (j \).LoHaMMer使用最近的类型化变体,使用类似于BEAGLE的方法来估计沿非类型化变体传递的路径
在哪里\ ({j} _ {\ varnothing} \)表示基因型矩阵中未分型的变异指数,和\ (k \)变项指标是这样的吗\ (k \)和\左(k + 1 \右)(\ \)最接近类型化变体的是非类型化变体吗\ (j \).来自亲本拷贝的等位基因概率被归一化并组合以产生3种可能的基因型的最终基因型概率,即分配给个体的两个单倍型的等位基因的组合。基因型概率计算为
在哪里\ (P ^{\ '} \离开({G} _ {i, j} ^{\左(h \右)}= t \) \)(左\ (t \ \ \ {\ mathrm{0,1} \右\},h \中\ {\ mathrm {0,1} \} \))为单倍型归一化等位基因概率\ \ (h)和等位基因\ \ (t).\ (P ^{\ '} \离开({G} _ {i, j} ^{\左(h \右)}= t \) \)是通过对变体的总等位基因概率进行归一化计算的,因此范围在(\ \ (mathrm {0,1}] \).具体来说,我们使用
在哪里对亲本副本的两个可能的等位基因概率进行归一化\ \ (h)等位基因的\ \ (t).
Viterbi算法的最大似然单倍型路径估计
与前向矩阵类似,Viterbi方法跟踪每种可能的单倍型状态下每种类型变体的最大得分矩阵:
在哪里\ (P ^{\楔}\离开({\打翻{\ lower0.5em \ hbox{$ \粉碎{\ scriptscriptstyle \微笑}$}}{年代}}_ {i, j} ^ = {(h)}, {G} _{我,[1]}^ {(h)} \) \)表示该等位基因序列存在的概率\ ({G} _{我,[1]}^ {(h)} \)由最可能的状态序列发出\({\检查{年代}}_{我,[1]}^ {(h)} \)加上附加的约束条件\({\检查{年代}}_ {i, j} ^ {(h)} = \).这条路径是LoHaMMer用来推断最可能产生类型化等位基因序列的单倍型镶嵌的最可能路径。(23)与(中的前向变量完全相同。12)中最左边的和(12)被替换为最大值运算符。因此,\ (P ^{\楔}\离开({\打翻{\ lower0.5em \ hbox{$ \粉碎{\ scriptscriptstyle \微笑}$}}{年代}}_ {i, j} ^ = {(h)}, {G} _{我,[1]}^ {(h)} \) \)可以使用类似的递归计算,将求和替换为(中的最大值操作符)。13):
LoHaMMer使用这种递归关系,对所有单倍型从左到右的每个类型变体计算Viterbi矩阵,并具有边界条件:
对于前向和后向矩阵,Viterbi矩阵是在类型变量上计算的。
LoHaMMer计算出Viterbi矩阵后,追溯Viterbi矩阵,确定最优状态序列,即发射完整等位基因序列的最优单倍型集合:
在分配最佳状态后,LoHaMMer将等位基因分配给未类型化的变体,类似于正向向后算法。对于索引处的未类型化变体\ (j \), LoHaMMer识别最接近的类型变体,并根据类型变体上的最大似然状态分配等位基因:
在哪里\ (k \)与非类型化变体最接近的类型化变体是否在\ (j \).
变异块中的单倍型聚类
Viterbi变量和forward-backward变量的递归关系表明,有必要对参考面板中的所有单倍型(对于每个类型的变体)执行求和(或最大操作)。随着单倍型的数量(\ (N \))增加。与之前的方法类似,LoHaMMer对单倍型进行聚类,在参考单倍型的集群上计算每个正向、向后和Viterbi数组,以尽量减少冗余操作的数量。聚类极大地提高了效率,因为(1)短时间内唯一单倍型的数量比单倍型的数量增加得慢得多,(2)状态之间的转移概率仅取决于自转移和重组。这些优化在前面的方法中有广泛的描述。我们简要地描述了聚类在维特比数组计算中的应用。LoHaMMer选择一些变体,这些变体将用于聚类参考单倍型,默认情况下,块长度被选择为10个变体。给定一个局部窗口,LoHaMMer将窗口分成10个变量的块。接下来,将每个块上的参考单倍型聚集在一起,使每个簇对应于10个等位基因的唯一序列,对应于块中的10个变体。接下来,对于每个集群,计算Viterbi变量为集群中单倍型上Viterbi变量的最大值。由于集群完全共享等位基因序列,因此使用块中10个变体的递归关系在集群级别计算集群的Viterbi变量。 After cluster-level Viterbi variables are computed for each cluster, LoHaMMer assigns the Viterbi variable to each haplotype from their corresponding cluster-level Viterbi variables.
数值稳定性
转变和排放概率小于1,它们在所有转变和排放上相互相乘。因此,Viterbi变量和前后向变量可能超出或低于数值精度。为了解决这些数值稳定性问题,LoHaMMer可以在对数域中执行计算,或者通过缩放因子缩放Viterbi和前后向变量。对于对数域计算,LoHaMMer将每个值保持为对数。在对数域中,乘法被转换为求和,这很方便,因为溢出实际上是不可能的。然而,我们观察到对数域的近似求和需要大量缓慢的操作(对数域的求和需要幂运算),并且增加了时间要求。因此,LoHaMMer默认使用线性缩放值。为此,LoHaMMer将每个数组值乘以一个常数缩放因子。我们通过试错观察到比例因子为\ (\ mathrm {exp} (0.2) \)启用最少数量的下溢或溢出问题。LoHaMMer在每个计算步骤中跟踪任何溢出和下溢。如果数组值过高或过低,则会重新缩放值以确保数值稳定性。
精度指标的计算
非参考基因型一致性
基因型一致性计算为已知非参考基因型之间的重叠。更正式的表述是
在哪里\ ({\ kappa} _ {j} ^ {(NR)} \)表示变异的已知非参考基因型之间的非参考一致性\ (j \)以及所有个体的估算变异。
数据源
1000个基因组计划基因型从NCBI ftp数据门户下载。Illumina Duo v3变体是从阵列文档中提取的,可在:https://zenodo.org/record/5482126#.YTcAEM9On3g.1000基因组计划中与阵列类型变体重叠的变体被用作类型变体。
数据和材料的可用性
1000个基因组计划基因型从NCBI ftp数据门户下载http://ftp.1000genomes.ebi.ac.uk/vol1/ftp/release/20130502/.Illumina Duo v3变体是从阵列文档中提取的,可在:https://zenodo.org/record/5482126#.YTcAEM9On3g.1000基因组计划中与阵列类型变体重叠的变体被用作类型变体。源代码基于附近的输入,数据处理脚本,和中间数据文件可以从https://github.com/harmancilab/LoHaMMer.hg19人类染色体的遗传图谱可以从https://mathgen.stats.ox.ac.uk/impute/1000GP_Phase3.html.
参考文献
Goldfeder RL, Wall DP, Khoury MJ, Ioannidis JPA, Ashley EA.基于种群规模的人类基因组测序:高通量DNA测序和分析引物。中华流行病学杂志,2017;36(1):1 - 4。
雷姆曾为此写过霍奇金淋巴瘤。通过个人基因组学发展医疗保健。农学通报,2017;18:259-67。
我们应该对每个人的基因组进行测序吗?是的。BMJ。2013; 3133:2-3。
Schloss JA, Gibbs RA, Makhijani VB, Marziali A.人类基因组计划后的DNA测序技术培养。中国生物医学工程学报,2020;21(1):117 - 38。https://doi.org/10.1146/annurev-genom-111919-082433.
Pepke S, Wold B, Mortazavi A. ChIP-seq和RNA-seq研究的计算方法。自然科学学报,2009;6:522 - 532。
Bycroft C, Freeman C, Petkova D, Band G, Elliott L, Sharp K,等。约50万英国生物银行参与者的全基因组遗传数据。bioRxiv。2017.
Collins FS, Varmus H.精准医疗的新倡议。中华医学杂志。2015。
Chisholm J, Caulfield M, Parker M, Davies J, Palin M. briefen - genomics England and 100K Genome Project。英国基因组学,2013。http://www.genomicsengland.co.uk/briefing/.
李敏,李涛,邓凯威奇,李志强,李志强,等。解释和报告癌症序列变异的标准和指南:分子病理学协会、美国临床肿瘤学会和美国病理学家学会的联合共识推荐。中华医学杂志,2017;19(4):444 - 444。
刘建军,刘建军,刘建军,刘建军,等。ACMG对报告临床外显子组和基因组测序中偶然发现的建议。基因医学。2013。
Guerrini CJ, Contreras JL。基因组公民科学研究成果的信用和控制。中国生物医学工程学报,2020;21(1):465 - 469。https://doi.org/10.1146/annurev-genom-083117-021812.
埃文斯JP。休闲基因组学;这对你有什么好处?中华医学杂志。2008;10:709-10。https://doi.org/10.1097/GIM.0b013e3181859959.
Bergström A, McCarthy SA, Hui R, Almarri MA, Ayub Q, Danecek P,等。从929个不同的基因组了解人类遗传变异和种群历史。科学》2020。https://doi.org/10.1126/science.aay5012.
张建军,张建军,张建军,等。英国生物银行:一个开放获取的资源,用于确定各种复杂的中老年疾病的原因。中华医学杂志。2015;12:e1001779。
张建军,张建军,张建军,等。多基因风险评分的个人和临床应用。中华宗教学报,2018;19:58 81 - 90。
布什WS,摩尔JH。第11章:全基因组关联研究。计算机工程学报,2012;33(2):591 - 591。
陈伟,陈文杰,李建平。从全基因组关联到统计精细映射的候选因果变异。中华宗教学报,2018;19:491-504。
Sella G, Barton NH。全基因组关联研究时代对复杂性状进化的思考。生物医学学报,2019;20(2):461 - 93。https://doi.org/10.1146/annurev-genom-083115-022316.
蹒跚。关联研究的标签SNP选择。遗传流行病学杂志。2004;27:365-74。
Fallin D, Schork NJ。非分期二倍体基因型数据的期望最大化算法对双等位基因座单倍型频率估计的准确性。[J] .中国生物医学工程学报。2000;37(2):947 - 959。
shoner A, Mu X, Greenbaum D, Auerbach RK, Gerstein MB.测序的实际成本:比你想象的要高!中国生物医学工程学报。2011;12:125。
Illumina宣布了一个新的基因分型阵列和对三个基因组中心的科学贡献,以支持我们所有人的研究计划。https://www.illumina.com/company/news-center/press-releases/2018/2379861.html.2020年10月5日访问。
Das S, Abecasis GR, Browning BL.大型参考面板的基因型插入。植物学报,2018;19:73-96。
李云林,王志强,王志强,王志强。生物医学学报。2009;10:387-406。
国际HapMap 3联盟。整合不同人群中常见和罕见的遗传变异。大自然。2010;467:52-8。https://doi.org/10.1038/nature09298.
血统的同一性:减数分裂的变异,跨基因组,在群体中。遗传学。2013;194:301-26。https://doi.org/10.1534/genetics.112.148825.
王志强,李建平。基于血缘关系的远亲身份识别方法研究。中国生物医学工程学报,2012;46(6):617 - 633。
周勇,陈建军,李建军。一种快速简便的大规模数据下降识别方法。[J] .中国生物医学工程学报。2010;26(1):426 - 426。
刘霞,唐凯,张生,志东。快速:生物库规模队列中超快速、高效、准确的IBD基因段同源检测方法。中国生物医学工程学报,2019;20:143。https://doi.org/10.1186/s13059-019-1754-8.
拉宾LR。一个关于隐马尔可夫模型和语音识别中选定应用的教程。Proc IEEE。1989; 77:257 - 86。
Yoon B-J。隐马尔可夫模型及其在生物序列分析中的应用。基因工程学报。2009;10:42 - 15。https://doi.org/10.2174/138920209789177575.
张洪波,Rabiner LR。语音识别的隐马尔可夫模型。技术计量学。1991;33:251 - 72。
陈建军,陈建军。基于潜马尔可夫模型的纵向数据分析。2012.
李廷辉,Macdonald 1, Zucchini W.离散值时间序列的隐马尔可夫模型。[J]中华医学会杂志。1998;93:1525-7。
李娜,Stephens M.基于单核苷酸多态性数据的连锁不平衡建模与重组热点识别。遗传学。2003;165:2213-33。
周毅,周玉华,陈建平,等。一种基于基因序列的基因组图谱。[J] .中国生物医学工程学报,2018;33(3):391 - 391。
王晓明,王晓明,王晓明,等。基于位置burrows wheeler变换的基因型估算方法。科学通报,2020;16:e1009049。
王晓明,王晓明,王晓明,王晓明,等。下一代基因型插补服务与方法。生物医学工程学报,2016;48:1284 - 1287。
王晓明,王晓明,王晓明,等。基于基因分型的全基因组关联分析。生物医学学报,2012;44(4):955 - 959。
Van Leeuwen EM, Kanterakis A, Deelen P, Kattenberg MV, Slagboom PE, De Bakker PIW等。使用minimac或IMPUTE2进行人群特异性基因型估算。Nat protocol . 2015; 10:1285-96。https://doi.org/10.1038/nprot.2015.077.
Taliun D, Harris DN, Kessler MD, Carlson J, Szpiech ZA, Torres R,等。来自NHLBI TOPMed项目的53831个不同基因组测序。大自然。2021;590:290-9。
黄杰,Howie B, McCarthy S, Memari Y, Walter K, Min JL,等。使用UK10K单倍型参考面板改进低频和罕见变异的插入。自然科学学报。2015;6:1-9。
段强,刘永英,Croteau-Chonka DC, Mohlke KL,李毅。一个综合SNP和indel归责性数据库。生物信息学。2013;29:528-31。
陈静,石鑫。稀疏卷积去噪自编码器的基因型插入。基因。2019;10:652。
李建军,李建军,李建军,等。一种基于神经网络的单倍型参考信息基因型估算方法。计算机工程学报,2016;16(2):591 - 591。
Kim M, Harmanci A, Bossuat J-P, Carpov S, Cheon JH, Chillotti I,等。超高速同态加密模型使基因型植入的安全外包成为可能。bioRxiv。2020.
Kim M, Harmanci AO, Bossuat J-P, Carpov S, Cheon JH, Chillotti I,等。超快同态加密模型使基因型植入的安全外包。Cell system, 2021。https://doi.org/10.1016/j.cels.2021.07.010.
郝曼奇,金敏,王松,李伟,宋勇,柯劳特,等。开放的代入服务器提供安全的代入服务,具有可证明的基因组隐私。bioRxiv。2021.https://doi.org/10.1101/2021.09.30.462262.
Loh PR, Danecek P, Palamara PF, Fuchsberger C, Reshef YA, Finucane HK,等。使用单倍型参考联盟面板的基于参考的分期。中国生物医学工程学报,2016;48:1443-8。
王晓明,王晓明,王晓明,赵辉。基于可信任执行环境的隐私保护基因型植入。bioRxiv。2021.https://doi.org/10.1101/2021.02.02.429428.
grsoy G, Chielle E, Brannon M, Maniatakos M, Gerstein M.全同态加密的隐私保护基因型插入。细胞系统。2022;13:173-82。
一个完全同态的加密方案。博士学位论文。2009;1 - 209。https://doi.org/10.1145/1536414.1536440.
金明,宋勇,姜鑫。基于隐马尔可夫模型评估的基因组序列隐私感知比对。2021.https://doi.org/10.21203/rs.3.rs-954109/v1.
李建军,李建军,李建军,等。一种线性码误码率的优化译码方法。计算机工程学报。2004;19(2):387 - 398。
韦尔奇LR。隐马尔可夫模型和鲍姆-韦尔奇算法。光电工程学报,2003;33(3):1010 - 1010。
李建军,李建军,李建军,等。一种马尔可夫链概率函数统计分析中的最大化方法。数学统计。1970;41(1):164 - 71。
福尼GD。维特比算法。Proc IEEE。1973; 61:268 - 78。
Skelly DA, Magwene PM, Stone EA.非连锁分离位点间的零星连锁不平衡。遗传学。2016;202:427-37。https://doi.org/10.1534/genetics.115.177816.
千人基因组计划联盟。人类遗传变异的全球参考。大自然。2015;526:68 - 74。https://doi.org/10.1038/nature15393.
Frazer KA, Ballinger DG, Cox DR, Hinds DA, Stuve LL, Gibbs RA等。第二代人类单倍型图谱超过310万个snp。大自然。2007;449:851 - 61。
Bryc K, Durand EY, Macpherson JM, Reich D, Mountain JL。美国各地的非裔美国人、拉丁裔美国人和欧裔美国人的遗传祖先。[J] .中国生物医学工程学报。2015;36(6):557 - 557。
crucani F, La Fratta R, Santolamazza P, Sellitto D, Pascone R, Moral P,等。单倍群E3b (E-M215) Y染色体的系统地理分析揭示了非洲内外的多次迁徙事件。[J] .中国生物医学工程学报。2004;24(4):444 - 444。
Dempster AP, Laird NM, Rubin DB。通过EM算法从不完整数据中获得最大似然。[J] .社会科学与技术。2007;19(1):1 - 2。
Malik R, Chauhan G, Traylor M, Sargurupremraj M, Okada Y, Mishra A,等。52万名受试者的多祖先全基因组关联研究确定了与中风和中风亚型相关的32个位点。生物医学工程学报,2018;50:524-37。
宋玉杰,Winkler TW, de las Fuentes L, Bentley AR, Brown MR, Kraja AT,等。一项考虑吸烟行为的大规模多祖先全基因组研究发现了多个与血压有关的重要基因位点。[J] .中国生物医学工程学报,2018;22(2):379 - 379。
Mitt M, Kals M, Pärn K, Gabriel SB, Lander ES, Palotie A,等。使用人群特异性高覆盖率基于wgs的输入参考面板提高罕见和低频变异的输入精度。中国生物医学工程学报,2017;25(5):869 - 876。
致谢
我们感谢陈路遥为建立基准测试实验的计算环境提供技术支持。
资金
在这项工作中,SW和AH得到了德克萨斯大学休斯顿健康科学中心的启动资金支持。MK由UNIST(蔚山国家科学技术研究所)的结算研究基金(No. 1.200109.01)和韩国政府(MSIT)资助的信息和通信技术规划与评估研究所(IITP)资助(No. 2020-0-01336,人工智能研究生院支持(UNIST))支持。XJ是CPRIT癌症研究学者(RR180012),他得到了Christopher Sarofim家庭教授,UT Stars奖,UTHealth创业公司,美国国立卫生研究院(NIH)(奖励号R13HG009072, R01GM114612)和美国国家科学基金会(NSF) RAPID #2027790的部分支持。
作者信息
作者及单位
贡献
SW和AH构思了方法论思想,收集了数据集,设计并实施了实验。SW进行实验,收集所有图的结果并将其制成表格。AH和SW起草了手稿。XJ和MK监督研究设计,并编辑手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1。
这个额外的文件包含补充文本和数字与扩展的讨论和准确性的结果。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
王,S.,金,M.,蒋,X.。et al。基于邻近的隐马尔可夫模型用于基因型估算的评价。BMC生物信息学23, 356(2022)。https://doi.org/10.1186/s12859-022-04896-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04896-4
关键字
- 基因型归责
- 隐马尔可夫模型
- Forward-Backward算法
- 维特比算法