摘要
背景
从生物医学文献和其他文本数据中提取药物相互作用是监测药物安全性的重要组成部分,这引起了许多医疗保健研究人员的关注。现有的工作更多地围绕着使用双向长短期记忆网络(BiLSTM)和BERT模型进行关系提取,这并不能获得最佳的特征表示。
结果
我们提出的DDI(药物-药物相互作用)预测模型具有多个优点:(1)加入新提出的注意向量,更好地处理重叠关系问题;(2)将药物的分子结构信息整合到模型中,更好地表达药物的官能团结构;(3)我们还添加了结合t分布和卡方分布的文本特征,使模型更专注于药物实体。(4)在基准数据集上测试时,与最先进的DDI模型相比,它实现了相似或更好的预测性能(F-scores高达85.16%)。
结论
我们的模型利用了最先进的变压器架构,并结合多种特征,可以增强生物医学领域药物-药物相互作用任务的性能。特别是,我们相信我们的研究将有助于识别潜在的药物不良反应。
背景
多药联用,即同时使用多种药物,近年来在患者中越来越多[1,2,3.].当使用多种药物时,它们之间可能会发生相互作用,通常称为药物-药物相互作用(DDI)。因此,一种药物的预期效果可能会被另一种药物的作用所改变。这些影响可能导致药物协同作用[4],降低疗效,甚至致毒性[5].因此,DDI相互作用提取是改善患者治疗和安全性的重要一步。
传统上,医生获得关于DDI的最新信息主要有两个来源:阅读大量生物医学论文来了解DDI或从生物医学数据库中查询DDI。在生物医学领域,生物医学文献的数量迅速增加。显然,阅读大量的论文是没有效率的。对于生物医学数据库,似乎可以,但考虑到生物医学文献的数量,手动更新和修改一个专业的数据库需要大量的资源。因此,这两种方法都不是理想的获取DDI的方法。
DDI提取任务[6]旨在从生物医学领域的自由文本中提取DDI。DDIExtraction 2013任务试图根据五种类型(“建议”、“效果”、“机制”、“Int”和“假”)之一对每个DDI候选人进行分类。
在早期,人们经常采用基于模式的方法和基于特征的机器学习方法[7,8],但基于模式的方法要求标注者具有一定的领域知识,该方法的主要缺点是费时且效率低。
深度学习是目前应用最广泛、最有效的解决这一问题的方法,主要包括基于cnn的方法[9,10,11,12,13,14,15],基于rnn的方法[16,17,18,19,20.,21,22],目前最好的方法是基于预训练。一般来说,由于循环连接,RNN适用于NLP应用[23,24],但RNN存在爆炸和梯度消失的问题[25].为了解决这些问题,长期短期记忆(LSTM) [26,27]单元和门控循环单元(GRU) [28网络被提出。
近年来,基于预训练的方法[29,30.取得了很好的效果。Lee等人引入了BioBERT(用于生物医学文本挖掘的变压器双向编码器表示)来改进DDI提取[31],作者在PubMed摘要(PubMed)和PubMed Central全文文章(PMC)上对BioBERT进行预训练。Boukkouri等人提出了BERT的新变体[32],它完全抛弃了chunk系统,并使用了一个character-CNN模块,而不是通过查询它们的字符来表示整个单词。最近,Sun等人。[33]在BioBERT的基础上,引入高斯向量等外部知识,进一步提高了DDIExtraction 2013任务的提取效果。
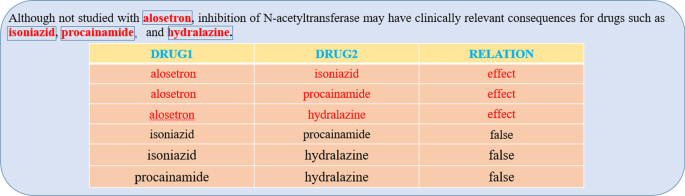
上面提到的解决方案有一些缺点。首先,如图所示。1,在含有DDIs的句子中,存在多种复杂的药物-药物相互作用。例如,药物“阿洛司琼”和其他三种药物(“异烟肼”、“普鲁卡因酰胺”、“联氨嗪”)都有作用。我们把这种关系称为重叠,对于这种复杂的关系重叠,以上的方法都没有很好的解决方法。新提出的注意向量旨在更好地处理重叠关系的问题。其次,许多已批准的药物都有大量的药物特征信息,包括分子结构、药物smile等。以上所有方法都忽略了这个额外但非常有用的信息。因此,我们通过DrugBank获得了药物的SMILES分子式[34]数据库,并通过RDKit工具包将SMILES分子公式转化为指纹和邻接矩阵[35来获取药物的分子特征。此外,文本中药物实体的位置也非常有助于提取具体的药物关系。为此,我们引入t分布和卡方分布来获得关注药物实体的句子特征。
我们其余的工作安排如下。我们首先详细讨论了我们的方法,即系统架构,然后是实验设置,培训和评估指标。然后对实验结果进行了讨论,最后给出了结论。
方法
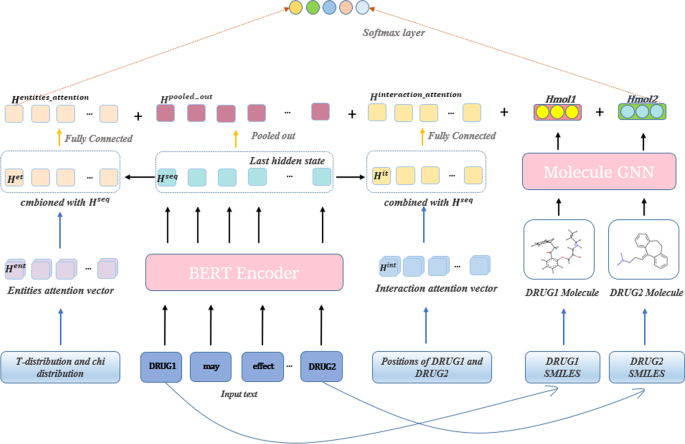
在本节中,我们将介绍我们的系统架构(图。2),并按顺序解释它调用的不同模块。数字2展示了我们方法的体系结构。我们的模型分为四个部分。首先,我们使用Biobert对输入句子进行编码,并获得最后一个隐藏状态(\ ({H} ^ {seq} \))的Biobert。然后根据DRUG1和DRUG2的位置生成注意向量,得到有助于识别重叠关系的“交互特征”,通过引入卡方分布和t分布得到关注实体的“实体注意特征”。最后,根据DrugBank数据库和RDKit工具库获得药物结构,并利用分子图神经网络[36]以生成高质量的分子表征。最后,我们结合所有的信息对DDI进行分类。我们将在下面的内容中详细介绍我们的模型和方法。
数据预处理
药物面具
对于句中需要分类关系类型的两个药物实体,使用“DRUG1”和“DRUG2”替换,对于句中其他药物实体,使用“DRUGOTHER”替换。
过度抽样和不足抽样
DDI 2013数据集存在长尾分布现象。因此,采用欠采样和过采样的方法来优化训练集。与原始数据相比,过采样过滤掉了大量重复的负样本,而过采样可以显著增加稀疏样本的数量。
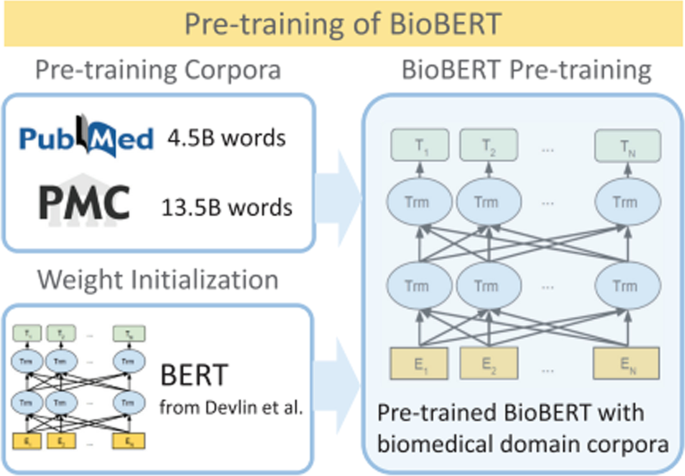
BioBERT预训练概述[31].BioBERT是一个在大规模生物医学语料库上预训练的领域特定语言表示模型。通过几乎相同的任务架构,当在生物医学语料库上进行预训练时,BioBERT在各种生物医学文本挖掘任务中大大优于BERT和以前最先进的模型
句子编码器
该组件的目标是获得句子中每个标记的上下文感知表示。鉴于最近在语言建模变体上训练的深度变压器的令人印象深刻的性能,我们使用BioBERT模型作为句子编码器。BioBERT在大量PubMed摘要和PubMed Central全文文章上进行了预训练,见图。3.对于预培训的概述。给定一个序列(\ (S = \{间的{1},间的{2},\点间{n} \} \))作为输入,生物机器人的公式如下:
在哪里西是我th令牌,l为BERT的总层数,l(1\ (< < l \))是lth层。方程(1)表示输入嵌入,式(3.的表示我-th令牌,Eq. (4)表示序列的表示。式中的transformer_block (2)包含多头注意层、全连接层和输出层。此外,参数\ ({\ varvec {W}} ^ {e} \),\ ({\ varvec {W}} ^ {b} \),变压器\ _block \ \ ()在大规模语料库上使用两个无监督的预训练任务,蒙面语言模型和下句预测进行预训练。BioBERT模型的输出是标记的上下文感知嵌入,表示为\({\varvec{H}}^{seq} \in R^{n \ * d}\),在那里n是句子长度(包括[CLS]和[SEP],两个特殊的开始和结束标记),和d为BERT模型中隐藏单元的数量。
交互注意向量
在这一部分中,我们将介绍交互注意向量。如图中的句子所示。1,如果这两种药物有某种相互作用,它们在句子中就会相距很远,而如果它们没有相互作用,它们就会靠得更近,这表明表征相互作用的重要信息往往在这两个实体之间。此外,我们在绝大多数生物学文献中都发现了这种模式。因此,在处理重叠关系时,我们应该关注当前药物对之间的信息。在表1,给出SEMEVAL-2013数据集中实体间交互信息的统计。我们分别对训练集、验证集和测试集进行了统计,结果表明,数据集之间的大量数据符合这一模式。也就是说,我们应该更加关注实体之间的内容。下面我们将详细解释如何生成交互注意向量以及如何使用它。
我们定义了一个高权重范围和一个低权重范围:
我们将高权重分配给两种药物之间的信息,将低权重分配给句子的其余部分。为了保持这个权重范围的弹性,我们添加了一个振荡因子σ\ (\ \)到体重范围。加入冲击因子后的权重如下:\ ({\ varvec {W}} _{高}\)是我们分配给两种药物之间信息的权重范围,\ ({\ varvec {W}} _{低}\)是我们将分配给句子其余部分的权重范围:
在哪里σ\ (\ \)是振荡因子(这里取0.1)。
我们定义一个句子\ (S = \{间的{1},间的{2},\点间{n} \} \),我们最终提出的交互注意向量定义如下:
在哪里开始而且结束意思是句子的开头和结尾,j而且k意思是句子中头实体和尾实体的位置,\ ({\ varvec {H}} ^ {int} \)为最终交互注意向量,其可视化表示如下:
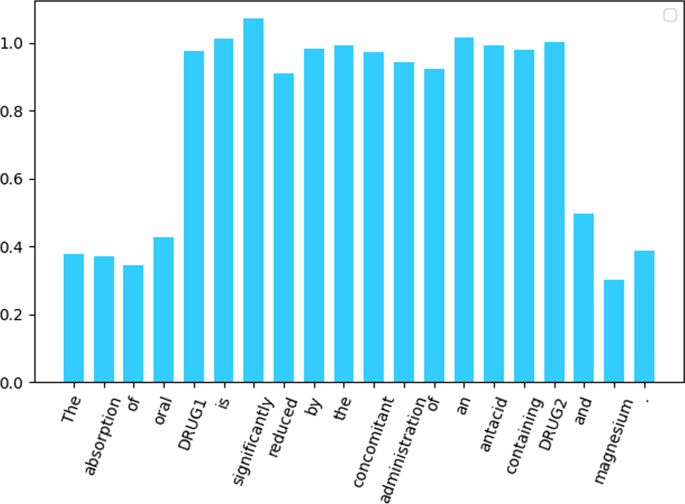
BERT模型的输出(\(H^{s e q} \in R^{d \乘以1}\))是标记的上下文感知嵌入。然后我们做一个矩阵运算\ ({\ varvec {H}} ^ {int} \),这一步的输出如图所示。4,每个词的嵌入\ ({\ varvec {H}} ^ {seq} \)给出不同的权重,公式如下:
在哪里输入\ \ ({_sentence} \)是句子的原始输入,\ ({\ varvec {H}} ^{我t} \)(\({\varvec{H}}^{i t} \in R^{d \乘1}\))为融合后得到的合成向量\ ({\ varvec {H}} ^ {int} \)而且\ ({\ varvec {H}} ^ {seq} \),\ \ otimes \ ()表示矩阵乘法。
我们还将平均运算应用于综合向量表示法\ ({\ varvec {H}} _{我t} \):
在哪里\ ({\ varvec {H}} ^{交互\ _attention} \)是经过平均处理后的输出吗\ ({\ varvec {H}} ^{我t} \)一个完全连接的层,j而且k是第一种药物和第二种药物的位置。
用于隐藏状态输出\ ({\ varvec {H}} ^ {seq} \),我们首先从它获得pooled输出,然后添加一个激活操作和一个全连接层,其形式表示为:
在矩阵\({\mathbf{W}}_{\text{int}}\ in R^{n \times 1}\),\({\mathbf{W}}_{\text{0}} \in R^{n \times 1}\)是权重矩阵,它们有相同的维数,\ ({\ mathbf {b}} _ {\ mathbf {int}} \),\ ({\ mathbf {b}} _ {\ mathbf {0}} \)是神经网络的偏差。
实体注意力特征
在本研究中,我们引入卡方概率分布和t分布来增强目标实体及其相邻单词的权重,使模型能够学习实体的局部结构。我们把这两个经过修改的分布统称为实体注意力向量。卡方概率密度函数为:
卡方概率分布函数为:
T概率密度函数为:
T概率分布函数为:
在哪里x是一个实值,\γ(\ \)是函数,k为卡方分布的自由度,n是T分布的自由度和t为卡方函数的步长,w为T函数的步长。
我们首先得到分布的值,然后定位两个实体在句子中的位置。我们根据从右向左位置和高低值的规则在第一个实体之前映射15%,根据从左向右位置和高低值的规则在第一个实体之后映射25%,然后对右边的实体做对称操作。
最后得到实体的注意向量\ ({\ varvec {H}} ^ {ent} \)无花果。5,然后与BioBERT的最后一个隐藏状态输出(\ ({\ varvec {H}} ^ {seq} \)),并添加激活操作和全连接层。公式如下:
在哪里\ ({H} ^ {ent} \)为实体注意向量,\ ({\ varvec {H}} ^ {e t} \)(\(H^{e t} \in R^{1 \ * d}\))为融合后得到的合成向量\ ({\ varvec {H}} ^ {ent} \)而且\ ({\ varvec {H}} ^ {seq} \),\ ({\ varvec {H}} ^{实体\ _attention} \)是激活操作和全连接层后的输出,\ ({\ mathbf {W}} _{\文本{ent}} \在R ^ {n \ * 1} \)是权重矩阵,\ ({\ mathbf {b}} _ {\ mathbf {ent}} \)是全连通层的偏置。
分子结构
DrugBank是一个免费提供的药物数据库,包含1万多种药物。根据药物名称,我们首先在DrugBank数据库中找到相应药物的SMILES公式,然后利用Tsubaki等人提供的提取方法提取相应的药物结构[36].数字6这是我们得到的药物的分子结构。我们使用r-半径子图,由邻近顶点和距离顶点半径r内的边诱导。这r是从当前顶点到附近顶点的跳数,我们取半径r1。
利用分子图神经网络对分子图结构进行编码。gnn转换药物分子图G变成一个固定大小的向量。在图中,我们将原子表示为节点,将键表示为边。然后将指纹向量输入分子图神经网络,该神经网络将输入的指纹向量作为初始向量,并根据分子图的结构进行更新。我们dene向量我药物分子中的原子\ ({\ varvec {m}} _{我}\)相邻原子的集合是\ (N_{我}\).向量\ ({\ varvec {m}} _{我}\)已在\ \)(\拼-第一步如下:
在哪里f()表示一个ReLU函数。药物分子向量是通过将所有原子向量相加得到的,然后将得到的向量送入线性层。
在哪里米是指纹的数量,\ ({\ varvec {H}} ^{摩尔}\)为MGNN的输出,由此得到两种药物实体的分子结构输出:\ ({\ varvec {H}} ^ {mol1} \)而且\ ({\ varvec {H}} ^ {mol2} \).
Softmax层
在这一步中,我们将结合前面获得的输出来生成融合表示\ ({\ varvec {H}} \).然后采用全连接神经网络学习表征\ ({\ varvec {H}} \).最后,利用softmax函数进行概率计算P属于DDI类型r:
在哪里\ ({\ mathbf {W}} _ {\ mathbf {*}} \)而且\ ({\ mathbf {b}} _ {\ mathbf {*}} \)为权重参数和偏置参数,我们使用交叉熵函数作为损失函数。
数据集和实验设置
DDIExtraction 2013语料库是一个基于DrugBank数据库和MEDLINE摘要的人工注释药物-药物相互作用(DDI)语料库。这个语料库包含四种用于评估的DDI类型,分别是“Advice”、“Effect”、“Mechanism”、“Int”,以及一个名为“False”的标签,表示没有交互作用。我们将DDI提取表述为一个多类分类问题。我们遵循DDIEXTRACT-2013共享任务(semeval-2013 Task9.2),这里我们参考Asada等人提供的DDI语料库。[15].表格2说明了DDIExtraction 2013数据集中实例的统计信息。
该任务定义了以下四个交互标签。
机制:在输入语句中描述药代动力学机制时指定此类型。
Effect:在描述DDI的效果时分配该类型。
建议:当描述有关两种药物同时使用的建议或建议时,会指定此选项。
Int(交互):当句子只是说明发生了交互,而没有提供关于交互的任何详细信息时,就分配这种类型。
在实验中,我们使用PyTorch (https://pytorch.org/)框架,以落实我们建议的模型。对于BERT模型的选择,我们选择了BioBERT模型对输入的句子进行编码。所有的DDI提取方法都使用标准的评价指标(精密度、召回率和F-score)作为评价指标。f分数的定义为:\(f1 = 2pr /(p + r)\).
结果
IMSE在基准数据集上的性能及分析
表格3.详细说明了实验结果。我们将该模型与基于CNN、RNN和预训练的典型模型进行了比较。BioBERT使用PubMed Abstracts (PubMed)和PubMed Central full - text Articles (PMC)进行预训练。CharacterBERT模型使用了一个字符cnn模块,而不是通过查询它们的字符来表示整个单词。ChemicalBERT + AGGCN (Parallel)是将GCN和ChemicalBERT结合起来的DDI任务模型。总的来说,基于LSTM的模型比基于cnn的模型取得了更好的结果,因为LSTM结构可以处理较长的文本,而cnn更关注局部特征。此外,基于预训练的模型比其他方法表现更好,例如,使用大量生物医学文本预训练的BioBERT模型达到了78.1%的f1评分。实验结果表明,我们的方法(IMSE)优于所有最新模型,f1评分比基线模型BioBERT高7.06%,f1评分比最新的DESC_MOL和ChemicalBERT + AGGCN分别高1.56%和2.28%。与其他基于预训练的方法相比,交互注意操作使模型更加关注有助于正确分类结果的信息,即关键信息。实体关注操作使模型更关注药物实体本身,而忽略其他干扰药物。 Molecular structure as additional information can also provide the model with structural features in addition to text.
烧蚀实验
在本节中,为了探索每个组件对整体性能的贡献,我们对我们提出的模型进行了消融研究。我们一共做了6个对比实验,实验结果见表4.如表所示,我们未添加任何操作时BERT(BioBERT)模型的F1-score为78.1%。当我们只添加Entities注意向量时,f1得分比BERT提高了2.79%,这说明添加Entities注意信息有助于确定药物之间的关系。接下来,我们评估了我们提出的交互注意向量的影响。当加入Interaction注意向量时,F1-score较BERT提高了5.66%,达到了83.76%。然后我们在第四次实验中同时加入了Interaction注意向量和Entities注意向量,我们将该方法标记为‘BERT + Int* + Ent*’,从表中可以看出,该方法的精密度得分、回忆得分和f1得分分别达到了85.54%、83.56%和84.47%,充分说明了Interaction注意和Entities注意的信息是可以很好地协调的。在第5个实验(BERT + Int* + Ent* + MOL)中,我们考察了分子结构对模型的影响,我们在第4个实验的基础上增加了分子结构,f1评分比第4个实验提高了0.41%。实验结果表明,该方法是有效的,与现有模型相比,也获得了目前最高的评分(85.16%)。
五倍交叉验证的性能
我们用五重交叉验证进一步探讨了实验结果的稳定性和方法的实用性。从Table可以看出5,在实验结果中,我们提出的交互注意向量对基线模型起到了很强的促进作用。对于我们所关注的四个关系类别,交互注意向量都有助于模型的性能。分子结构在大多数结果中也表现良好,但效果不如交互注意向量。最后,加入所有有用信息后,模型的性能有了很大的提高,实验结果也表明,该方法具有较强的泛化能力和稳定性。
讨论
误差与分析
为了保证结果的公平性,我们对训练集只采用过采样和欠采样处理。虽然这是有效的,但测试集本身的长尾分布是无法解决的,这也是生物数据的一大特点和难点。在我们的测试集中,数据分布非常不均匀。所以,这是影响最终结果的一个重要原因。那么交互注意向量主要是为了更好地提取句子中两个实体之间的信息,有些句子很短,可以提供很少的信息来判断关系。因此,通过交互注意向量所能提高的性能是有限的,不可避免地会出现错误的分类结果。
此外,在从药物名称获取药物结构的过程中,我们首先需要从医学知识库中推导出药物的分子式。在这个过程中,有些药物找不到相应的分子式。另一方面,在SMILES获取分子结构的过程中,我们得到的是一个平面结构或者是错误的信息,而药物的实际分子结构是三维的,这会导致我们失去很多平面结构之外的信息。所有这些误差在转移到模型时会产生很大的影响。
可解释性
IMSE的性能受益于几个主要因素。(1)交互信息包含药物-药物交互的关键信息,交互注意向量的引入提高了特征的准确性。(2)实体信息的引入可以充分提取特征空间中实体的局部信息,有效降低了其他实体对模型的干扰。(3)分子结构的加入有效提高了特征空间的丰富度,提供了文本以外的信息。
结论
在本文中,我们提出了一种基于BioBERT的DDI提取模型,称为IMSE,以提高DDI提取的性能。在我们的模型中,我们使用我们提出的交互注意向量来增强句子中的交互信息,以更好地处理关系重叠问题。我们添加的分子结构信息可以利用文本中无法学到的知识,可以更好地表征药物特征空间。此外,我们还添加了实体注意向量来增强实体周围的权重,使模型能够更好地捕获药物实体周围的信息,而不受其他药物的干扰。在基准数据集上的对比实验表明,IMSE具有比现有预测模型更好的预测性能,提高了DDI的识别能力。模型在烧蚀实验中的表现也说明了模型的每一部分都是不可或缺的,这也为后续的研究提供了新的思路,即我们可以从数据的特征中获得更好的特征表示。
尽管我们提出的方法在生物医学文献中提取DDI方面表现出了很好的性能,但仍有一些改进空间。在未来的工作中,我们将继续探索生物医学数据的特征,并将其与深度学习方法相结合,更好地解决生物医学应用中的问题。
数据和材料的可用性
方法细节和实现流程可以从我们的Github存储库中获得:https://github.com/db-bionlp/IMSE.
缩写
- 有线电视新闻网:
-
卷积神经网络
- RNN:
-
循环神经网络
- LSTM:
-
长短期记忆网络
- 伯特:
-
来自变压器的双向编码器表示
- DDI:
-
药物-药物相互作用
- DDIExtraction:
-
药物-药物相互作用提取
- BioBERT:
-
来自变压器的生物医学双向编码器表示
- 微笑:
-
简化的分子输入线输入规范
- MGNN:
-
分子图神经网络
- NLP:
-
自然语言处理
- 格勒乌:
-
栅极循环装置
参考文献
Kantor ED, Rehm CD, Haas JS, Chan AT, Giovannucci EL。1999-2012年美国成年人处方药使用趋势。《美国医学协会杂志》上。314(17): 1818 - 2015; 30。
张娜,孙德奎斯特J,孙德奎斯特K,季杰。瑞典综合用药的流行趋势:一项基于全国注册的研究。Front Pharmacol, 2020;11:326。
Oktora MP, Denig P, Bos JH, Schuiling-Veninga CC, Hak E.荷兰成年人中多重药房和分发药物的趋势与美国相比。PLoS ONE。2019; 14 (3): e0214240。
Siniscalchi A, Gallelli L, Avenoso T, Squillace A, De Sarro G.卡马西平/羟考酮联合给药治疗三叉神经痛的疗效。中国药物学杂志。2011;45(6):e33-e33。
Franceschi A, Tuccori M, Bocci G, Vannozzi F, Di Paolo A, Barbara C,等。急诊科病人的药物治疗失败:大学医院的经验。药物学杂志2004;49(1):85-91。
Segura Bedmar I, Martínez P, Herrero Zazo M. Semeval-2013任务9:从生物医学文本中提取药物-药物相互作用(DDIExtraction 2013)。斯特劳兹堡:计算语言学协会;2013.
信息提取技术在药理学领域的应用:提取药物-药物相互作用。马德里:马德里卡洛斯三世大学;2010.
杨晓东,王晓明。基于最大频率序列的药物间相互作用检测方法研究[J]. García-Blasco2010.
刘松,唐波,陈强,王霞。基于卷积神经网络的药物-药物相互作用提取。计算数学方法医学2016;2016:6918381。
权春,华林,孙霞,白伟。多通道卷积神经网络在生物关系提取中的应用。BioMed Res Int. 2016;2016:1850404。
刘胜,陈凯,陈强,唐波。基于依赖的卷积神经网络药物-药物相互作用提取。见:IEEE生物信息学和生物医学国际会议(BIBM), 2016卷。IEEE;2016.p . 1074 - 80。
赵智,杨智,罗林,林华,王杰。基于句法卷积神经网络的生物医学文献药物相互作用提取。生物信息学。2016;32(22):3444 - 53。
尹德维,董松,胡娟。基于深度卷积神经网络的药物-药物相互作用关系提取。参加:2017年IEEE生物信息学和生物医学国际会议。IEEE;2017.p . 1795 - 802。
孙鑫,马琳,杜鑫,冯杰,董凯。深度卷积神经网络用于药物-药物相互作用提取。见:2018年IEEE生物信息学和生物医学国际会议(BIBM), 2018卷。IEEE;2018.1662 - 8页。
孙晓明,杨晓明,杨晓明。基于神经网络的药物-药物间相互作用的神经网络研究。In: BioNLP 2017;2017.9到18页。
黄东,蒋震,邹玲,李磊。基于支持向量机和长短期记忆网络的生物医学文献中药物-药物相互作用提取。科学通报2017;415:100-9。
蒋震,顾磊,蒋强。基于骨架长短期记忆神经网络的文献药物相互作用提取。参加:2017年IEEE生物信息学和生物医学国际会议。IEEE;2017.552 - 5页。
王伟,杨旭,杨超,郭旭,张旭,吴晨。基于依赖的长短期记忆网络药物-药物相互作用提取。生物工程学报,2017;18(16):99-109。
郑伟,林华,罗林,赵忠,李忠,张勇,等。基于注意的药物-药物相互作用提取有效神经模型。生物工程学报。2017;18(1):1 - 11。
张勇,郑伟,林浩,王军,杨忠,杜蒙提尔。基于序列和最短依赖路径的分级rnn药物-药物相互作用提取。生物信息学。2018;34(5):828 - 35。
易志,李松,余杰,谭勇,吴强,袁辉,等。多注意层递归神经网络药物-药物相互作用提取。进:高级数据挖掘及应用国际会议。施普林格;2017.p . 554 - 66。
周东,苗林,何勇。位置感知深度多任务学习的药物-药物相互作用提取。Artif Intell Med. 2018; 87:1-8。
Collobert R, Weston J, Bottou L, Karlen M, Kavukcuoglu K, Kuksa P.自然语言处理(几乎从头开始)。中国机械工程学报,2011;12(ARTICLE): 2493-537。
Sutskever I, Vinyals O, Le QV。用神经网络进行序列对序列学习。在:神经信息处理系统的进展;2014.3104 - 12页。
帕斯卡努R, Mikolov T, Bengio Y.关于训练递归神经网络的难度。正确的做法:机器学习国际会议。PMLR;2013.1310 - 8页。
张志刚,张志刚。长短期记忆。神经计算。1997;9(8):1735-80。
冯志刚,李志刚,李志刚。基于LSTM的连续预测方法研究。神经网络学报,2000;12(10):2451-71。
赵k, Van Merriënboer B, Bahdanau D, Bengio Y.关于神经机器翻译的性质:编码器-解码器方法。arXiv预印本arXiv: 1409.1259(2014)。
Devlin J, Chang MW, Lee K, Toutanova K. Bert:基于深度双向转换器的语言理解预训练。arXiv预印本arXiv: 1810.04805(2018)。
张志刚,张志刚,张志刚,等。你所需要的就是关注。arXiv预印本arXiv: 1706.03762(2017)。
李俊,尹伟,金松,金丁,金松,苏超,等。BioBERT:用于生物医学文本挖掘的预训练生物医学语言表示模型。生物信息学。2020;36(4):1234 - 40。
Boukkouri HE, Ferret O, Lavergne T, Noji H, Zweigenbaum P, Tsujii J. CharacterBERT:调和ELMo和BERT用于字符的词级开放词汇表。arXiv预印本arXiv: 2010.10392(2020)。
孙超,杨震,苏玲,王玲,张勇,林华,等。基于高斯概率分布和外部生物医学知识的化学蛋白相互作用提取。生物信息学。2020;36(15):4323 - 30。
王志强,郭志强,王志强,等。DrugBank 5.0: 2018年对DrugBank数据库的重大更新。核酸研究,2018;46(D1): D1074-82。
它。RDKit:开源化学信息学。释放2014.03.1。2010.
张晓明,李志强,张晓明。基于神经网络的蛋白质相互作用预测方法研究。生物信息学,2019,35(2):309 - 18。
林山,李凯,姜杰。基于递归神经网络的药物相互作用提取方法。PLoS ONE。2018; 13 (1): e0190926。
秦玲,董刚,彭娟。基于化学albert和注意力引导图卷积网络的化学-蛋白质相互作用提取。参加:2020年IEEE生物信息学和生物医学国际会议(BIBM)。IEEE;2020.708 - 15页。
浅田浩,杨晓明,杨晓明。基于药物描述和分子结构的药物-药物相互作用提取方法。生物信息学。2021;37(12):1739 - 46。
确认
不适用。
关于这个补充剂
本文已作为BMC生物信息学第23卷第7补编的一部分发表,第20届亚太生物信息学会议(APBC 2022):生物信息学。该补充的全部内容可在网上查阅https://bmcbioinformatics.biomedcentral.com/articles/supplements/volume-23-supplement-7.
资金
不适用。
作者信息
作者及隶属关系
贡献
BD参与了处理和分析工作流程、算法和模型的开发。JP分析和解释了数据。BD起草了手稿。JP和YZ对手稿进行了监督和编辑。所有作者都赞同最后的文章。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
段斌,彭俊,张艳。基于药物相互作用信息关注和分子结构的药物相互作用提取。BMC生物信息学23(增刊7)338(2022)。https://doi.org/10.1186/s12859-022-04876-8
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04876-8
关键字
- 药物之间相互作用
- 方防治效果
- 挖掘分子结构