摘要
背景
必需蛋白对细胞的发育和生存是必不可少的。必需蛋白的鉴定不仅有助于了解细胞生存的最低要求,而且在疾病诊断、药物设计和医疗方面具有实际意义。随着蛋白质相互作用(PPI)数据的快速积累,从蛋白质相互作用网络(PINs)中计算识别必需蛋白质越来越受欢迎。到目前为止,基于pin的多种必需蛋白鉴定方法已经被开发出来。
结果
在本文中,我们提出了一种新的有效方法,称为iMEPP,通过融合多种类型的生物学数据,并将影响最大化机制应用于pin,从pin中识别必需蛋白。具体来说,我们首先整合PPI数据、基因表达数据和基因本体构建加权pin,以缓解原始PPI数据高假阳性的影响。然后,我们定义影响成绩包含正交数据和PIN拓扑信息的PIN节点。最后,我们开发了一种基于影响最大化机制的影响折扣算法来识别必需蛋白。
结论
我们将我们的方法应用于从酿酒酵母销。实验结果表明,本文提出的iMEPP方法优于现有方法,验证了该方法的有效性和优越性。
背景
蛋白质(1,2]是细胞重要的结构和功能组成部分,它们在生物体中发挥着许多重要的功能,包括载体运输、抗体免疫、激素调节等。其中,必需蛋白是指细胞发育和生存所必需的蛋白质。致病基因与必需蛋白密切相关。因此,必需蛋白的鉴定不仅有助于了解细胞生存的最低要求,而且对病原生物学的研究具有重要的现实意义[3.]和药物设计[4].
湿实验室实验首次用于识别必需蛋白,包括单基因敲除[5], RNA干扰和反义RNA [6)等。虽然这些方法非常准确,但它们既昂贵又耗时。随着高通量实验技术的快速发展,获取大量蛋白质相互作用(protein-protein interaction, PPI)数据非常方便。这启发了计算方法的发展[7,8,9来识别必需蛋白质。现有的大多数计算方法都是基于PPI网络(pin),它是PPI数据的图形表示。一个PIN可以被建模为一个图形G(E,V),V节点集合是否代表蛋白质,和E是表示蛋白质之间相互作用的边的集合。从图论的角度来看,必需蛋白可以被视为PIN中的重要或关键节点。因此,关键的蛋白质识别转向了寻找PIN中的重要节点。
郑等人。[10]建议centrality-lethality规则,这表明必需蛋白质往往比其他蛋白质对细胞的生存更重要。因此,必需蛋白的缺失比其他蛋白的缺失更致命。基于centrality-lethality规则,提出了各种中心性测量方法来识别必需蛋白,包括度中心性(DC) [10]、居间中心性(BC) [11],亲近中心性[12,子图中心性(SC) [13]),特征向量中心性(EC) [14)等。
在此基础上,还提出了利用pin的深层拓扑信息来识别pin中的必需蛋白质的更复杂的指标,这可以获得比基于中心性的方法更好的性能。此外,考虑到pin的高假阳性,一些方法使用额外的生物数据来提高性能。Li等人提出了PeC [15结合基因表达数据和pin的拓扑信息。Zhang等人开发了CoEWc [16该方法利用基因表达数据的局部聚类系数和皮尔逊相关系数(PCC)。后来,Zhang等人介绍了TEO [17]方法整合基因表达数据、基因本体(GO)和矫形学数据进行必需蛋白鉴定。最近,Xu等人。[9]提出了一种基于随机游走的方法EssRank,该方法利用基因表达数据、功能注释、结构域相互作用和系统发育谱来提高pin的质量,从而获得更好的识别精度。
在本文中,受社交网络中病毒式营销的影响最大化(IM)机制的启发,我们提出了一种新的方法,称为iMEPP,从pin中识别必要蛋白质。一方面,我们利用PPI数据、基因表达数据和GO构建加权pin,以减少原始PPI数据高假阳性的影响。另一方面,我们将社交网络中的IM机制应用于基本蛋白质识别问题。为此,我们定义影响成绩(IS),并开发了一种影响折扣(ID)算法来识别PIN中的必需蛋白。我们的实验酿酒酵母数据表明,本文提出的iMEPP方法能取得比现有方法更好的性能。
结果
在本节中,我们首先介绍的PPI数据和基因表达数据酿酒酵母.然后给出实验设置。最后,报告了实验结果。
数据集
PPI数据和基因表达数据酿酒酵母都用于我们的实验。PPI数据来自BioGRID数据库[18],包括4860个蛋白质和22138个蛋白质之间的相互作用。必需蛋白质数据来自SGD [19], deg [20.]及SGDP [21]数据库,总共1194种必需蛋白质。骨科数据来自InParanoid(版本7)数据库[22],包含100个基因组,其中99个是真核生物,1个是原核生物。
实验设置
\λ(\ \)是一个权衡参数,以平衡拓扑学和正交学的贡献。当\ \(λ= 0 \),必需蛋白的鉴定完全取决于PIN拓扑结构的影响;如果\ \(λ= 1 \),仅由蛋白质矫形学测定。通过设置\(p = 0.001\)[23的值\λ(\ \)到0,0.1,0.2,…,1分别we check the number of essential proteins correctly identified by our method.
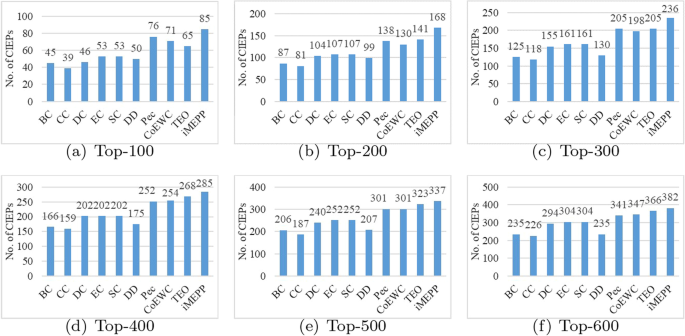
为了展示我们的方法的优势,我们将其与现有的几种方法进行了比较,包括五种基于中心性的方法(BC [11], cc [12], dc [10]和EC [14], sc [13])、三种综合多种生物信息的方法(PeC [15], CoEWc [16]和TEO [17])。此外,我们还实现了另一种影响最大化算法程度折扣[24)作比较。我们让每个方法输出top-k(k从100到1000)个必需蛋白质候选中选出,从中我们计算出正确识别的蛋白质的数量。
实验结果
的影响\λ(\ \)
表格1给出正确识别的不同蛋白质的数量\λ(\ \)而且k值。我们设置k从100到600,每个都有k价值,我们增加\λ(\ \)从0到1.0。从表1,我们可以看到,给定k价值,既不\ \(λ= 0 \)也不\ \λ= 1.0 (\)才能得到最好的结果。这意味着将PIN拓扑结构与蛋白质矫形学相结合有利于必需蛋白的鉴定。当\λ(\ \)在0.2到0.5之间,我们可以得到更好的结果。这表明在必需蛋白的鉴定中,PIN拓扑结构比蛋白质正交结构更为重要。此外,在大多数情况下,当我们得到最好的结果\ \λ= 0.2 (\),所以在剩下的实验中我们设置\ \λ= 0.2 (\)在我们的方法中。
与现有方法的比较
首先,我们分别检查前100、200、300、400、500、600个输出候选蛋白,并计算正确识别的必需蛋白的相应数量。对比结果如图所示。1.我们可以看到,我们的方法比其他方法可以正确识别更多的必需蛋白质。
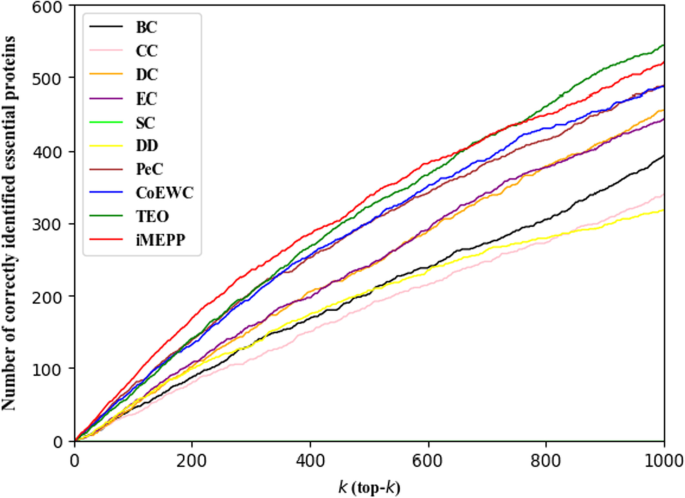
数字2说明了比较结果的大范围k取值范围:top-1 ~ top-1000。我们可以看到\(k < 667\),我们的方法明显优于其他方法。当k为[667,764]时,我们的方法与TEO的性能相似。然而,当\(k > 764\), TEO优于我们的方法,我们的方法在这些方法中排名第二。
讨论
基于PIN的计算方法在必需蛋白的鉴定中取得了很大的成功。由于pin与社会网络在拓扑性质上的相似性,将社会网络的IM机制应用于pin,进而提出了iMEPP方法来识别必需蛋白。首先,收集PPI数据、基因表达数据和GO构建加权pin。然后,利用PIN拓扑和蛋白质正交学,计算每个蛋白质的IS,量化其为必需蛋白的概率。最后,设计了一种ID算法,以迭代的方式逐个枚举候选必需蛋白。虽然实验结果酿酒酵母数据集显示了iMEPP方法的有效性,以及它相对于现有计算方法的优势,该方法仍有一些可能的改进之处。一方面,在iMEPP中,每次迭代只识别出一个必要的候选蛋白,并且全部k迭代是为了挖掘所有内容k必需的蛋白质候选人。换句话说,就是时间复杂度\ (O (k V * | | + | | E) \)和数字有关吗k的迭代。可以通过在每次迭代中选择一个以上的必需候选蛋白来减少迭代次数。因此,我们可以在保持其性能的同时加快方法的速度。另一方面,在社交网络领域,已经有许多影响最大化的算法,我们正在考虑采用更先进的IM方法来促进从pin码识别必需蛋白。此外,我们将把iMEPP应用于其他物种的PIN数据,以鉴定必需蛋白,以证明其适用性。
结论
本文介绍了一种基于IM的从pin中识别必需蛋白的新方法,该方法最初用于社交网络的病毒式营销。为此,我们定义了具有矫形学数据和PIN拓扑信息的PIN中节点的影响评分,并设计了一种影响折扣算法来识别PIN中的必要蛋白质。此外,我们将PPI数据、基因表达数据和GO结合起来构建加权pin,可以有效提高pin的质量。实验结果表明,iMEPP方法优于现有方法,证明了其有效性和优越性。
方法
在本节中,我们将介绍从pin中鉴定必需蛋白的iMEPP方法。首先,我们介绍了IM的基本概念,然后对iMEPP方法进行了概述。接着,我们给出了所提出方法的技术细节。最后给出了算法和复杂度分析。
预赛
即时通讯是社交网络中一个重要且被广泛研究的算法问题,最初是由病毒式营销驱动的[25].从本质上讲,它是从一个社会网络中选择少量的种子节点,这样被选择的节点可以将其影响力传播到网络中尽可能多的其他节点。到目前为止,针对IM问题已经提出了大量的算法,如贪婪算法[23]和DD算法[24)等。
影响最大化的定义
社交网络可以被建模为一个加权图\ (G = (V, E) \),在那里V个体(用户)的集合是否被视为节点,E是被视为边的个体(用户)之间的连接集,每条边都与一个权重相关联。基于随机级联模型,影响在网络中扩散。级联模型有三种类型:1)独立级联模型[23], 2)线性阈值级联模型,3)加权级联模型。
考虑到社交网络\ (G = (V, E) \),一个影响级联模型和一个数字k的节点,IM的问题是找到k节点受网络中这样的节点预期数量的影响k根据影响级联模型,所选节点尽可能大。在这里,k节点被视为k种子,以及受节点数影响的期望k节点被视为影响扩散。
度折现算法
在这里,我们简单介绍了度折扣(DD)算法,它是一种典型的IM算法,将在本文中使用。一般有些贪心算法直接用度来表示节点的影响,倾向于选择度最大的节点。与这些贪婪算法不同,DD算法将在每次迭代中通过折扣重新计算新种子节点的邻居度。
给定已经选定的种子节点集,以便从图中找到一个新的种子节点G的子图G没有种子集和与种子相关的边,然后重新计算子图中节点的度。注意,对于这些不是种子邻居的节点,它们的度保持不变。也就是说,我们只重新计算相邻种子的度。假设u是种子节点和v的邻居u在子图中。我们折现的程度v直观上是1。实际上,学位折扣并不是这么简单的。相反,它依赖于影响扩散模型,并被建模为一个优化问题。
iMEPP方法概述
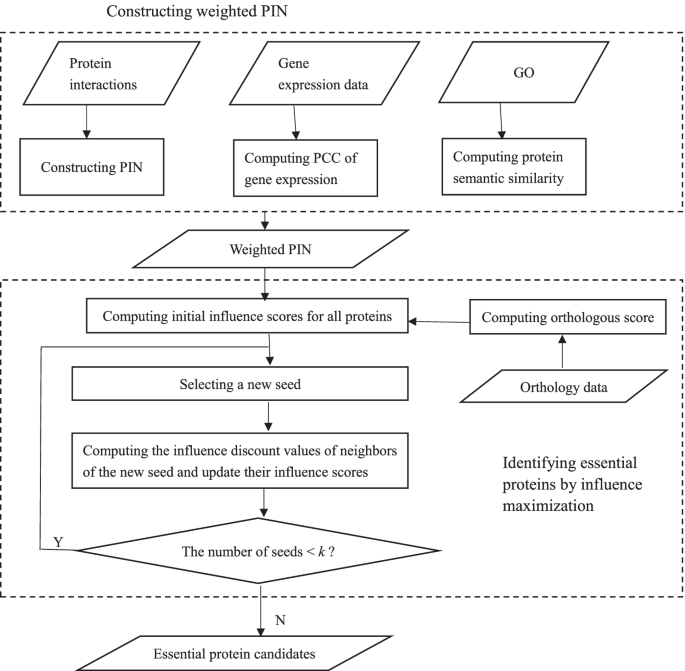
数字3.显示了iMEPP方法的工作流程。它由两个主要模块组成:加权PIN构建(顶部为虚线矩形)和IM必需蛋白识别(底部为虚线矩形)。
为了构建加权PIN,我们使用PPI数据、基因表达数据和GO。用基因表达的PCC对PIN边进行加权GO语义相似性.
为了通过IM识别必需蛋白质,我们首先计算PIN中所有蛋白质的初始IS。每个蛋白质的初始IS值由两部分组成:一部分来自它的正交信息,另一部分来自它连接边的权重。然后,我们以迭代的方式逐一列举必要的候选蛋白。在每次迭代中,有三个主要步骤:
- 1.
选择一个新的种子\ (s_{新}\)当前剩余蛋白质的IS值最大(不包括种子集中的节点)
- 2.
计算影响折扣的非种子邻居的ID\ (s_{新}\),并更新其IS值
- 3.
检查所选种子的数量是否达到理想值(例如k).如果没有,请进行下一次迭代;否则,迭代结束,所有选择的种子输出为必需蛋白候选。
在接下来的小节中,我们将介绍通过IM识别必需候选蛋白的过程的技术细节。
技术细节
给出原始PIN码G(V,E),基因表达数据,GO和orthology数据,我们首先描述了如何构建加权PIN,然后介绍了如何评估网络中蛋白质的IS和ID。
加权PIN结构
为了提高pin的质量,从而提高必需蛋白的识别精度,我们用基因表达数据和GO构建了加权pin。给定两种蛋白质u而且v,它们对应的基因表达谱\ (p_u \)而且\ (p_v \),我们使用Pearson相关系数(PCC) [26的基因共表达水平u而且v如下:
在哪里米为基因表达谱的采样点数,\ (p_{你}(i) \)而且\ (p_ {v} (i) \)的基因表达水平我-蛋白质的采样点u而且v分别酒吧\ (\ p{} _{你}\)而且酒吧\ (\ p {} _ {v} \)是对应的表达水平平均值,\ \(σ_{你}\)而且\ \(σ_ {v} \)是对应的标准差。
然后我们计算语义相似度两种蛋白质u而且v通过去。一个蛋白质通常是由几个GO术语来注释的语义相似度蛋白质之间u而且v的计算方法为
在哪里u而且v由米去上\(\{t_{u}^{i}|i=1, \ldots, m\}\)而且n去上\(\{t_{v}^{j}|j=1, \ldots, n\}\)分别。\ (Sim_{去}\)(t,P)是语义相似度GO项之间t和蛋白质P带注释的k术语:
以上,语义相似度两个GO项的\ (t_1 \)而且\ (t_2 \)如下:
在哪里\ (T_ {T_{1}}识别识别\)(或\ (T_ {t_2} \识别))是GO项的祖先GO项的集合\ (t_{1} \识别)(或\ (t_2 \))和自身,以及\ (S_ {t_{1}}识别(t) \)(或\ \ (S_ {t_2} (t)))是年代值(27GO项的]t有关\ (t_{1} \识别)(或\ (t_{2} \识别)).
的重量边的连接u而且v计算为
用来测量PIN中两个蛋白质的结合程度。
影响评分(IS)
一个节点在网络中的影响力意味着它在网络中的重要性。在我们的场景中,蛋白质的IS表示它是必需蛋白质的概率。我们从两个角度考虑这个问题:PIN拓扑结构和蛋白质矫形学。
从PIN拓扑来看,蛋白质的ISu如下:
在哪里\ (Inf_{威尼斯平底渔船}(u) \)=\(\sum _{v \in N_{u}} w(u,v)\),\ (N_{你}\)的邻域集合是什么u.
从蛋白质的同源性来看,必需蛋白通常比非必需蛋白具有更多的同源性。所以同源评分(OS) [28]可以用来测量蛋白质的重要性。对蛋白质u,操作系统(u) =\ (n_{你}\)/N在哪里\ (n_{你}\)蛋白质的种类有多少u有骨科和N是参考物种的总数。实际上,我们使用归一化OS从orthology的角度来测量蛋白质的IS。也就是说,
结合\ (IS_{威尼斯平底渔船}\)而且\ (IS_ {OS} \),即蛋白质的ISu计算如下:
在哪里\λ(\ \)是[0,1]中的一个权衡参数,以平衡拓扑和正交学的贡献。
影响折扣(ID)
当一个蛋白质被选为种子时,这个新种子的邻居的影响将被贴现并更新。注意:1)折扣只对is的拓扑部分进行,因为只有这部分与蛋白质之间的相互作用有关。2)折扣操作依赖于所采用的影响扩散模型。在这里,我们使用独立级联模型。3)在每次迭代中,对一个蛋白质的贴现操作独立于之前迭代中对它的贴现操作,它考虑了到当前迭代为止的所有种子邻居。我们给出下面的定理来说明如何计算蛋白质的ID。
定理1
给定的蛋白质v,N(v)是它的邻居集合,t(v的种子节点数N(v),tt(v的和权重边的连接v种子结进去N(v),明星(v的所有节点组成的子图N(v)和连接到的边v.在具有扩散概率的独立级联模型下p,假设下列方程成立:
的影响折扣v,表示为ID(v),为节点影响的期望值v,由之间的拓扑信息导出v而非种子节点明星(v).在形式上,
证明
的节点v不受任何种子节点的影响N(v)概率\ ((1 - p) ^ {t (v)} \).在扩散概率下p,节点的影响值v由之间的权重生成v而非种子节点明星(v)是\((Inf_{topo}(v) - tt(v)) * p\).因此,节点的IDv是\((1-p)^{t(v)} * (Inf_{topo}(v) - tt(v)) * p\).它推导出
以上,第二个等式由于方程的存在而有效\(t(v)* p = o(1)\)时,由于方程,第三个等式成立\ (Inf_{威尼斯平底渔船}(v) - tt (v)) * p = O (Inf_{威尼斯平底渔船}(v) * p) = O (1) \),最后一个等式是有效的,因为方程\(t(v) = o(1/p)\.\广场(\ \)
注意,我们可以保证Eq. (9)通过设置一个小的值来保持p在实验。根据定理1,我们得出蛋白质的ISv在拓扑中更新如下:
算法
算法1概述了iMEPP的过程。第1行初始化必需候选蛋白集和参数。第2-8行计算PIN中所有蛋白质的初始IS值,其中第3-5行计算任意两个相互作用蛋白质之间的权重。第9行得到最大值\ (Inf_{威尼斯平底渔船}\).第10-19行描述了选择种子的迭代过程:第11行选择了一个新的种子\ (s_{新}\)对于最大的IS,第12行更新种子集,第13-18行用于计算的非种子邻居的ID值\ (s_{新}\),并更新他们的IS值。第20行返回作为必需蛋白质候选的种子集。

复杂性分析
iMEPP的时间复杂度由两部分组成。第一部分是初始值的计算是一个PIN中所有蛋白质的值,这完全由边的数量决定。因此,该部分的时间复杂度为O(|E|)。第二部分是关于种子选择的迭代过程。每次迭代的时间复杂度为\ (O日志V | |) (\ \).因此,第二部分的时间复杂度为\(O(k * \log |V|)\).综上所述,iMEPP的复杂性为\(O(k * \log |V| + |E|)\).
数据和材料的可用性
本研究中使用和/或分析的数据集可在相应的文章中获得。iMEPP的源代码和数据可在https://github.com/xuweixia88/iMEPP.git.
缩写
- PPI:
-
蛋白质相互作用
- 销:
-
蛋白质-蛋白质相互作用网络
- 走:
-
基因本体论
- iMEPP:
-
基本蛋白质预测的影响最大化
- RNA:
-
核糖核酸
- DC:
-
学位中心
- 公元前:
-
中间性中心
- 答:
-
亲密关系中心
- SC:
-
子图中心
- 电子商务:
-
特征向量中心
- PCC:
-
皮尔逊相关系数
- BioGRID:
-
交互数据集生物通用存储库
- SGD:
-
酵母基因组数据库
- 度:
-
必需基因数据库
- 弟弟:
-
学位的折扣
- 即时通讯:
-
影响最大化
- 是:
-
影响得分
- ID:
-
影响折扣
- 操作系统:
-
直系同源的分数
参考文献
Branden CI, Tooze J.蛋白质结构导论。纽约:嘉兰科学;2012.
王志强,王志强,王志强。基于结构模型的蛋白质- dna结合特异性预测。中国生物医学工程学报,2005;33(18):5781-98。
Furney SJ, Albà MM, López-Bigas N.显性或隐性突变对疾病基因进化历史影响的差异。BMC基因组学杂志,2006;7(1):165。
Clatworthy AE, Pierson E, Hung DT。靶向毒性:抗菌治疗的新范式。中国生物化学杂志,2007;3(9):541-8。
小林K, Ehrlich SD, Albertini A,等。枯草芽孢杆菌必需基因。自然科学进展,2003;21(3):344 - 344。
季艳,张波,范顺丰,杨晓明,杨晓明,杨晓明。葡萄球菌关键基因的反义RNA条件表型鉴定。科学。2001;293(5538):2266 - 9。
雷霞,赵娟,藤田宏,张安。基于rna序列、亚细胞定位和go注释数据集的必需蛋白预测。系统工程理论与实践。2018;
李敏,李伟,吴峰,潘勇,王杰。基于亚细胞定位信息的亚网络划分和优先级识别必需蛋白。中国生物医学杂志,2018;447:65-73。
许斌,关健,王勇,王哲。基于加权蛋白相互作用网络的随机游走必需蛋白检测。中国生物医学工程学报。2019;16(2):377-87。
郑浩SP, Barabási AL;蛋白质网络中的致死率和中心性。大自然。2001;411(6833):41-2。
黄晓明,王晓明,王晓明,黄晓明。酵母蛋白相互作用网络中高中介蛋白的研究进展。生物科学通报,2005;(2):96-103。
吴奇提S, Stadler PF.复杂网络中心。中国生物医学杂志,2003;223(1):45-53。
Estrada E, Rodriguez-Velazquez JA。复杂网络中的子图中心性。物理学报,2005;21(5):567 - 567。
权力与中心:一系列措施。中国社会科学学报。1987;32(5):366 - 366。
李敏,张宏,王娟,潘艳。一种基于蛋白质相互作用与基因表达数据整合的必需蛋白发现新方法。中国生物医学工程学报。2012;6(1):15。
张旭,徐娟,肖伟。一种发现必需蛋白的新方法。PLoS ONE。2013; 8(3): 58763。
张伟,徐娟,李勇,邹霞。基于网络拓扑、基因表达数据和基因本体信息的必需蛋白检测。中国生物医学工程学报。2018;15(1):109-16。
Stark, C., Breitkreutz, b.j., rely, T., Boucher, L., Breitkreutz, A., Tyers, M.:生物网格:交互数据集的通用存储库。核酸Res. 34(suppl_1), 535-539 (2006)
Cherry, j.m., Hong, e.l., Amundsen, C., Balakrishnan, R., Binkley, G., Chan, e.t., Christie, K.R, Costanzo, m.c., Dwight, s.s., Engel, S.R:酵母菌基因组数据库:出芽酵母基因组资源。核酸Res. 40(数据库issue), 700-705 (2012)
Luo H., Lin Y., Gao F., Zhang C., Zhang R.: Deg 10,基本基因数据库的更新,包括蛋白质编码基因和非编码基因组元素。核酸Res. 42(数据库issue), 574-580 (2014)
温泽勒,e.a.,舒梅克,d.d.,阿斯特罗莫夫,梁,H.,安德森,K.,安德烈,B.,邦汉,R.,本尼托,R.,勃伊克,J.D.,布西,H.,朱,a.m.,康奈利,C.,戴维斯,K.,迪特里希,F.,道,s.w.,巴克库里,m.e.,福里,弗兰德,s.h.,根塔伦,E.,吉埃弗,G.,海格曼,J.H.,琼斯,T.,劳布,M.,廖氏,H.,李邦古特,N.,洛克哈特,d.j.,卢考-达尼拉,A.,卢西尔,M., M.,莱贝特,N.,梅纳德,P.,米特曼,M.,派,C.,雷比松,C.,雷维埃塔,j .,赖尔斯,L.,罗伯茨,c.j.,罗斯-麦克唐纳,P.,Scherens, B., Snyder, M., Mahadeo, s.s., Storms, r.k., Véronneau, S., Voet, M., Volckaert, G., Ward, t.r., Wysocki, R., Yen, g.s., Yu, K., Zimmermann, K., Philippsen, P., Johnston, M., Davis, r.w.:通过基因缺失和并行分析对啤酒酵母基因组的功能描述。科学285(5429),901-906 (1999)
Östlund, G., Schmitt, T., Forslund, K., Köstler, T., Messina, D.N., Roopra, S., Frings, O., Sonnhammer, E.L.: Inparanoid 7:真核矫形学分析的新算法和工具。核酸Res. 38(suppl_1), 196-203 (2010)
肯普,D.,克莱因伯格,J.,塔尔多斯,É。:Maximizing the spread of influence through a social network. In: Proceedings of the Ninth ACM SIGKDD international conference on knowledge discovery and data mining, pp. 137–146 (2003)
陈伟,王勇,杨生:社会网络中有效的影响力最大化。见:第15届ACM SIGKDD知识发现和数据挖掘国际会议论文集,pp. 199-208 (2009)
多明戈斯:挖掘客户的网络价值。见:第七届ACM SIGKDD知识发现和数据挖掘国际会议论文集,第57-66页(2001年)
王晓明,王晓明,王晓明,等。标准化实验室间和跨平台的全球基因表达分析。光子学报。2005;2(5):351-6。
王建泽,杜震,Payattakool R,余鹏,陈cf .一种新的围棋词语义相似度度量方法。生物信息学。2007;23(10):1274 - 81。
李,G。,,,,,,,,F。,,Y:基本预测蛋白质亚细胞定位的基础上,orthology和PPI网络。BMC Bioinform. 17(Suppl_8), 279 (2016)
确认
不适用。
关于这个补充剂
本文已作为BMC生物信息学第23卷补编8的一部分发表,2022:第16届生物信息学研究与应用国际研讨会(ISBRA-20):生物信息学的选文。该补充的全部内容可在网上查阅https://bmcbioinformatics.biomedcentral.com/articles/supplements/volume23-supplement-8.
资金
WX、YD和SZ项目由国家自然科学基金项目(No. 61972100)和国家重点研发计划项目(No. 2016YFC0901704)资助。国家自然科学基金项目(No. 62172300)资助。NSFC资助了研究的设计,以及数据的分析和解释;中国国家重点研发计划资助了数据的收集和手稿的撰写。国家自然科学基金资助(61972100)。
作者信息
作者及隶属关系
贡献
SZ构思了这项研究,提出了最初的想法并修改了手稿。WX对实验结果进行分析并起草论文。YD准备了数据,实现了算法并进行了实验。JG参与了数据分析和论文修改。所有作者均已阅读并批准最终稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
徐伟,董勇,管俊,董勇。et al。基于影响最大化的蛋白质-蛋白质相互作用网络中必需蛋白质的识别。BMC生物信息学23(增刊8),339(2022)。https://doi.org/10.1186/s12859-022-04874-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04874-w
关键字
- 蛋白质-蛋白质相互作用网络
- 至关重要的蛋白质
- 影响最大化
- 影响折扣