摘要
背景
蛋白质琥珀酰化是近年来出现在赖氨酸残基上的一种重要而常见的翻译后修饰(PTM)。琥珀酰化在其大小(例如,在100 Da时,它是较大的化学PTMs之一)和在生理ph下将修饰过的赖氨酸残基的净电荷从+ 1修改为- 1的能力方面都是值得注意的。琥珀酰化后蛋白质中发生的总体局部变化已被证明与基因活性的变化相对应,并受到柠檬酸循环缺陷的干扰。这些观察结果,以及琥珀酸盐在细胞呼吸过程中作为代谢中间体产生的事实,导致了蛋白质琥珀酸化可能在细胞代谢和重要细胞功能之间的相互作用中发挥作用。例如,琥珀酰化可能代表基因组调控和修复的一个重要方面,并可能在许多疾病状态的病因学中产生重要后果。在这项研究中,我们开发了DeepSuccinylSite,这是一种新型的预测工具,它使用深度学习方法以及嵌入来根据蛋白质的主要结构识别琥珀酰化位点。
结果
使用实验确定的琥珀酰化位点的独立测试集,我们的方法在敏感性、特异性和MCC方面分别获得了79%、68.7%和0.48的效率分数,接受者操作特征(ROC)曲线下的面积为0.8。在与之前描述的琥珀酰化预测因子的并列比较中,DeepSuccinylSite在琥珀酰化位点预测的总体准确性方面有了显著提高。
结论
总之,这些结果表明,我们的方法代表了一种强大的和补充的技术,用于蛋白质琥珀酰化的高级探索。
背景
蛋白质翻译后修饰(PTM)是蛋白质合成后发生的重要细胞调节过程。PTMs通过将功能部分共价添加到蛋白质上,调节亚基的蛋白水解裂解来增加蛋白质组的功能多样性,并在整个蛋白质降解的信号传导中发挥重要作用。PTMs包括磷酸化、糖基化、泛素化和最近描述的修饰,如琥珀酰化。琥珀酰化是一种PTM,通过添加琥珀酰基(−CO-CH)而发生2ch2有限公司2H)到目标赖氨酸残基的ε-氨基。
蛋白质PTMs已被多种实验技术检测到[1],包括质谱分析[2,3.],液相色谱法[4]、放射性化学标签[5]和免疫检测,如染色质免疫沉淀[6]和western blotting [7]。通常,PTMs的实验分析需要耗时、劳动和资本密集的技术,并使用危险/昂贵的化学试剂。Due to importance of PTMs in both disease states and normal biological functions, it is imperative to invest in developing options that can screen proteins for potential PTM sites in a rapid, cost-effective manner.
近年来,机器学习已经成为一种具有成本效益的方法来预测不同的PTM站点。一些基于机器学习的琥珀酰化位点预测方法有iSuc-PseAAC [8], iSuc-PseOpt [9], psu - lys [10],琥珀酸[11], SuccineSite2.0 [12], GPSuc [13]及psuccess [14]。尽管结果很有希望,但由于人工选择特征以及可能缺乏有助于琥珀酰化的未知特征,存在偏差的可能性。此外,这些方法的预测性能还不够令人满意,不能用于高通量的研究。
最近,深度学习(DL)方法已被开发用于阐明细胞蛋白中假定的PTM位点。例如,MusiteDeep [15]和DeepPhos [16]已被开发用于预测磷酸化位点,而Fu等人。[17]和吴等人。[18]使用基于dl的方法分别识别假定的泛素化和乙酰化位点。这些DL方法在综合度量方法性能方面,如曲线下面积(AUC)和马修斯相关系数(MCC)方面取得了相对的改进。通常,这些模型利用单热编码和提取的特征的某种组合作为输入,在很大程度上试图避免对手动特征提取的依赖。据我们所知,DL模型以前还没有应用于琥珀酰化位点的预测。In this study, we developed a succinylation site predictor, termed DeepSuccinylSite, based on a convolutional neural network (CNN) deep learning framework [19] using Keras library [20.]。
方法
基准数据集
在本研究中,我们使用了与Hasan等人从实验衍生的赖氨酸琥珀酰化位点收集的相同训练和独立数据集。[13]和宁等人。[14]。Ning等人像Hasan等人一样,使用UniProtKB/Swiss-Prot数据库和NCBI蛋白序列数据库创建琥珀酰化数据集。用CD-HIT去除序列同一性超过30%的蛋白质后,剩下5009个琥珀酰化位点和53542个未知琥珀酰化位点。其中,4755个琥珀酰化位点和50565个非琥珀酰化位点用于训练集,254个琥珀酰化位点和2977个非琥珀酰化位点用于独立测试。此外,对于我们的方法,最佳窗口大小是33,一些序列有其他字符,我们在训练集中损失了5个(4755个)阳性位点。
对于训练集和测试集,使用欠抽样来平衡数据。最终的训练数据集包含4750个阳性站点和4750个阴性站点,而独立测试数据集在平衡后包含254个阳性站点和254个阴性站点。表格1显示平衡后用于训练和独立测试的最终数据集。为了生成蛋白质的局部表示并优化模型,在每个感兴趣的赖氨酸(K)周围设置了一个窗口参数。如果K的左侧或右侧小于窗口大小的一半,则使用伪残数“-”以保留所有的正位点。
编码
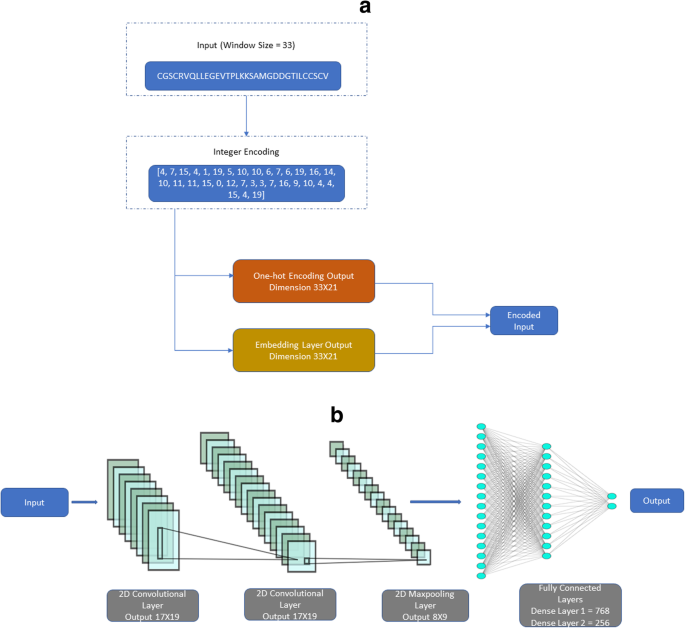
与传统的机器学习方法相比,我们基于dl的方法将窗口形式的序列数据直接作为输入,减少了手工特征提取的需要。这种方法的先决条件是序列数据必须以DL模型可读的形式进行编码。因此,我们使用了两种编码类型:(i)单热编码和(ii)嵌入层。与其他用于其他类型翻译后修饰位点预测的DL方法相比,主要的区别之一是我们的嵌入编码。
在一个炎热的编码
一种热编码将分类变量转换为各自的二进制变量。我们以类似于MusiteDeep开发过程中使用的方式实现了单热编码[15]。为了将20种常见氨基酸和伪残基“-”转换为数值,将这21个字符转换为0到20的整数。每个氨基酸都由一个二进制代码表示,由一串零和一个单数组成,其位置编码了氨基酸的身份。在我们的研究中,二进制表示是基于字母顺序。例如,丙氨酸(A)表示为100000000000000000000,精氨酸(R)表示为010000000000000000000,以此类推。相应地,在我们的模型中,大小为N的窗口对应于大小为N × 21的输入向量。
单热编码的主要缺点之一是映射是完全统一的。因此,性质相似的氨基酸不在向量空间中放置在一起。
嵌入层
我们的方法的亮点之一是嵌入层。我们使用的第二种编码是嵌入编码[20.,21]。嵌入找到了氨基酸序列的最佳表示,就像DeepGO [22],以克服单热编码的缺点。简单地说,首先将20个氨基酸残基和1个伪残基转换为0 ~ 20的整数。这是作为嵌入层的输入提供的,嵌入层位于DL体系结构的开始。嵌入层用随机权重初始化。然后,该层在训练期间通过后续的epoch学习更好的基于向量的表示。每个向量化都是另一个维度的正交表示,因此保持了它的同一性。因此,使其比静态的单一热编码更具动态性。在我们的研究中,K在训练后的21维向量空间中嵌入编码(word to vec)为:[−0.03372079,0.01156038,−0.00370798,0.0072626882,−0.00622324,0.01516087,0.02321764,0.00389882,−0.01039953,−0.02650939,0.01174229,−0.0204078,−0.06951248,−0.01470334,−0.03336572,0.01336034,−0.00045607,0.01492316,0.02321628,−0.02551141]。在向量空间中嵌入通常同时出现的项目组。必须在嵌入层中指定两个关键参数。 These are:
output_dim:向量空间的大小。
input_length:输入的大小,即窗口大小。
训练和测试数据集
训练数据集进一步细分为80%的训练集和20%的验证集。该模型在80%的训练数据上进行训练,并在每个时代使用剩余的20%的训练数据集进行验证。执行该验证方法是为了跟踪训练进度并识别过拟合。当验证精度开始下降而训练精度继续增加时,就会识别出过拟合。利用校验指针根据验证精度从epoch中选择最优模型;这种方法还有助于最小化任何潜在的过拟合。然后将生成的模型用于独立测试数据集的独立测试。
输入
与传统的机器学习方法相比,使用深度学习的主要优点是排除了人工特征提取。DL方法的输入是FASTA格式的序列窗口。例如,窗口大小为33,对于单热编码,输入维度为33 × 21。对于相同窗口大小的嵌入,当嵌入的输出维数为21时,输入维数为33 × 21。
DeepSuccinylSite架构
DeepSuccinylSite的整体结构如图所示。1.
对输入数据进行编码后,将编码后的数据送入网络。这两种编码方法都使用了相同的体系结构,只是在嵌入编码的情况下包含了嵌入层和lambda层。
下一层是卷积层。先前基于dll的磷酸化位点模型(DeepPhos, MusiteDeep)[19,20]使用了1-D(维)卷积层,而我们使用了2-D(维)卷积层,从而增加了我们选择2-D大小的灵活性。如果我们使用一维卷积层并做同样的事情,那么我们将无法推断出许多特征信息,因为x轴是固定的(它将保持在21),并且只会垂直步幅。此后,其他图层也选择了2D。我们使用二维卷积层来优先包含滤波器大小为17 × 3(对于窗口大小为33的窗口,PTM站点位于第17位),这将包括每个步幅中的PTM站点。使用这个过滤器大小,以及禁用填充,允许模型在不影响性能的情况下对训练时间进行优化。为了避免过拟合,使用了0.6的较高落差。此外,采用整流线性单元(ReLU)作为各层的激活函数。ReLU被认为是最优的激活函数,因为它的激活稀疏,最小化了过拟合的可能性,最大化了模型的预测能力。我们使用了两个卷积层,一个maxpooling层,一个具有两个密集层的全连接层,以及一个输出层。 The parameters used in the model are given in Table2.
Adam优化被用作我们架构的优化器,正如Kingma等人之前所描述的那样。[23]。Adam使用自适应学习率方法来计算每个参数的个人学习率。Adam与经典的随机梯度下降法不同之处在于,随机梯度下降法在训练过程中对所有权重更新都保持单一、恒定的学习率[24]。具体来说,Adam结合了自适应梯度算法和均方根传播的优点,允许对模型进行有效的训练。由于本研究是一个二元分类问题,因此使用二元交叉熵(与给定分布相关的不确定性度量)或对数损失作为损失函数。二元交叉熵为:
标签在哪里(1表示正,0表示负)和\({\hat{y}}_i \)为所有N点均为正的预测概率。对于每个阳性站点(y = 1),并补充说\(\log \left({\hat{y}}_i\right) \)损失的对数次方,也就是它为正的对数概率.相反,对于每个负面站点(y = 0),并补充说\(\log \left(1-{\hat{y}}_i\right) \)也就是它为负的对数概率。
全连接层包含两个密集层,分别为768和256个节点,最终输出层包含2个节点。
模型评估和性能度量
在本研究中,采用10倍交叉验证来评价模型的性能。在10倍交叉验证中,数据被划分为10个相等的部分。然后,省略一个零件进行验证,并对其余9个零件进行训练。重复此过程,直到所有部件都用于验证。
采用混淆矩阵(CM)、马修相关系数(MCC)和受试者工作特征(ROC)曲线作为绩效指标。ROC曲线是一个图形图,说明了二元分类器的诊断能力,而曲线下面积(AUC)表示可分离性的程度或度量。由于琥珀酰化位点的识别是一个二元分类问题,因此混淆矩阵大小为2 × 2,由真阳性(TP)、真阴性(TN)、假阳性(FP)和假阴性(FN)组成。使用这些变量计算的其他指标包括准确性、敏感性(即真阳性率)和特异性(即真阴性率)。
结果
最佳窗口大小和编码
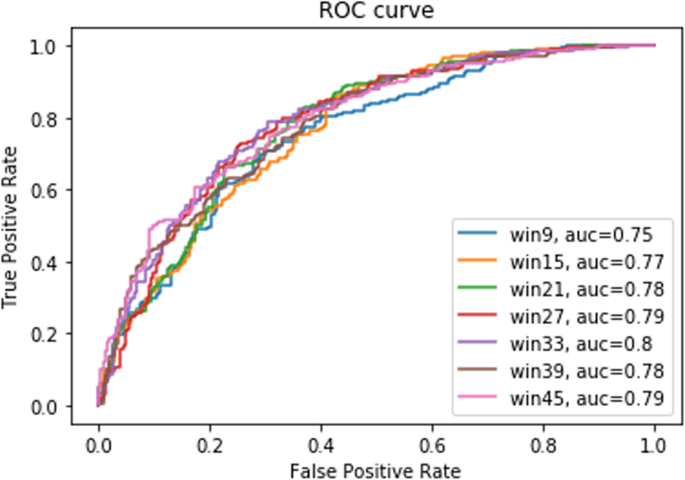
最初,窗口大小从9到45进行了一次性编码和嵌入测试。例如,当窗口大小为9时,赖氨酸(K)残基设置在窗口中间,上游有4个氨基酸残基,下游有4个氨基酸残基。当窗口大小为33时,单热编码和嵌入的MCC都最高,窗口大小的进一步增加导致MCC的降低(表2)3.).Likewise, the highest specificity and AUC were achieved using a window size of 33, with only a marginal reduction in sensitivity when using embedding (Table3.和无花果。2).因此,窗口大小为33被认为是本研究的最佳窗口大小。有趣的是,Wang等人也利用33的窗口大小,使用one-hot编码进行磷酸化位点预测[15]。值得注意的是,本研究与之前Wang等人的研究之间窗口大小的一致性与许多蛋白质间氨基酸相互作用的已知范围相关。重要的是,对于测试的每个窗口大小,嵌入都比单一热编码表现得更好,只有少数例外。因此,本研究选择嵌入编码。
最佳嵌入尺寸的识别
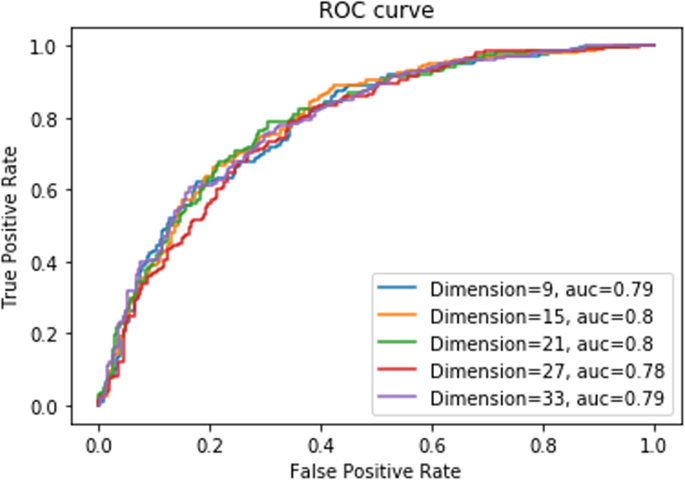
接下来,我们试图确定最佳的嵌入尺寸。为此,对9到33的尺寸进行了嵌入测试。需要注意的是,增加嵌入的维数将导致更高的计算成本。因此,我们的目标是确定在所有指标之间取得平衡的最小维度。因为MCC经常被用作整体模型性能的替代品,所以它的优先级略高于其他参数。尽管15和21维度大小都达到了这样的平衡,但使用21维度大小的性能指标通常更好。事实上,21的维度尺寸达到了最高的MCC,其敏感性和特异性得分在这些领域的最高得分的7%以内(表2)4).15和21维度尺寸的AUC得分最高(图2)。3.).综上所述,这些数据表明,使用我们的体系结构,21的维度大小是最优的。因此,选择21的尺寸尺寸进行模型开发。尺寸大小与每个载体中存在20个氨基酸残基和1个伪残基相一致。
交叉验证和可选分类器
我们的最终模型,我们称之为DeepSuccinylSite,使用窗口和尺寸大小分别为33和21的嵌入。基于5轮10倍交叉验证,DeepSuccinylSite表现出鲁棒性,具有一致的性能指标,平均MCC为0.519 +/−0.023,AUC为0.823(补充文件)1:表S3)。我们还实现了额外的深度学习架构和不同的机器学习模型,其中输入是手工制作的“物理化学”特征,而不仅仅是蛋白质序列。从本质上讲,该实现将各种物理化学特征与XGBoost结合起来,以提取突出的特征。在计算特征时,我们排除了任何带有' - '的序列。然后,我们使用XGBoost提取突出特征,该方法具有更好的精度,在阈值为0.00145时共获得160个特征。有趣的是,使用这些方法的方法的性能不如DeepSuccinylSite,其输入仅是蛋白质序列(附加文件)1: Table S2).关于我们模型性能的进一步信息包含在附加文件中1.此外,基于特征的深度学习架构的结果显示在附加文件中1:图S1。
与其他深度学习架构的比较
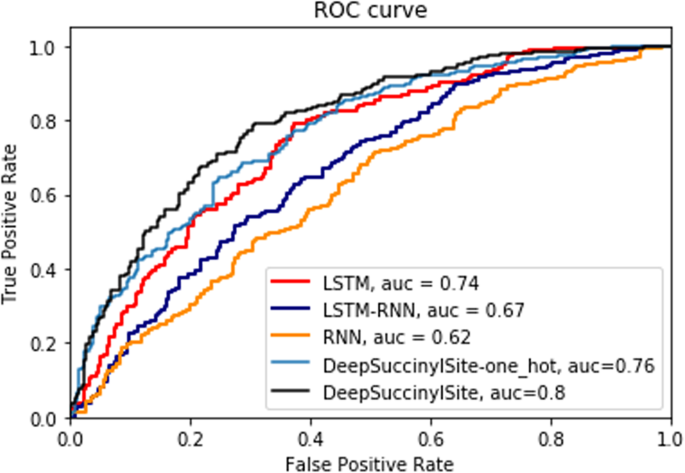
其他深度学习架构,例如循环神经网络[25]及长短期记忆[26],以及组合模型LSTM-RNN对单热编码(DeepSuccinylSite-one_hot)进行了编码,并与DeepSuccinylSite的独立测试结果进行了比较(表5).此外,我们实现了一个额外的DL架构,其中输入包括除了主氨基酸序列之外的其他特征。具体来说,该实现利用了1)物理化学特征的组合,如伪氨基酸组成(PAAC),“k-间隔氨基酸对”(AAP);2)自相关特征,如Moreau-Broto自相关和成分、过渡和分布(CTD)特征;3)熵特征,如Shannon熵、相对熵和信息增益。在计算特征时,我们排除了任何带有' - '的序列。然后,我们使用XGBoost提取具有更好精度的突出特征,在阈值0.00145处共获得160个特征。使用特征的算法版本被称为基于deepsuccinylsite特征的算法。
为了进行公平的比较,我们使用相同的平衡训练和测试数据集,对这三种DL架构使用33个窗口大小和一个热编码。结果如表所示5ROC曲线如图所示。4.The results for our DL model with embedding (DeepSuccinylSite) is also shown.The detailed architecture of these models, including results for other window sizes are discussed in Additional file1并在附加文件中介绍了这些方法的性能1:表S1。对于单热编码,DeepSuccinylSite的MCC和AUC得分均优于其他DL架构。同样,我们使用嵌入的最终模型在所有模型中获得了最高的MCC和AUC分数(表5).
与现有模型的独立测试比较
Next, the performance of DeepSuccinylSite was compared with other succinylation site predictors using an independent test set as mentioned in the benchmark dataset earlier.在这些分析过程中,一些最广泛使用的琥珀酰化位点预测工具,如iSuc-PseAAC [8], iSuc-PseOpt [9], psu - lys [10],琥珀酸[11], SuccineSite2.0 [12], GPSuc [13]及psuccess [14], were considered.所有这些方法都使用与表中相同的训练和独立测试数据集6.这些先前发表的方法的性能指标取自各自的手稿,主要基于PSuccE中所做的比较[14]。
DeepSuccinylSite的敏感性评分比第二高的模型高58.3%(表2)6).相比之下,我们的模型在所有测试模型中表现出最低的特异性得分。而DeepSuccinylSite的特异性评分仅比排名靠前的方法低22.2%。因此,DeepSuccinylSite在MCC测试中获得了显著更高的性能。事实上,与第二高的方法GPSuc相比,DeepSuccinylSite显示出约62%的MCC增加。总之,我们所描述的新架构,称为DeepSuccinylSite,在精确和准确预测琥珀酰化位点方面显示出显著提高的性能。
讨论
琥珀酰化是相对新近发现的PTM,由于引入一个大的(100 Da)化学部分改变修饰残渣的电荷的生物学意义而引起了人们的兴趣。琥珀酰化的实验检测是劳动密集型和昂贵的。由于有一个相对较大的数据集,其中包含4750个用于训练的积极站点,因此我们可以实现不同的DL架构。与之前在文献中描述的模型相比,本文中描述的模型优化过程导致了在精确预测琥珀酰化位点方面的显著改进。本研究考虑了两种类型的编码,单热编码和嵌入。我们的研究结果表明,嵌入是一种最佳方法,因为它允许模型学习与氨基酸特征相似的表示,从而进一步提高识别假定修饰位点的能力。
此外,DeepSuccinylSite证实了文献中先前的指示,这些指示表明33的窗口大小最佳地反映了蛋白质中的局部化学相互作用,由于其在MCC等指标中的表现,可以预测PTM的位置。其中一个重要参数是嵌入尺寸。DeepSuccinylSite使用从9到33的不同维度进行训练。随着维度的增加,训练时间也随之增加。虽然尺寸15和21之间没有显著性差异,但考虑到氨基酸残基的数量和稍好的结果,本研究选择21作为包埋尺寸。最后,在嵌入维数为21、窗口大小为33的情况下,DeepSuccinylSite在敏感性、特异性和MCC方面的效率得分分别为0.79、0.69和0.48。
为了进一步改进,我们可以代替目前基于蛋白质序列的窗口序列,以感兴趣的位点为中心提取基于结构的窗口序列,并将该窗口作为输入。当蛋白质的结构不可用时,蛋白质结构预测管道如I-TASSER [27]或ROSETTA [28],可以首先用于预测结构。由于蛋白质的结构比序列更保守,我们希望更好地捕捉进化信息,从而获得更好的预测精度。此外,我们还可以通过使用基于序列的窗口、基于结构的窗口、物理化学性质创建多个模型,然后利用投票方法来提高该方法的性能。最后,多窗口输入,如在DeepPhos [16],使用我们的编码技术可以提高性能。然而,这些方案需要更多的数据集,一旦有更多的实验数据,我们可以更详细地探讨这一点。我们还探讨了数据大小对预测性能的影响(附加文件1:表S4和附加文件1:图S2)。这些研究表明,最初,我们的模型的性能随着数据量的增加而增加,然后达到一个平台。这在某种程度上与深度学习的普遍共识相反,深度学习的性能会根据幂律随数据量的增加而增加。然而,随着未来可能会有更多的实验数据,我们可以对性能如何随着数据量的增加而变化进行更全面的研究。也许,这也表明,随着数据的增加,我们可能不得不开发更复杂的深度学习模型。
利用本文中描述的独特架构,DeepSuccinylSite模型在预测质量方面比现有模型有了实质性的改进。该模型的实用之处在于它能够预测可能被琥珀化的赖氨酸残基。因此,该模型可用于优化琥珀酰化位点实验验证的工作流程。具体而言,使用该模型可以显著减少识别这些站点的时间和成本。当PTM本身可能解释所观察到的生物现象时,该模型在假设生成方面也可能具有一定的效用。
结论
在这项研究中,我们描述了DeepSuccinylSite的发展,这是一种新颖而有效的深度学习架构,用于预测琥珀酰化位点。与其他机器学习架构相比,使用该模型的主要优势是消除了特征提取。因此,其他PTM站点可以很容易地应用到该模型中。由于该模型仅利用了两个卷积层和一个max-pooling层来避免对当前数据的过拟合,因此提供新的数据源可能会允许在未来进一步修改该模型。总之,DeepSuccinylSite是一种有效的深度学习架构,在预测琥珀酰化位点方面具有一流的结果,并有可能用于一般的PTM预测问题。
数据和材料的可用性
在本研究中分析的数据集和模型以及补充资料可在https://github.com/dukkakc/DeepSuccinylSite.
改变历史
2022年8月22日
缩写
- AUC:
-
ROC曲线下面积
- 有线电视新闻网:
-
卷积神经网络
- DL:
-
深度学习
- LSTM:
-
长短期记忆
- 世纪挑战集团:
-
Mathew correlation coefficient
- 铝:
-
翻译后修饰
- ReLU:
-
线性整流函数
- RNN:
-
循环神经网络
- 中华民国:
-
Receiver operator characteristics
参考文献
Hasan MM, Khatun MS. Prediction of protein Post-Translational Modification sites: An overview.Ann Proteom Bioinform. 2018;2:049-57.https://doi.org/10.29328/journal.apb.1001005.
Medzihradszky KF。肽序列分析。方法酶学杂志2005;402:209-44。
Agarwal KL, Kenner GW, Sheppard RC。猫胃泌激素。质谱分析肽序列的一个例子。化学学报,2009;29(6):516 - 516。
Welsch DJ, nelstuen GL.氨基端丙氨酸在凝血酶原片段1膜结合的钙特异性过程中的作用。生物化学。1988;27(13):4939 - 45。
李文杰,李文杰,李文杰,等。精氨酸翻译后修饰的生物化学检测方法。生物聚合物。2014;101(2):133 - 43。
Umlauf D, Goto Y, Feil R. Site-specific analysis of histone methylation and acetylation.方法Mol生物学,2004;287:99-120。
Jaffrey SR, Erdjument-Bromage H, Ferris CD, Tempst P, Snyder SH. Protein S-nitrosylation: a physiological signal for neuronal nitric oxide.Nat Cell Biol. 2001;3(2):193–7.
徐勇,丁彦烨,丁俊,雷玉华,吴丽丽,邓妮。iSuc-PseAAC:结合肽位置特异性倾向预测蛋白质中赖氨酸琥珀酰化。科学通报2015;5:10184。
贾娟,刘忠,肖晓霞,刘波,周kc。iSuc-PseOpt:利用序列耦合效应识别蛋白质中赖氨酸琥珀酰化位点和优化不平衡训练数据集。《肛肠生物化学》2016;47:48 - 56。
贾娟,刘忠,肖晓霞,刘波,周建昌。pSuc-Lys:基于PseAAC和集成随机森林方法预测蛋白质中赖氨酸琥珀酰化位点。中国生物医学工程学报。2016;394:223-30。
Hasan MM, Yang S, Zhou Y, Mollah MNH.SuccinSite: a computational tool for the prediction of protein succinylation sites by exploiting the amino acid patterns and properties.摩尔BioSyst。2016; 12(3): 786 - 95。
王晓明,王晓明,王晓明,王晓明。植物蛋白琥珀酰化位点的研究进展。纳米医学杂志,2017;12:6303-15。
Hasan MM, Kurata H. GPSuc:通过聚合多个序列特征预测通用和种特异性琥珀酰化位点。PLoS One. 2018;13(10):e0200283。
宁强,赵霞,鲍林,马志,赵霞。基于集成支持向量机的蛋白质序列琥珀酰化位点检测。生物信息学杂志,2018;19(1):237。
王东,曾松,徐超,邱伟,梁勇,乔士涛,等。MusiteDeep:用于一般和激酶特异性磷酸化位点预测的深度学习框架。生物信息学。2017;33(24):3909 - 16。
罗凤林,王明辉,刘宇,赵兴明,李奥。基于深度学习的蛋白质磷酸化位点预测,生物信息学,2019;35(16):2766-73。
付辉,杨颖,王旭,王宏,徐勇。DeepUbi:一种基于深度学习的蛋白质泛素化位点预测框架。BMC生物信息学。2019;20(1):86。
吴敏,杨颖,王宏,徐勇。一种更准确地回忆已知赖氨酸乙酰化位点的深度学习方法。BMC生物信息学。2019;20(1):49。
李文杰,李文杰,李文杰。深度学习。大自然。2015;521:436。
Chollet F,等。Keras;2015.https://keras.io.
在:本吉奥Y,埃让杜沙尔梅R,文森特P,数学研究C,编辑。一种神经概率语言模型2001.
Kulmanov M, Khan MA, Hoehndorf R. DeepGO: predicting protein functions from sequence and interactions using a deep ontology-aware classifier.生物信息学。2017;34(4):660 - 8。
邓金玛,张立军。一种随机优化方法。arXiv电子印刷[互联网]。2014;01:2014https://ui.adsabs.harvard.edu/abs/2014arXiv1412.6980K.
刘国强,李志强。回归函数最大值的随机估计。数学学报,2002;23(3):462-6。
Jain LC, Medsker LR。循环神经网络:设计和应用:CRC press, Inc.;1999.416便士。
霍克瑞特,#252,施米德胡伯,长短期记忆。神经计算。1997;9(8):1735-80。
张艳,张志刚,张志刚,张志刚。一种基于tasser的蛋白质结构和功能预测方法。应用物理学报,2010;5(4):725-38。
李建平,李志强,李志强,李志强。基于rosett3的对称大分子结构建模。公共科学学报,2011;6(6):e20450。
确认
不适用。
关于这个补充剂
本文已作为BMC生物信息学第21卷补充3,2020:联合国际GIW和ABACBS-2019会议论文集:生物信息学(第2部分)。该补充的全部内容可在网上查阅https://bmcbioinformatics.biomedcentral.com/articles/supplements/volume-21-supplement-3.
资金
这项工作得到了美国国家科学基金会(NSF)资助号1901793,1564606和1901086 (DK)的支持。RHN获得了美国国家科学基金会(NSF)颁发的HBCU-UP卓越研究奖(1901793)和美国国立卫生研究院国家普通医学科学研究所颁发的SC1奖(5SC1GM130545)。HS得到了JSPS KAKENHI资助号JP18H01762和JP19H04176的支持。
作者信息
作者及隶属关系
贡献
DK, SH, RN, KR构想并设计了实验。NT和MC进行了实验和数据分析。NT, DK, SMM和MC撰写了论文。RN、SH、DK、KR、SMM对稿件进行了修改。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:
包含补充表和图中提到的文本。我们描述了各种其他深度学习架构、其他机器学习架构、交叉验证结果和不同样本量的独立测试结果。表S1。独立测试结果。表S2。不同机器学习架构的独立测试结果。图S1。基于特征的dl模型的ROC曲线。表S3。不同运行的交叉验证(CV)结果。表S4。不同样本量的独立检验结果。图S2。MCC和AUC用于不同样本量的独立测试。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
Thapa, N., Chaudhari, M., McManus, S.。et al。DeepSuccinylSite:一种基于深度学习的蛋白质琥珀酰化位点预测方法。BMC生物信息学21(增刊3)63(2020)。https://doi.org/10.1186/s12859-020-3342-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-020-3342-z
关键字
- Succinylation
- 深度学习
- 卷积神经网络
- 循环神经网络
- 长短期记忆
- 嵌入